
Сome to the bioinformatics side: Институт биоинформатики в Санкт-Петербурге
17 апреля 2016
Сome to the bioinformatics side: Институт биоинформатики в Санкт-Петербурге
- 4412
- 0
- 4
Если бы Дарт Вейдер жил в Санкт-Петербурге, он бы тоже поступил в Институт биоинформатики и учился собирать геномы и писать классы в Python
иллюстрация Елены Беловой
-
Автор
-
Редакторы
Институт биоинформатики существует в Петербурге с 2013 года. За несколько лет малоизвестные курсы по биоинформатике, открытые в 2010 на базе Академического университета РАН, превратились в годовую программу, на которой студентов (биологов и информатиков) учат видеть и решать серьезные биоинформатические проблемы. Миссия института — популяризовать биоинформатику и создать сообщество ученых, способных развивать это направление в России. Здесь мы расскажем, как устроен Институт биоинформатики, как в нём «делают» биоинформатиков, какие проблемы в целом стоят перед этой наукой и почему ее важно (и круто) изучать.
Институт биоинформатики (далее — ИБ) находится в бизнес-центре «Таймс» и делит пространство с офисом компании JetBrains. Много места нам не нужно: в составе ИБ примерно 15 сотрудников, включая преподавателей, и 30-40 студентов, а ноутбук — это единственное, что нужно для работы или учебы. А зачем вообще идти учиться в ИБ? Если задать этот вопрос руководителю, он может ответить, что это хороший способ научиться взаимодействовать со своими данными, произведенными в экспериментах, обрабатывать и визуализировать их. И будет, конечно, прав. Но помимо этого у биоинформатики низкий порог вхождения: всё, что вам нужно — это компьютер и мозги, не надо искать дорогостоящие реагенты, три месяца ждать, пока их доставят, следить и ухаживать за подопытным животным. Это очевидные вещи, и мы надеемся, что если вы решили прочитать этот текст, то зачатки мотивации у вас уже есть.
Если вы всё еще думаете, что математика — не ваш конек, программирование — это что-то для гиков, а графики можно смастерить и в Excel, то самое время познакомиться с автором этой статьи. Я биолог. Что еще хуже — биолог, свернувший в биологию из журналистики. Большее расстояние между мной и математикой сложно представить. Ровно год назад начала учиться программировать, впервые в жизни. Одним словом, даже если вы подходите под это описание — не всё потеряно, просто в жизни вам еще не попадались люди, способные заинтересовать своим предметом (будь то математика, статистика или финская ходьба с палками).
Биоинформатика чаще всего ассоциируется у малознакомых с ней людей со словами «генόм» и «секвенирование», однако ими всё далеко не ограничивается. На сегодняшний день биоинформатика подгребла под себя все исследования, где так или иначе для анализа данных используется компьютер в чуть более продвинутом режиме, чем простенькая статистическая обработка и визуализация. Часто это действительно работа с последовательностями нуклеотидов или аминокислот, но уже тут можно перейти на следующий уровень — анализ и моделирование трехмерных структур белков, а также РНК и ДНК, чем занимается структурная биология [1], [2]. Идем дальше — зная трехмерные структуры молекул, можно предсказывать их взаимодействия (как для поиска новых лекарств, так и для анализа того, что происходит в организме с уже существующими), а имея на руках много данных возможно реконструировать целые метаболические сети [3], [4].
Честно говоря, с тем, как кого называть, есть некоторая путаница. Лично мне импонирует версия из онлайн-курса «Основы вычислительной и системной биологии», читаемого в МТИ, где за биоинформатику строго принимается вид деятельности по созданию специализированных программ и алгоритмов (например, blast — кажется, что он существовал всегда, но кто-то всё-таки придумал алгоритм локального выравнивания и реализовал его, и именно эти люди заслуживают именоваться биоинформатиками). Всё остальное с некоторыми оговорками отправляется в раздел вычислительной биологии (computational biology), то есть если вы собираете геном, скажем, верблюда, аннотируете его и сравниваете с геномом альпаки, не создавая для этого нового софта, — то, увы, вы не биоинформатик. Однако здесь мы не собираемся жестко придерживаться этой классификации. Наша цель — рассказать про ИБ, а в нём, так или иначе, есть и те, и другие персонажи.
Как всё начиналось
Ты помнишь, как всё начиналось?
Всё было впервые и вновь.
Как строили лодки, и лодки звались
Вера, Надежда, Любовь.
За тех, кто в море («Машина Времени»)
«А помнишь, как всё начиналось»
Институт вырос из курсов по биоинформатике при Санкт-Петербургском академическом университете РАН (рис. 1). Курсы запустил в 2010 году Николай Вяххи — в то время аспирант матмеха СПбГУ, занимавшийся научной работой в области биоинформатики.
Рисунок 1. Первое объявление на habrahabr.ru о первых курсах по биоинформатике в Петербурге
Биоинформатика к тому времени уже активно развивалась в мире, но в России её становление запаздывало. А мест, где можно учиться биоинформатике, по всей стране были единицы. В Москве существовала активная школа биоинформатики, организованная Михаилом Сергеевичем Гельфандом, но в Петербурге всё было тише воды, ниже травы.
Сначала это были курсы по биологии и биоинформатике для всех желающих, потом они обрели более строгий формат — появился конкурсный отбор, больше предметов, учет успеваемости и зачеты. Программа была рассчитана на «технарей»: чтобы попасть на обучение, требовалось уже уметь программировать и знать базовые алгоритмы.
Постепенно, с ростом числа биоинформатиков в Петербурге и России в целом, курсы усложнялись и дополнялись (подробную историю создания биоинформатического сообщества в Петербурге можно прочитать, например, здесь). Коля организовывал курсы в одиночку, потом, в 2012, к нему присоединилась Катя Чайкина, до этого занимавшаяся образовательной программой GameChangers в индустрии ИТ и исследованиями в области образования.
Трансформация курсов в институт произошла в марте 2013 года, а уже летом ИБ провел первое глобальное мероприятие, организованное с нуля — летнюю школу по биоинформатике (рис. 2), на которую собрались студенты и аспиранты со всей России и СНГ. Также расширилась и команда: к нам присоединилась куратор Анна Черныш, волонтеры Кирилл Григорьев и Ярослав Баранов — в то время студенты СПХФА и участники вышеупомянутой программы GameChangers. Впоследствии они не только помогали организовывать школы, но и приняли активное участие в запуске программы для биологов.

Рисунок 2. Команда ИБ на первой летней школе по биоинформатике в Подмосковье
Почему же всё-таки «институт», и зачем понадобились такие перемены?
В какой-то момент мы поняли, что c таким количеством идей и планов мы вырастаем из формата курсов. В первую очередь нам хотелось расширять образовательные программы и запустить обучение для биологов. От них мы получали много сообщений о том, что в Петербурге негде изучать жизненно важную биоинформатику. Еще мы давно задумывали и уже готовились к проведению Всероссийской летней школы по биоинформатике. Немаловажным фактором стало и желание дать проекту больше независимости.
Пару месяцев мы готовились, и в марте 2013 года объявили в паблике ВКонтакте о создании Института биоинформатики (рис. 3). И закрутилось — в июле 2013-го провели первую летнюю школу, в августе — набор на программу для биологов.
Почему именно „институт“, а не что-то другое? Мы долго думали над названием. В Москве уже была Школа биоинформатики, поэтому обсуждались самые разные варианты от „академии“ до „хаба“. Остановились на „институте“. Хоть это и казалось сначала слишком глобальным, но удачно объединяло в себе и академичность, и долгосрочные образовательные программы, и все другие планы — школы, мероприятия, сообщество, исследования.

Рисунок 3. Мы открылись!
Про финансирование
С самого начала институт поддерживался ИТ-компанией JetBrains и лабораторией алгоритмической биологии Павла Певзнера (ныне — лаборатория алгоритмической биотехнологии в СПбГУ) — профессора отделения компьютерных наук и инженерии Университета Калифорнии (Сан-Диего), победителя первого конкурса мегагрантов. Лаборатория была создана немного позже курсов по биоинформатике, и сейчас ее сотрудники принимают активное участие в преподавании и руководстве студенческими научными проектами.
Однако на все мероприятия, дополнительные курсы и активности ИБ привлекает внешнее финансирование от фондов (РФФИ, РВК, до недавнего времени — «Династия»), компаний и различных грантовых программ. Сейчас это становится всё сложнее. Наши мероприятия поддерживал фонд «Династия» вплоть до его закрытия, и без этой поддержки очень грустно, особенно при подготовке новой летней школы. Институту помогает ряд компаний (за что мы им очень благодарны!), но всё это не постоянное финансирование — для каждого мероприятия мы проходим через фандрайзинговые кампании снова и снова. Коммерческие компании пока не готовы поддерживать образование по биоинформатике на постоянной основе, даже если заинтересованы в «выходящих» из него специалистах.
Все наши проекты некоммерческие — даже на платных мероприятиях мы не зарабатываем: все вырученные средства идут на организацию, гранты талантливым и иногородним участникам, оплату труда преподавателей.
Кто все эти люди
В основной команде института сейчас три человека: Коля Вяххи, Катя Вяххи и Наташа Машьянова.
Можно сказать, что мы работаем в режиме стартапа — хоть у каждого и есть своя зона ответственности, все занимаются всем. Каждый наш проект, мероприятие или программа — это полностью общий результат и вклад всей команды. Чаще всего работа кипит и по выходным, а размах идей всё равно превышает количество рук и человеко-часов.
ИБ располагается в любезно предоставленном компанией JetBrains офисе (рис. 4). В нём же проходят занятия студентов — в двух аудиториях, для «биологов» и «информатиков» (рис. 5).

Рисунок 4. Офис внутри и снаружи. Биоинформатика здесь! Нажмите на рисунок, чтобы посмотреть его в полном размере.

Рисунок 5. Студенты в биоинформатических футболках, которые ИБ дарит каждый семестр всем, успешно закрывшим все предметы
Кроме команды, есть замечательные люди, которые нам помогают: Алла Львовна Лапидус (научный советник ИБ, заместитель директора лаборатории алгоритмической биотехнологии СПбГУ), Вадим Назаров (биоинформатик-исследователь из ИБХ РАН и НИУ ВШЭ), Андрей Пржибельский (биоинформатик из лаборатории алгоритмической биотехнологии СПбГУ), наши преподаватели и выпускники. Без выпускников мы просто не справились бы с потоком собеседований абитуриентов!
В организации мероприятий Института биоинформатики для меня воплотились целых три моих интереса — биоинформатика (для понимания специфики области), психология (для понимания людей) и системное мышление (для организации процессов в масштабных проектах). Образовательная деятельность, наверное, самый медленный, но самый эффективный способ дать людям возможность найти свои интересы, и я очень рад работать вместе с ИБ над этим. И хотя я базируюсь в Москве, это только подталкивает к общению и рабочей деятельности с замечательными людьми из Северной столицы.
В августе 2015 года в ИБ открылось исследовательское подразделение (рис. 6), которое возглавил Александр Предеус, об этом мы расскажем в деталях в следующей части.

Рисунок 6. Трудовой полдень исследовательской группы ИБ в субботу
Что происходит сейчас
Помимо основных образовательных программ и ряда регулярных мероприятий (рис. 7), сейчас ИБ занимается несколькими направлениями. Во-первых, это онлайн-образование: уже вышло несколько открытых бесплатных онлайн-курсов, и активно готовятся новые. Скоро ИБ объявит о новом долгожданном онлайн-проекте — тут пока не будем раскрывать детали (следите за новостями!), но мы давно над ним работаем и надеемся, что он увидит свет в ближайшее время.
Второе направление — это сообщество биоинформатиков. 1 апреля вместо шуток запустили чат по биоинформатике в реальном времени в Slack. Осенью 2015 года совместно с компанией Parseq Lab — платформу вопросов и ответов по биоинформатике Bioinformatics — help для поддержки русскоязычного сообщества биоинформатиков и биологов, публикуем вакансии по биоинформатике. Но обо всём по порядку.

Рисунок 7. А еще мы отмечаем веселыми тортиками дни рождения Института биоинформатики со студентами и преподавателями
Как всё устроено: поступление и обучение
Этот раздел про то, как в нашем институте проходит обучение, а именно: про лекции, задания, темы и преподавателей, — короче, про субботы, которые мы все добровольно подписались проводить в ИБ целый год. Итак, на входе в программу (рис. 8) имеется две выборки — биологи и информатики, каждая примерно по 20 человек. На выходе ожидаем получить отсортированную группу биоинформатиков, способную совместно читать и писать код, обсуждать проблемы сборки очередного референсного генома человека и вообще общаться на одном языке. Стратегическая цель — научить биологов статистике и программированию, а информатикам объяснить, чем отличаются SNP от CNV, археи от бактерий и как применять навыки программирования в биологии.
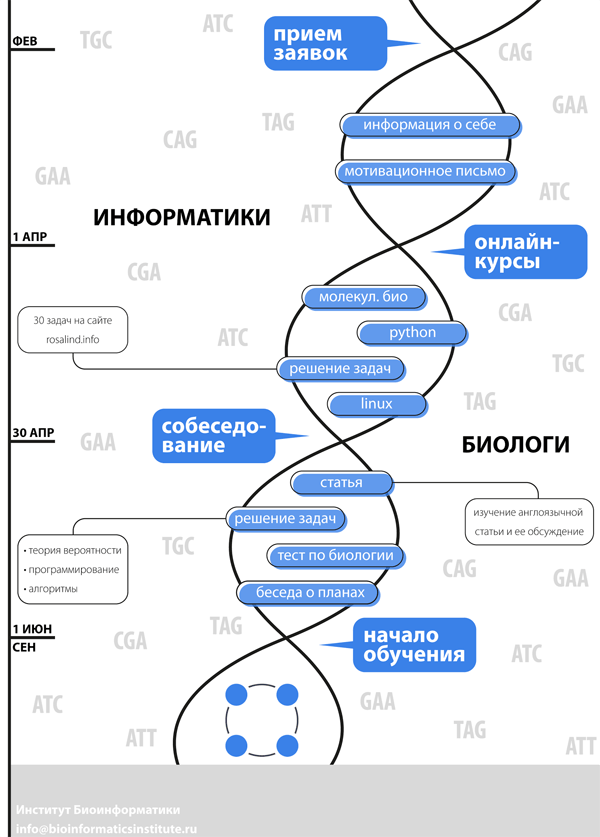
Рисунок 8. Информацию о поступлении можно найти на сайте — с подробной инструкцией для биологов и информатиков и даже примерами заданий и статей, необходимых для успешного прохождения собеседования.
Bitten Pixel Studio

Рисунок 9. Шутки шутками, а вот в BioMed Central есть целая статья о том, как обработка данных с микрочипа в Excel может обернуться непреднамеренными ошибками в именах генов [5]
Чтобы научиться программировать, нужно программировать. Чтобы окончательно не впасть в отчаянье, биологов учат двум языкам: Python и R. Первый в некоторой степени универсален в биоинформатике и относительно лёгок в освоении, второй — незаменим при статистической обработке данных (рис. 9). После появления нашего вводного онлайн-курса по Python (его необходимо пройти для поступления) «вливаться» в язык стало гораздо проще: во время обучения на базовые вещи (что такое типы данных: строки, списки, функции) тратится гораздо меньше времени, а значит, можно успеть изучить больше. Занятия по статистике и программированию на R сильно перекликаются и охватывают всё от базовых статистических тестов (хи-квадрат, тест Фишера, непараметрический критерий Шапиро-Уилка) до более сложных методов, таких как кластеризация, метод главных компонент и элементы нейронных сетей.
Эти курсы есть и на Stepic.org, и меня часто спрашивают — а почему бы просто не учиться биоинформатике по онлайн-курсам? Это можно, но есть три принципиальных момента: гораздо больше практики, обратная связь с преподавателем, возможность применять знания «не отходя от кассы» — в собственных проектах (о чём речь пойдет чуть позже). Помимо этих предметов, которые длятся весь год, в осеннем семестре у биологов есть дискретная математика — курс, напоминающий элементы комбинаторики и теории вероятностей, а также дающий базовое представление об алгоритмах в биоинформатике (да, да, у биоинформатиков бывает так много данных, что приходится придумывать множество математических трюков, чтобы как-то с ними обойтись). Курс по введению в биоинформатику знакомит с программами, работающими с геномными данными: это сборка генома de novo, оценка качества этой сборки, выравнивание фрагментов на референсный геном и так далее (подробнее можно прочитать в статье о некоторых этапах биоинформатического анализа [6]). В весеннем семестре добавляется молекулярная филогения — курс о методах и подходах к построению филогенетических деревьев на основе анализа ДНК и белковых последовательностей.
Информатики для поступления тоже проходят онлайн-курс, но по основам молекулярной биологии. Смысл тот же: меньше времени тратить на совсем элементарные вещи и побольше дискутировать о пользе клонирования генов, применения CRISPR/Cas9 для лечения наследственных заболеваний и даже для улучшения физического состояния и интеллекта. Дискуссии часто уезжают на обеденные перерывы, а на занятиях информатики успевают повторить такие вещи, как строение про- и эукариотических клеток, структура генома, эволюционные модели и элементы биохимии и филогенетики.
«Биомолекула» писала, как в целом работает система CRISPR/Cas9, как ее можно усовершенствовать и как с помощью этой технологии уже пытаются лечить миодистрофию Дюшена и один из видов рака — острый лимфобластный лейкоз [7–10].
С Павлом Добрыниным, который ведет молекулярную биологию, можно обсудить много чего, в том числе его недавно вышедшую статью про секвенирование и анализ генома гепарда (рис. 10), из которой вы почерпнете много драматических деталей об истории и непростой судьбе этого вида [11]. Например, давно было известно, что если поймать гепарда в Африке и пересадить кусок его кожи другому гепарду — он приживется без всяких иммуносупрессоров, а всё потому, что уровень гомозиготности в популяции гепардов достигает 95% (да они почти что клоны!), и гены, отвечающие за гистосовместимость, у них практически идентичные. Как гепарды докатились до такого и какие еще у них есть проблемы — читайте в статье. На самом деле, на этом чистая биология заканчивается.
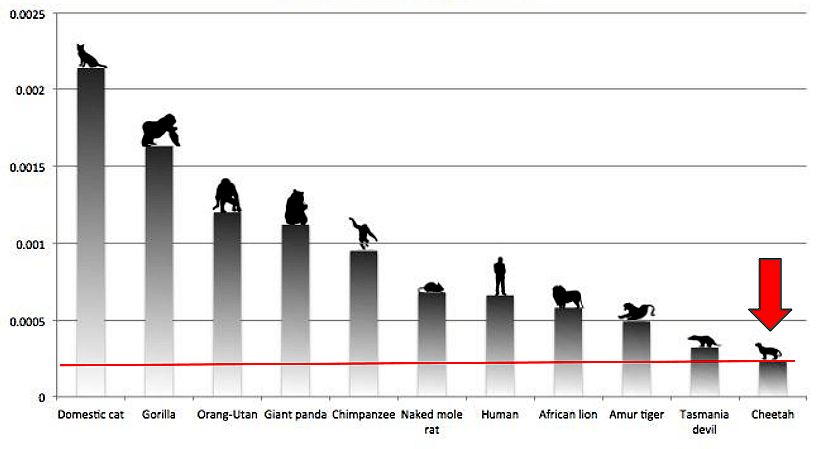
Рисунок 10. Уровень вариаций числа копий (CNV), то есть делеций или дупликаций, в геномах разных видов. Среди представленных организмов максимальное количество вариаций встречается в геноме домашней кошки, а минимальное — в геноме гепарда. Человек, кстати, по этому критерию ближе к гепарду, чем к кошке.
Остальные предметы — это либо алгоритмы в биоинформатике и статистика, либо их приложения в биологии: сборка геномов, анализ данных NGS (Next Generation Sequencing), анализ молекулярных последовательностей. Алгоритмы включают в себя всё от общеинформатических строковых алгоритмов и марковских моделей до специализированных алгоритмов, применяемых при выравнивании геномов или при построении филогенетических деревьев. Статистика длится весь год и охватывает всё от классических методов проверки гипотез до statistical learning — раздела машинного обучения. Анализ данных NGS преподает Андрей Пржибельский, один из создателей геномного сборщика SPAdes, поэтому существенная часть курса посвящена различным подходам к сборке генома и написанию мини-ассемблера на основе графа де Брюйна [12].
Еще один предмет весеннего семестра — сборка геномов — это скорее сборная солянка из разных биоинформатических методов, о которых рассказывают эксперты из соответствующих областей: сборка геномов (Антон Банкевич, у которого совсем недавно вышла об этом статья в Nature Methods [13]), иммуносеквенирование (Яна Сафонова), протеомика (Кира Вяткина) и даже natural products discovery — раздел, занимающийся поиском химических соединений, обладающих антибактериальной, противовирусной или противораковой активностью и синтезированных живыми организмами. Например, недавно открытый многообещающий антибиотик теиксобактин можно отнести к таким natural products, но его открыли при помощи специально сконструированного устройства под названием iChip, которое изображало почвенные условия, необходимые для некультивируемых бактерий — так удалось «поймать» антибиотик [14]. Но подобное открытие — скорее удача: можно поставить сотню экспериментов и ничего не найти. Поэтому существуют вычислительные методы, позволяющие провести скрининг соединений, для которых в базах данных есть масс-спектры, поискать в них определенные мотивы и предсказать их свойства — и только потом искать в лаборатории, но уже сузив рамки. Этой теме посвящен один из студенческих научных проектов 2016 года, его задача — научиться находить часто встречающиеся мотивы (определенный фрагмент химической молекулы — несколько аминокислот или конфигурация ковалентной связи) в данных масс-спектроскопии, характерные для natural products и не встречающиеся в других классах соединений.
Но вернемся к обучению: у двух групп есть единственный общий курс — журнальный клуб, где каждую неделю биологи в паре с информатиками разбирают до пяти научных статей по одной теме и делают по ним доклад. Темы самые разные: single-cell-секвенирование [15], изучение предковых геномов, анализ альтернативного сплайсинга биоинформатическими методами и многое другое. Такой формат учит разбираться в методах, описанных в статьях, искать логику в проведенных экспериментах и расширяет кругозор в современной необъятной биоинформатике.
Привычных зачетов или экзаменов в ИБ нет. Оценки ставятся по результатам работы на протяжении семестра: обычно это домашние задания, небольшие тесты на занятиях и промежуточные тесты. В целом, по личным ощущениям автора, такая система обучения работает куда лучше университетской, где за пару-тройку дней до экзамена наконец открываешь учебники и пару-тройку дней и ночей напролет учишь.
Как всё устроено: студенческие научные проекты
Это очень важно. Если честно — это самое главное, что отличает ИБ от других мест, где учат биоинформатике. Это те навыки, которые вы не получите, даже прослушав самые крутые лекции по биоинформатике. Проект — это то, чему вы будете посвящать целые бессонные ночи и все обеденные перерывы, если он по-настоящему вас заинтересует.
Если говорить более формально, то в начале каждого семестра проходят презентации проектов, где будущие руководители коротко рассказывают о задачах и целях своего проекта. После презентаций неделя отводится на то, чтобы пообщаться с руководителями, выбрать из примерно 40 проектов три самых подходящих и довериться случаю — окончательно ваш проект определят кураторы (приоритеты почти всегда учитываются). После этого 3-4 месяца всё свободное время вы посвящаете ЕМУ, и дважды представляете свои результаты — на промежуточной и финальной защитах проектов (рис. 11). Чему обычно посвящены проекты? Как правило, это небольшая, но реальная и четко сформулированная биоинформатическая проблема, которую возможно решить за короткий срок. Для нее может существовать решение, которое только нужно реализовать, а может и не существовать (здесь речь чаще о поиске алгоритма, чем занимаются информатики).

Рисунок 11. Защита проекта «Поиск иммуногенных фрагментов на белковых последовательностях»
Например, поиск генов, относящихся к MHC-региону (главный комплекс гистосовместимости — гены, отвечающие за презентацию антигенов Т-лимфоцитам на поверхности клеток и развитие иммунного ответа), — в разные годы разные студенты нашли гены MHC у гепарда, дельфина, панды и альпаки. Или нахождение оптимального клеточного базиса для сборки транскриптома: в каждой ткани транскрибируются разные гены, и набор РНК в печени очень далек от набора РНК в мозге. Для некоторых задач требуется получить наиболее полный набор РНК-транскриптов организма (мРНК, по которым на этапе подготовки библиотек к секвенированию синтезируют кДНК — а уже ее секвенируют), но проанализировать все ткани человека (которых, по некоторым данным, минимум 44, не говоря уже о клеточных типах [16]) и посчитать в них транскрипты — непросто с вычислительной точки зрения, и для прикладных задач хорошим решением будет выбрать несколько тканей, РНК-транскрипты из которых будут наиболее полно перекрываться с набором мРНК всего организма. Грубо говоря, кроме генов домашнего хозяйства, работающих во всех тканях, важно учесть, что в мозге, например, много уникальных транскрибирующихся генов, которые «молчат» в других тканях, поэтому при анализе транскриптома человека важно учесть мРНК из мозга, а если посчитать уникальные транскрипты в фаллопиевых трубах, то их окажется немного. То есть, не учитывая фаллопиевы трубы, мы ничего серьезного не потеряем (если только не изучаем нарушение экспрессии какого-то специфичного гена именно в них), зато сильно сэкономим вычислительные силы.
Такие проекты хорошо вписаны в биологический контекст, но есть и проекты, требующие исключительно алгоритмического подхода: это проблемы сборки геномов и транскриптомов (например, обход пути в графе) или выравнивание нуклеотидных и аминокислотных последовательностей. На сайте ИБ есть краткие аннотации и презентации студенческих проектов — можно заметить, что круг тем никак не ограничивается, и есть шанс, что студенту попадется тема, которой он продолжит заниматься в будущем или напишет по ней дипломную работу. Руководителем проекта может быть преподаватель ИБ, бывший выпускник или любой человек, у которого есть интересная задача для студента. Например, студенты делают проекты в таких компаниях, как Parseq Lab, биомедицинский холдинг «Атлас» [17], iBinom, BIOCAD, а также сотрудничают с Центром алгоритмической биотехнологии СПбГУ (бывшая лаборатория Академического университета РАН), Калифорнийским, Университетом Вашингтона в Сент-Луисе, Институтом Кюри в Париже, Центром «Фундаментальные основы биотехнологии» РАН в Москве и многими другими.
Инфраструктура: какие еще проекты есть у ИБ
Годовая программа в ИБ — не единственный (хоть и самый эффективный) способ научиться биоинформатике. Помимо этого есть еще летняя школа, семинары, воркшоп и открытые лекции — всё это призвано развивать научное биоинформатическое сообщество в России и привлекать в него новых людей. Аудитория у этих проектов та же — в основном это биологи, осознавшие, как биоинформатика может помочь в исследованиях, и информатики, которым интересно решать реальные биологические проблемы, а не кодить сферических коней в вакууме, а также примкнувшие к ним физики и медики. Давайте обо всём по порядку.
Летняя школа по биоинформатике (начальный уровень)
В 2016 году школа проводится четвертый раз; в ней примут участие 100 человек — половина биологов/медиков и половина информатиков/математиков. Школа длится 6–7 дней, в программе — лекции по системной биологии, сборке и анализу данных геномов и транскриптомов, метагеномике, семинары по статистике и программированию на R и Python. Участники, у которых уже есть небольшой бэкграунд в биоинформатике, могут попробовать себя в работе над проектом. Впрочем, новички тоже успешно участвуют в проектах, примеры которых можно посмотреть на страничке школы 2014 года (с презентациями и иногда даже кодом на github). В этом году фокус школы — медицинская биоинформатика; до 1 мая 2016 года принимаются заявки на участие, и это отличный шанс, если вы еще не определились, связывать ли с биоинформатикой свою карьеру. Попробуйте!
Алла Лапидус рассказывает про развитие и применение биоинформатики
Мария Логачева — о разных способах секвенирования нового поколения
Если эти лекции показались вам слишком легкими или вводными, по ссылке можно найти много более специализированных видеолекций про статистику, транскриптомику, регуляцию экспрессии генов и многое другое со школы 2014 года.

Рисунок 12. Участники первой летней школы практически в полном составе
Интенсив по геномной биоинформатике (средний уровень)
Раньше интенсивы проводились осенью и весной в Москве и Петербурге, и на них был конкурсный отбор. Теперь в Петербурге запись на интенсив открыта постоянно, и его дата назначается, когда наберется необходимое количество участников. Интенсив полезен тем, кому необходимо работать с геномными данными. Во время знакомства участников можно услышать типичную фразу: «Я из лаборатории N, мы купили секвенатор Illumina MiSeq и не знаем, что с ним делать». Правда, что делать с самим секвенатором, здесь не рассказывают, но показывают, что делать с данными, которые он произвел (рис. 13). Это последовательные этапы от оценки качества прочтений, их сборки или выравнивания до поиска в них нуклеотидных замен и других мутаций. Например, как собрать геном de novo, проще показывать на бактерии, так как геном кого-то более крупного обычный ноутбук за приемлемое время пока не собирает. А искать мутации можно и в геноме человека — на последнем интенсиве на практике нужно было найти мутации, связанные с развитием муковисцидоза в экзомах восьми пациентов и предсказать, в какую категорию пациент попадает: больной, здоровый или носитель мутации. Задача нетривиальная, так как правильные диагнозы ставились только в 40% случаев. Чтобы не теряться, для участия в интенсиве нужно пройти онлайн-курс по базовому владению «Линуксом» — это поможет запускать многочисленные биоинформатические программы и разбираться с тем, что они выдают.

Рисунок 13. Антон Брагин (выпускник ИБ, ныне — Parseq Lab) пугает биологов кодом проводит практику по приложению биоинформатики в генетике человека
Выездной семинар по системной биологии (продвинутый уровень)
По сравнению с первыми двумя мероприятиями этот семинар — более продвинутый уровень, и уже в первый день участники погружаются в РНК-секвенирование и анализ данных экспрессии, например. Обычно темы охватывают анализ транскрипционной и эпигенетической регуляции, медицинскую и популяционную генетику, то есть поиск различных вариаций в геномах, а также введение в метаболомику. Семинар проводится за городом (рис. 14), вдали от шума и искушений, способных отвлечь вас от системной биологии. Специальный гость в 2016 году — Марк Дейли из Broad Institute (а также из Harvard University и Massachusetts General Hospital — вы удивитесь, сколько у него аффилиаций). Постоянные спикеры — Никита и Максим Артемовы из Harvard University и Washington University in St. Louis соответственно, а также Александр Предеус, ставший с 2015 года директором по исследованиям в ИБ и курирующий семинар идеологически [18]. Проводить практику помогают биоинформатики из лаборатории «Алгоритмы сборки геномных последовательностей» в Университете ИТМО, и это здорово помогает интегрировать теоретические знания в долговременную память (если вы так не считаете, значит вы ни разу не запускали биоинформатическую программу).

Рисунок 14. На выездном семинаре по системной биологии в 2015 году
И еще многое другое...
Наверное, вы уже заметили, что преподаватели Института биоинформатики создают онлайн-курсы на Стэпике. Полный список можно найти на соответствующей страничке; многие курсы открыты без дедлайнов — это значит, что можно пройти курс в любое удобное время и получить сертификат. Эти курсы — для начинающих биоинформатиков, как для биологов, которые хотят познакомиться с Linux, Python или статистикой и R, так и для программистов, начинающих изучать молекулярную биологию. Более продвинутые курсы по программированию, алгоритмам и математике делают в Computer Science Centre (CSC) и Академическом университете (СПбАУ).
Время от времени в ИБ проходят гостевые лекции на самые разные темы, расписание которых есть на сайте, но самый надежный способ не пропускать анонсы — подписаться на рассылку.
Своя наука: чем занимаются внутри Института биоинформатики
С 2015 года у ИБ, как у любого уважающего себя исследовательского института, есть собственное научное подразделение, возглавляемое Александром Предеусом, о биографии и приключениях которого можно почитать в его же статье. Если раньше большинство проектов было посвящено именно геномной биоинформатике (так уж сложилось, в Петербурге это направление процветало благодаря Центру геномной биоинформатики им. Ф.Г. Добржанского и лаборатории алгоритмической биологии Академического университета РАН), то сейчас в ИБ появляются люди, занимающиеся обработкой данных RNA-seq, СhIP-seq, микрочипов, что позволяет изучать экспрессию на геномном уровне. У ИБ появились друзья из лаборатории «Алгоритмы сборки геномных последовательностей» в ИТМО и из «Биобанка» при СПбГУ. Каждые две недели в институте проходят научные встречи, на которых люди, занимающиеся биоинформатикой в Петербурге, знакомятся, рассказывают про свои исследования, и обсуждают новости научной литературы.
Наш приоритет, помимо собственно научной работы, — создание сети экспертов-биоинформатиков, которые знают друг друга и способны эффективно помогать биологам и медикам в рамках города. Конечной целью исследования для нас всегда являются прикладные биологические или медицинские проблемы, и этот принцип соблюдается в каждом из наших активных проектов.
Среди проектов — анализ и квантификация транскриптомных экспериментов (студенческая работа осеннего семестра), поиск решения для деконволюции клеточных типов по данным экспрессии (разработка программы, которая «вслепую» будет находить клеточные типы в данных, где имеются только имена генов и уровень их экспрессии, — проект преподавателя ИБ Константина Зайцева) и другое. Здесь мы подробнее расскажем про некоторые из них.
GeneQuery
Один из таких проектов — разработка веб-сервера GeneQuery для генерации биологических гипотез на основании данных экспрессии — развивается еще со времен работы Александра в университете Вашингтона в Сент-Луисе. Не переключайтесь: представьте, что после проведения эксперимента (того же микрочипа) вы получаете список генов и знаете, что в вашем исследуемом объекте именно этот набор генов экспрессируется сильнее, чем в норме. Десять лет назад можно было смотреть на этот список глазами, читать литературу и думать, что бы все это могло означать. Сейчас думать все равно придется, но есть и другие зацепки.
В альтернативном подходе исходят из предположения, что гены, ко-экспрессирующиеся в каких-либо условиях, являются также ко-регулируемыми. Такой постулат позволяет нам использовать кластеры ко-экспрессирующихся генов для поиска биологических параллелей между экспериментами — вашим и неизвестным экспериментом из нашей глобальной базы данных. Это, в свою очередь, естественным образом ведет к интересным биологическим гипотезам. Например, если ваш эксперимент — гены из образца раковой опухоли, а найденный результат соответствует экспрессии макрофагов, возможно, в вашем образце происходит инфильтрация этих иммунных клеток внутрь опухоли.
Итак, за основу была взята крупнейшая база данных с экспериментами на микрочипах — GEO (Gene Expression Omnibus), где находятся результаты десятков тысяч экспериментов по экспрессии генов (на микрочипах, с помощью ChIP-seq или RNA-seq). Для создания GeneQuery отобрали подходящие по количеству образцов данные для мыши, крысы или человека, в каждом наборе выбрали около 6000 отчетливо экспрессирующихся генов и кластеризовали. При кластеризации гены в эксперименте разбиваются на связанные регуляцией группы, и уже по ним можно осуществлять поиск, в качестве запроса используя набор генов, в котором вы заинтересованы. На выходе GeneQuery вы получаете ссылки на эксперименты, в которых встречается похожий паттерн экспрессии такой группы генов, и статистику, по которой можно судить о достоверности, «степени уверенности» веб-сервера в этом результате (рис. 15).

Рисунок 15. Так выглядит результат работы GeneQuery. a — Таблица со всеми экспериментами, в которых найдено похожее сочетание или поведение вашего набора генов. Если нажать на цифру в колонке Module, появится heatmap экспрессии модуля, куда попали ваши гены — б. Далее ко колонкам: уровень значимости, Overlap (количество перекрывающихся генов в эксперименте и вашем списке) и ссылки на базу данных GEO и на скачивание датасета.
Подобные подходы применяли и раньше, но всегда только с базами данных заранее отобранных наборов генов — из KEGG, Reactome или MsigDB. Такой отбор требует огромных усилий кураторов, а полученные наборы обязательно устаревают — биология несется вперед на всех парах. К тому же теряется способность отразить биологическое разнообразие: например, гамма-интерфероновый ответ на вирусную инфекцию в клетках печени и в клетках эпителия может сильно различаться, а стандартный набор генов будет в обоих случаях один.
Попробуйте сами: в разделе примеров вы можете запустить поиск типовых генов, экспрессия которых повышается при гипоксии. В результатах можно найти раковые клетки, эксперименты по обработке химическими веществами, имитирующими гипоксию, или собственно эксперименты по ограничению кислорода. Для того, чтобы поиск по такой огромной базе работал эффективно, нужно немалое мастерство в дизайне и выполнении сложных программных проектов. Этой работой занимался Иван Арбузов, студент магистратуры ИТМО. В результате сервер сильно усовершенствовался — каждый запрос занимает не более нескольких секунд, поддерживается корректное преобразование ортологов между видами (то есть по генам человека можно искать эксперименты с мышью, и наоборот), можно использовать любые из четырех широко распространенных типов идентификаторов генов. Добро пожаловать!
Банк Экзомов
Еще одно направление ИБ — сотрудничество с РЦ «Биобанк» при СПбГУ, связанное с обработкой и анализом экзомных данных. При полноэкзомном секвенировании (WES, whole-exome sequencing) прочитывается примерно 1–2% генома человека, приходящиеся на белок-кодирующие регионы, — это отнимает меньше ресурсов и дает большее покрытие, чем полногеномное секвенирование (рис. 16). Более того, такие данные крайне актуальны для клиники, и, хотя уже давно известно, что болезни отнюдь не всегда вызываются мутациями в белок-кодирующих регионах [19], мутации в экзонах намного лучше изучены и проще интерпретируются.

Рисунок 16. При экзомном секвенировании анализируется лишь малая доля генома, но из нее можно получить максимум полезной информации.
сайт www.my46.org
В «Биобанке» стоит пока что единственный в Санкт-Петербурге секвенатор Illumina HiSeq 2500, позволяющий получать до 1 терабайта данных с одного запуска (а в ближайших планах — ввод в строй более мощного Illumina HiSeq 4000!). На нем просеквенировали экзомы нескольких десятков пациентов с эндокринологическими заболеваниями. Предстоит много работы по их анализу: от сравнения технологий приготовления библиотек перед секвенированием и нахождения оптимальной экспериментальной методики до построения полноценного пайплайна анализа данных и интерпретации вариантов — информации, которую можно будет предоставлять докторам. В планах также составление локальной базы вариаций, которая облегчит поиск клинически важных вариантов в экзомах пациентов и постановку диагноза.
ChIP-seq на IonTorrent
В еще одном проекте с группой Дмитрия Тентлера (Институт цитологии РАН) проводится мета-анализ экспериментов ChIP-seq для транскрипционных факторов группы Nf-kb. Одно из замечательных качеств полногеномных экспериментов заключается в том, что можно не делать дорогостоящих экспериментов в лаборатории, а взять данные, уже полученные кем-то ранее, и получить новые научные результаты. Используя открытые данные по эпигенетическому профайлингу (эксперименты ChIP-seq для различных гистонных модификаций), аннотируются состояния хроматина, после чего становится возможным проанализировать связывание факторов Nf-kb со своими целевыми последовательностями в зависимости от доступности хроматина. В данном случае мета-анализ помогает лучше спланировать собственный эксперимент.
Интересной особенностью проекта является экспериментальная часть: аспирант СПбГУ и студент Института биоинформатики Николай Панюшев работает над тем, чтобы сделать ChIP-Seq человеческих клеточных линий при помощи IonTorrent. Несмотря на известные недостатки этого прибора, в случае, когда его работа в лаборатории налажена, производительности вполне достаточно для среднестатистического эксперимента ChIP-seq на клетках человека или мыши. Насколько мне известно, такие работы в России еще никто не проводил.
Жизнь после Института биоинформатики
Среди выпускников, продолжающих работать в биоинформатике, есть и биологи, и информатики. Последних, конечно, больше, но и биологам часто удается совместить свои лабораторные исследования с вычислительной биологией или биоинформатикой.
Я училась на матмехе СПбГУ по специальности «Прикладная математика и информатика». На третьем курсе нужно было определяться с темой курсовой и диплома, и мой научный руководитель предложил бионформатический проект. Тогда я совсем не знала, что это. Мне понравилась тема, я узнала, что в Санкт-Петербурге есть курсы и летняя школа по биоинформатике, и решила попробовать. В итоге попала и на школу, и на курсы, и знания, полученные на них, так контрастировали с формальной, аккуратной и неизменной математикой, которая была на матмехе, что я была в восторге! Поняла, что биология — это то, к чему я хочу приложить свои умения. В ИБ потрясающие преподаватели, которые помогали мне с биологией и программированием и вдохновляли на учебу, — я даже не думала о том, чтобы пропускать пары. Когда есть проект, который тебе интересен, все навыки начинают иметь смысл и, кажется, начинается настоящая учеба. Я хотела продолжить заниматься биоинформатикой, встретила своего текущего научного руководителя, который предложил мне работу, и я согласилась. Сейчас я visiting scientist в WashU и занимаюсь системной биологией — анализом экспрессии генов, изучением предсказаний и объяснений ответа рака на иммунотерапию. Находиться в среде иммунологов, видеть, как проводят настоящие эксперименты, формулируют гипотезы, планируют исследования, встречаться с крупными учеными — интересно. Два года назад, когда я начинала, я не могла даже мечтать о таком. И еще хочу подчеркнуть, что все это было бы сложно провернуть без тех людей, которых я встретила, которые мало того, что любят свою работу, так еще и активно помогают другим.
С Институтом биоинформатики я познакомился в 2013 году, когда практически случайно подал заявление на участие в первой летней школе по биоинформатике, узнав о ней от коллег. Смысл поездки был в том, чтобы залатать прореху в компетенциях в нашей лаборатории — у нас назревало несколько биоинформатических проектов, которые предстояло кому-то делать, но никто не знал как. В некотором роде, поездка на летнюю школу стала для меня способом расширения круга доступных мне навыков. Разумеется, за неделю летней школы едва ли можно приобрести сколько-нибудь серьезные познания в биоинформатике, однако там я узнал, что можно продолжить обучение в Институте биоинформатики, который открыл набор на программу для биологов в 2013 году.
Я сразу же воспользовался возможностью поступить туда. Навыки, приобретённые мной за год, позволили приступить к проекту, которым я занимаюсь и сейчас, а именно изучением изменения экспрессии генов при образовании нерегулярных меристем у растений с помощью транскриптомного секвенирования. Если объяснять простыми словами, мы смотрим, что происходит при образовании новых точек роста там, где им вообще-то не положено образовываться. Так бывает, если растение заражает какой-нибудь патоген или образуются опухоли, обусловленные генотипически. Мы изучаем оба этих варианта. Помимо собственной работы, я также немного помогаю коллегам, если необходимо применить какой-либо биоинформатический метод (чаще всего, это сводится к поиску ортологичных генов у разных видов и изучению филогении).
Что касается самого Института биоинформатики, то я по-прежнему связан с ним, но теперь уже в статусе преподавателя. У меня была возможность наблюдать за развитием ИБ в динамике, и перемены в институте очень внушительны. Учебные программы за два года усовершенствовались настолько, что я практически жалею, что не поступил хотя бы на год позже :) Неотъемлемой частью обучения в ИБ является выполнение научно-исследовательских работ, и с каждым годом они становятся все сложнее и интереснее. Хотя проекты являются частью учебной программы, их трудно назвать игрушечными, так как многие из них вполне дотягивают до уровня серьезных публикаций.
Я закончил бакалавриат в ИТМО по направлению прикладной математики и стал думать, чем заниматься дальше. Тогда я даже не рассматривал карьеру в науке, но хотел научиться программировать, а от друзей услышал, что лучших программистов делают в АУ . Из предлагаемых программ SE (software engineering) мне казалась «слишком простой» (я, кстати, ошибался), а CS (computer science) — сложной (тут я был прав). Поэтому, я и выбрал биоинформатику, она мне казалась чем-то средним между теорией и практикой. Слушать разнообразные курсы и параллельно заниматься биоинфоратическими проектами было очень познавательно (хотя и тяжеловато). Благодаря же проектам я стал знакомиться с миром науки и достаточно быстро понял, что это — мое. Начиная со второго года обучения я начал проект по референсной сборке под руководством тогдашнего PhD-студента из UCSD. Проект перерос «обычную дипломную работу» — мы попали на одну из главных биоинформатических конференций, а теперь участвуем с ним в большой международной коллаборации (ENCODE) [20]. С таким багажом опыта и знаний я поступил в UCSD, где сейчас и учусь уже второй год. Тут я попробовал другие биоинформатические области (к примеру, протеомику [21]), но продолжаю заниматься и сборкой генома (в рамках старых и новых проектов) [22].
Санкт-Петербургскому Академическому университету РАН институт отчасти обязан своим существованием — в 2010 году все началось с курсов по биоинформатике на базе АУ, а сейчас АУ и ИБ реализуют совместную магистерскую программу по алгоритмической биоинформатике.
Летом после окончания бакалавриата Физического факультета СПбГУ я случайно наткнулась на сайт лаборатории алгоритмической биологии СПбАУ РАН и через него вышла на сайт Института биоинформатики. Мне понравилось решать вступительные задачки на Rosalind, я прошла собеседование и поступила. В моей жизни было много счастливых случайностей, и это одна из них. Преподаватели ИБ — безумно заинтересованные люди, мне, как и им, захотелось читать научные статьи и программировать с утра до позднего вечера. Я занималась биоинформатикой на перерывах в университете и дома, а по субботам у нас были самые интересные лекции и практические семинары из всех, что я когда-либо слышала. Следующим летом, в рамках обучения в ИБ, я попала на стажировку в лабораторию, на сайт которой я когда-то случайно вышла. Так сложилось, что теперь я работаю в Центре алгоритмической биотехнологии СПбГУ (по сути, это та же лаборатория) и пишу диссертацию под руководством Андрея Пржибельского. В нашей лаборатории — нескончаемые возможности участия в новых научных проектах, мы сами выбираем направления наших исследований. Я работаю с NGS-данными молекул РНК. Сложность анализа данных RNA-seq заключается в неравномерном покрытии, вызванным различными уровнями экспрессии изоформ и генов. De novo сборки таких данных помогают находить новые fusion-гены, в которых заинтересованы ученые, занимающиеся исследованиями раковых заболеваний. Мне важно применять свои знания в решении задач, полезных обществу, и сейчас я могу заниматься ими благодаря обучению в ИБ.
Я попала в ИБ, по большому счету, благодаря удачному стечению обстоятельств: на летней школе Future Biotech случайно услышала про набор на «Биоинформатику для биологов», сразу подала туда заявку, и, что самое удивительное, поступила. Слово «биоинформатика» я тогда уже знала: в магистерскую программу биотехнологов входил курс с таким названием, но этой областью самой по себе не интересовалась, целью поступления было, скорее, изучение математики и статистики плюс повышение компьютерной грамотности. Общение с преподавателями и другими студентами ИБ, обсуждение статей и научных проектов заставили взглянуть на предмет с совершенно иной стороны: меня поразило то, насколько широка эта область, насколько разные возможности для применения знаний она даёт и насколько интересные результаты можно получать. Думаю, здесь стоит сказать «спасибо» всем преподавателям и организаторам курсов за чудесную возможность познакомиться с биоинформатическим сообществом, расширить кругозор и найти те задачи, которые хотелось бы научиться решать. Собственно, дальше все достаточно банально: уже в ходе обучения поняла, что хотела бы применять новые знания, и с радостью согласилась, когда однокурсник позвал работать к ним в компанию. Сейчас работаю в Parseq Lab, главным образом, над проектом, связанным с применением таргетного секвенирования в криминалистике (ещё два года назад и представить не могла, что занесет в такую область!). Самым интересным в этой работе остается постоянная необходимость развиваться, изучать что-то новое: от популяционной генетики до методов машинного обучения, от протоколов пробоподготовки до программирования. Думаю, такая работа не стала бы для меня возможной без тех базовых знаний, которые дали в ИБ, и тех людей, с которыми довелось там познакомиться.
Грани биоинформатики: вместо заключения
Если читатель еще жив, то коротко опишем две истории из мира биоинформатики. Любая вводная лекция про биоинформатику обязательно содержит слайд о том, что стоимость секвенирования стремительно падает и даже обгоняет закон Мура — уже скоро свой просеквенированный геном можно будет получить за 1000 долларов [23], [24]. Хранить геном в облачном сервисе и иметь к нему доступ с любого девайса называется персональной геномикой [25]. Эта область занимается изучением генетических данных человека с целью выявить отличительные черты его генома, влияющие на фенотип, риск развития заболеваний, метаболизм различных веществ (будь то глютен, кофе или алкоголь) и другое. И в персональной геномике постоянно происходят какие-то казусы. Самая известная американская компания 23andme, с 2006 года разрабатывающая и продающая генетические тесты, проводила скрининг предрасположенности более чем к сотне заболеваний, до тех пор, пока FDA в 2013 году не запретило компании продавать тесты.
Управления по контролю за продовольствием и лекарственными препаратами США — это такое злое министерство в США, которое постоянно что-то запрещает: лекарства, продукты питания, генетически-модифицированную еду. До некоторых пор 23andme просто игнорировали предупреждения FDA о том, что пора бы перестать просто так предсказывать людям риск развития тяжелых заболеваний, иначе это может привести к необдуманным и опасным решениям клиентов. Но потом все пошло не так. Совпадение? Не думаем.
Позже стало известно, что компания все-таки получила право тестировать клиентов на одно заболевание — синдром Блума, так как его генетические детерминанты хорошо известны. Вам интересно, что представляет собой синдром Блума? В патологии синдрома фигурируют склеродермия, микрогнатия, умеренный иммунодефицит, гипогонадизм и иногда даже умственная отсталость. Есть подозрения, что имея в наличии этот синдром, клиент заподозрит, что с ним что-то не так, и без проведения генетического скрининга. На сегодняшний день список заболеваний, скрининг которых можно провести, находясь в США, расширился, а в Канаде, Великобритании и Европе по-прежнему доступно тестирование более сотни различных состояний .
В России сейчас тоже можно пройти генетическое тестирование. Про то, как критически относиться к таким тестам и какие есть возможности и компании на российском рынке читайте в обзоре «Генетическое тестирование и патернализм в медицине» [26] и в статье о биомедицинском холдинге «Атлас» [17].
Подобно этой истории еще один бум (уже в спортивной геномике) произошел, когда открыли ген альфа-актинина-3 (ACTN3), кодирующий белок быстросокращающихся мышечных волокон. Определять вариант этого гена предлагалось у детей от самого рождения до 8 лет, чтобы вовремя обнаружить задатки первоклассного футболиста или спринтера на пути к олимпийским победам — и толпы родителей уверовали в то, что гены не могут врать: сказано в них бегать — значит, бегать (рис. 17) [27]. В 2003 году вышло целое исследование о связи этого гена со спортивными достижениями [28]. В нем участвовало 429 атлетов, включая 50 олимпийских чемпионов, и какие-то корреляции, конечно, обнаружили: 25% спортсменов имели две копии ACTN3 (однако в контрольной группе — 18%). Но все же нашли одного испанского атлета и олимпийского чемпиона без единственной копии альфа-актинина-3, и его судьба могла бы сложиться иначе, если бы родители в свое время полагались на генетическое тестирование.

Рисунок 17. Кадр из фильма «Гаттака», который рекомендуем обязательно посмотреть, если еще не видели (никаких спойлеров)
Уже позже Карл Фостер (один из авторов открытия ACTN3, кстати) предлагал другое решение: «Просто поставьте школьников на беговую дорожку и посмотрите, кто прибежит первым». Но, несмотря на это, тест по-прежнему популярен. А совсем недавно вышла работа, в которой искали генетические детерминанты, обуславливающие выносливость и аэробные способности спортсменов мирового класса [29]. Среди 45 наиболее перспективных маркеров, замеченных в связи с выносливостью (в основном, гены, ответственные за функции кардиореспираторной системы), не было обнаружено ни одной комбинации вариантов, которая стабильно встречалась бы у всех спортсменов. Только вариация в одном гене — GALNTL6 — оказалась статистически значимой, но функция этого гена до сих пор не выяснена, и известно только то, что он экспрессируется в семенниках, мозге и скелетных мышцах.
Но сейчас самое время разбавить эти курьезы более мрачными историями, например, о том, как применяется биоинформатика в клинической практике. Одна из сложнейших клинических проблем — терапия гетерогенных опухолей [30], в которых клетки отличаются по разным параметрам, но для нас сейчас важно, что они отличаются генотипически. При заборе биопсии в пробу попадают далеко не все типы клеток, и даже если гетерогенность изучается целенаправленно, то используемые методы достаточно трудоемки (FISH, кариотипирование, сравнительная геномная гибридизация). В процессе химиотерапии в гетерогенных опухолях из-за эффекта бутылочного горлышка возникает множественная устойчивость к лекарствам (клетки, чувствительные к лекарствам, умирают, а устойчивые пролиферируют и образуют новую опухоль), это можно отслеживать, проводя биопсию после каждого курса химиотерапии, однако это не дает ответа на вопрос — как вообще развивается такая опухоль? Биоинформатика ищет ответы на комплексные вопросы, используя методы вроде single-cell-секвенирования [15]. В одной из работ по целой сотне клеток из опухоли груди построили настоящее филогенетическое дерево, чтобы выяснить структуру и эволюцию опухоли (рис. 18). В итоге выявили четыре эволюционные ветви, составляющие три крупные субпопуляции [31].
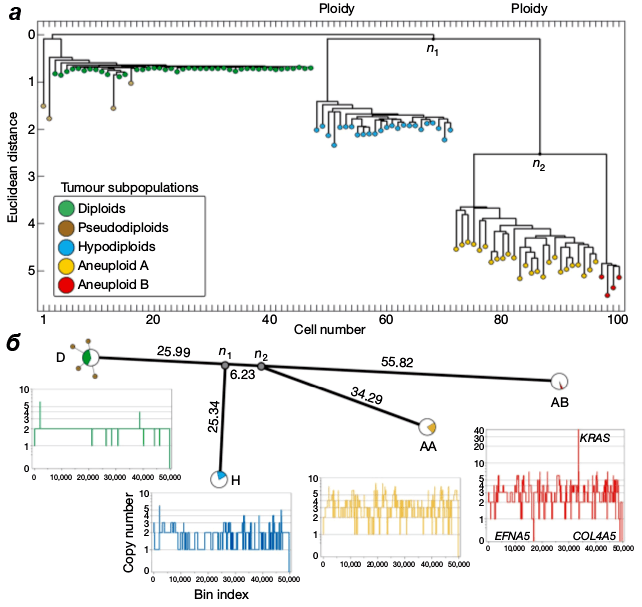
Рисунок 18. Из разных секций опухоли взяли 100 клеток, выделили из них ядра и пометили их флуоресцентным красителем DAPI, что позволило отсортировать клетки с разной плоидностью с помощью проточной цитометрии. Далее в них были найдены вариации числа копий (CNV) — крупные хромосомные перестройки, такие как дупликации и делеции, и по ним подсчитаны матрицы расстояний. а — В итоговом дереве клетки разделились на 3 субпопуляции: диплоидные и псевдодиплоидные (с крупными перестройками), гаплоидные (где произошла потеря одного набора хромосом) и анеуплоидные или субтетраплоидные (где вроде все дуплицировалось, но как попало). б — Дерево расстояний, на котором видно, что от диплоидных клеток (когда-то бывших здоровыми) до первого предка остальных субпопуляций (n1) прошло много времени, а до второго предка (n2) — совсем мало. Ветвь AB, куда попадают клетки с массивными геномными дупликациями (субтетраплоидные) дальше всего зашла в эволюции от диплоидных предшественников, и в них обнаружена 50-кратная амплификация онкогена KRAS.
И тот факт, что стоимость чтения генома стремительно падает, приобретает уже совершенно иные черты. Конечно, это далеко не полный спектр направлений, существующих в биоинформатике, а только самые отличительные и популярные. В один из дней вы легко можете обнаружить себя в области, о которой вряд ли задумывались ранее: например, присоединитесь к группе, изучающей геном Людовика XVI — короля Франции, кровь которого сохранилась в древней вазе после его казни [32]. Или будете собирать геном дикорастущего и окультуренного ананасов, чтобы узнать интересные факты об эволюции CAM-фотосинтеза [33].
На какую сторону переходить — выбирать вам.
Литература
- Калиевый канал in silico;
- Биоинформатика в мире РНК-структур;
- Драг-дизайн: как в современном мире создаются новые лекарства;
- ACSN — глобальный атлас сигнальных путей. От молекулярной географии рака к новым информационным технологиям в биологии;
- Zeeberg B.R., Riss J., Kane D.W., Bussey K.J., Uchio E., Linehan W.M. et al. (2004). Mistaken identifiers: gene name errors can be introduced inadvertently when using Excel in bioinformatics. BMC Bioinformatics. 5, 80;
- Код жизни: прочесть не значит понять;
- CRISPR-системы: иммунизация прокариот;
- Мутагенная цепная реакция: редактирование геномов на грани фантастики;
- Вылечить миодистрофию Дюшенна: конкуренция групп, единство методик;
- Успех в борьбе с лейкозом: на шаг ближе к клиническому применению геномного редактирования;
- Dobrynin P., Liu S., Tamazian G., Xiong Z., Yurchenko A.A., Krasheninnikova K. et al. (2015). Genomic legacy of the African cheetah, Acinonyx jubatus. Genome Biol. 16, 277;
- Compeau P.E., Pevzner P.A., Tesler G. (2011). How to apply de Bruijn graphs to genome assembly. Nat. Biotechnol. 29, 987–991;
- Bankevich A. and Pevzner P.A. (2016). TruSPAdes: barcode assembly of TruSeq synthetic long reads. Nat. Methods. 13, 248–250;
- Ling L.L., Schneider T., Peoples A.J., Spoering A.L., Engels I., Conlon B.P. et al. (2015). A new antibiotic kills pathogens without detectable resistance. Nature. 517, 455–459;
- Секвенирование единичных клеток (версия — Metazoa);
- Uhlén M., Fagerberg L., Hallström B.M., Lindskog C., Oksvold P., Mardinoglu A. et al. (2015). Proteomics. Tissue-based map of the human proteome. Science. 347, 1260419;
- Биомедицинский холдинг Atlas как кубик Рубика;
- Как перестать бояться и полюбить системную биологию;
- Ward L.D. and Kellis M. (2012). Interpreting noncoding genetic variation in complex traits and human disease. Nat. Biotechnol. 30, 1095–1106;
- Сколько сора в нашей ДНК;
- Kolmogorov M., Liu X., Pevzner P.A. (2016). SpectroGene: a tool for proteogenomic annotations using top-down spectra. J. Proteome Res. 15, 144–151;
- Kolmogorov M., Raney B., Paten B., Pham S. (2014). Ragout — a reference-assisted assembly tool for bacterial genomes. Bioinformatics. 30, i302–i309;
- Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур;
- Технология: $1000 за геном;
- От медицины для всех — к медицине для каждого!;
- Генетическое тестирование и патернализм в медицине;
- Macur J. (2008). Born to run? Little ones get test for sports gene. The New York Times;
- Yang N., MacArthur D.G., Gulbin J.P., Hahn A.G., Beggs A.H., Easteal S., North K. (2003). ACTN3 genotype is associated with human elite athletic performance. Am. J. Hum. Genet. 73, 627–631;
- Rankinen T., Fuku N., Wolfarth B., Wang G., Sarzynski M.A., Alexeev D.G. et al. (2016). No evidence of a common DNA variant profile specific to world class endurance athletes. PLoS One. 11, e0147330;
- Bedard P.L., Hansen A.R., Ratain M.J., Siu L.L. (2013). Tumour heterogeneity in the clinic. Nature. 501, 355–364;
- Navin N., Kendall J., Troge J., Andrews P., Rodgers L., McIndoo J. et al. (2011). Tumour evolution inferred by single-cell sequencing. Nature. 472, 90–94;
- Olalde I., Sánchez-Quinto F., Datta D., Marigorta U.M., Chiang C.W., Rodríguez J.A. et al. (2014). Genomic analysis of the blood attributed to Louis XVI (1754–1793), king of France. Sci. Rep. 4, 4666;
- Ming R., VanBuren R., Wai C.M., Tang H., Schatz M.C., Bowers J.E. et al. (2015). The pineapple genome and the evolution of CAM photosynthesis. Nat. Genet. 47, 1435–1442.











