
Марк Иванов: «Для чего протеомике нужно машинное обучение»
21 января 2025
Марк Иванов: «Для чего протеомике нужно машинное обучение»
- 257
- 0
- 0
Герой интервью в лаборатории Института энергетических проблем химической физики им. В. Л. Тальрозе.
Рисунок в полном размере.
личный архив Марка Иванова
-
Автор
-
Редакторы
Мы поговорили с Марком Ивановым — выпускником Физтеха, кандидатом физико-математических наук, ведущим научным сотрудником Института энергетических проблем химической физики им. В. Л. Тальрозе в составе Федерального научного центра химической физики им. Н.Н. Семенова РАН. Марк — представитель научной школы директора Института Михаила Горшкова, в которой уже много лет на высоком уровне ведутся исследования в области анализа данных биологической масс-спектрометрии. Совсем недавно наш собеседник стал лауреатом Научной премии Сбера в новой номинации «AI в науке. Науки о жизни». Мы узнали у Марка, какими проектами он занимался в последнее время, и почему протеомике понадобился искусственный интеллект.
DirectMS1 — оригинальный метод количественной оценки белков
— Первым делом я хочу узнать, что ты делал последние несколько лет, когда уже стал, большей частью, самостоятельным? Когда у тебя уже свои проекты появились. Что ты можешь по этому поводу сказать нашей «биомолекулярной» публике?
— Самое интересное из моего — это метод, который обеспечивает идентификацию белков по масс-спектрам первого уровня, который мы назвали DirectMS1 (рис. 1) [4]. Наш подход идет вразрез с тем, что обычно делается в области масс-спектрометрии белков, когда к масс-спектрам исходно анализируемых соединений добавляют еще спектры фрагментации. Я, как разработчик, верю, что он конкурентноспособен, и что так делать тоже правильно.

Рисунок 1. Алгоритм анализа данных в подходе DirectMS1. 1 — Данные хромато-масс-спектрометрии. 2 — Выбор характеристик. 3 — Предварительный поиск по MS1. 4 — Предсказание хроматографического времени удержания. 5 — Машинное обучение «на лету» методом градиентного бустинга. 6 — Конечный поиск. 7 — Результаты анализа.
— Масс-спектры первого уровня — это когда пептиды анализируют в масс-спектрометре с высоким разрешением без фрагментации?
— Да, это когда анализируют ионы целых пептидов: тут мы жертвуем фрагментацией и не получаем точную информацию об их последовательности. Поэтому с нашим подходом нельзя или гораздо сложнее изучать посттрансляционные модификации или мутации белков. Точно так же, пока нам сложно изучать, например, полутриптическую активность . Однако если вас интересует идентификация канонических белков как генных продуктов, не вдаваясь в протеоформы , их количественный анализ, то фрагментация пептидов, как мы показали, может быть не нужна. А самое главное, что за счет этого мы экономим масс-спектрометрическое время — самое дорогое в этой процедуре — и можем в рамках ограниченных ресурсов делать нормальные исследования.
В наиболее распространенном варианте протеомного метода bottom-up (то есть «снизу вверх») все белки в образце перед хромато-масс-спектрометрическим анализом расщепляют трипсином, который имеет высокую специфичность, оставляя на С-конце каждого полученного в результате пептида лизин или аргинин. Отклонения от этого правила создают проблемы при идентификации пептидов.
Совокупность природных белковых вариантов, кодируемых одним геном. Сюда входят варианты, возникшие в результате генного полиморфизма, альтернативного сплайсинга, посттрансляционных модификаций, протеазной активности и т.д.
— Ну то есть, у тебя, наверное, целевая аудитория состоит из двух противоположных частей. Первая — это бедные люди, у которых нет ресурсов на анализ; а вторая — кто очень много хочет: частные фирмы, работающие на фарму, на богатых клиницистов. Сейчас в профильных компаниях очень большой интерес к тому, чтобы за 10 минут работы масс-спектрометра делать один протеомный анализ. Правда, сейчас нишу твоего метода занимает DIA (data-independent acquisition) . Этот подход хорошо работает на таких быстрых приборах как timsTOF (масс-спектрометр фирмы Bruker) и, тем более, Astral (новейший прибор фирмы Thermo). Подтверждаю как пользователь первого, да и масс-спектры со второго я тоже недавно анализировал. Так вот, DIA дает достойные цифры по идентифицированным и количественно оцененным белкам даже за 10 минут, что сравнимо с твоим методом.
Панорамная протеомика началась с метода получения масс-спектров, который назывался зависимым от данных (Data Dependent Acquisition). В первичном масс-спектре выделяли наиболее интенсивные ионы, и в соответствии с этим фрагментировали именно эти ионы в узком интервале молекулярная масса/заряд (m/z). То есть, фрагментация ионов была задана данными первого масс-спектра. У этого метода имеется существенный недостаток. Во время фрагментации происходят потери попадающих в это время в масс-спектрометр первичных ионов. Поэтому у него неидеальная воспроизводимость между повторами, что отрицательно влияет на количественный анализ. Позднее были предложены методы получения масс-спектров, не зависимые от данных (Data Independent Acquisition), где на фрагментацию посылают все продукты, независимо от их интенсивности, в более широких, заданных интервалах m/z. В этом случае возникает сложность при определении того, к какому иону-предшественнику относятся фрагменты, хотя эти задачи решаются вычислительным путем. Зато количественный анализ методом DIA оказался значительно более воспроизводимым.
timsTOF — квадрупольно-времяпролетный масс-спектрометр высокого разрешения фирмы Bruker (основана в Германии, сейчас — США), в котором в одной вакуумной системе с масс-спектрометром перед ним установлен спектрометр ионной подвижности. Спектрометрия ионной подвижности основана на анализе движения ионов в направлении электрического поля против потока газа. Этот аналитический метод, например, используется в аэропортах для контроля запрещенных веществ.
Orbitrap Astral — новейший масс-спектрометр высокого разрешения фирмы Thermo (США), в котором ионная ловушка типа Orbitrap дополнена оригинальным масс-спектрометром, имеющим признаки времяпролетного. Высокая чувствительность Orbitrap в нем дополнена очень высокой скоростью получения тандемных масс-спектров.
— Вопрос, что сравнивать. Число идентифицированных белков зависит от корректного подхода к оценке уровня ложноположительных идентификаций (FDR, false discovery rate), с чем в DIA, на мой взгляд, есть проблемы. Также результаты любят сравнивать по коэффициенту вариации (CV), который в некоторых случаях ошибочно связывать с возможностями количественного анализа. Мы это доказали в одной из недавних публикаций [5]. Количественный анализ как таковой при сравнении DIA и DirectMS1, по нашим данным, в первом ничуть не лучше.
Наверное, из общих соображений следует, что если в процессе снятия масс-спектра не делать MS/MS , то жизнь станет гораздо проще. На самом деле, всё к тому и идет. Даже методы DDA сейчас во что эволюционировали? Всё измеряется с опцией Match Between Runs (сопоставление запусков системы), то есть фрагментация снимается не везде. Достаточно спектра фрагментации пептида в одном запуске, чтобы по масс-спектру первого уровня зарегистрировать его в других запусках, где он вовсе не фрагментировался.
MS/MS — получение «вторичных», тандемных спектров, или спектров фрагментации продуктов, в данном случае, пептидов в масс-спектрометре. Современные масс-спектрометры способы выбрать диапазон m/z (отношения молекулярная масса/заряд) и фрагментировать внутри своей вакуумной системы все ионы, находящиеся в этом диапазоне. Если этот диапазон узкий, большинство фрагментов относятся к единственному иону-предшественнику.
— Да, Match Between Runs — это нужная галочка, в принципе, и для DIA. Например, у Демичева [6] в описании к программе DIA-NN указано, что без этой кнопки у вас всё будет хуже, чем с ней.
Вадим Демичев, выпускник МГУ, в настоящее время руководитель группы в Университетской клинике Шарите в Берлине — главный разработчик популярного программного обеспечения (DIA-NN) для анализа масс-спектров пептидов, полученных способом DIA, с использованием машинного обучения, а именно нейронной сети (NN).
— Да. Вот сейчас в работе по болезни Альцгеймера мы проанализировали DDA-данные своим способом через масс-спектры первого уровня. Ну, естественно, рецензентам это не понравилось, поэтому мы всё переделали еще дополнительно классическим способом анализа DDA. Это мы делали в двух вариантах, с Match Between Runs и без. В итоге без этой опции ничего хорошего там и близко не получилось. С Match Between Runs получаются результаты, сопоставимые с нашим методом, они не полностью пересекаются.
— А тебе не имеет смысла делать заранее библиотеку масс-спектров? Как это обычно делается, проанализировать разделенный на фракции образец методом DDA. Для DIA это помогает тем, что с библиотекой сами вычисления гораздо быстрее.
— Мне лично что в библиотеках не нравится, что мы ищем то, что уже когда-то нашли. Да, мы делаем глубокое фракционирование, да, мы делаем длинные градиенты, но это всё же ограничение по информации.
— Ограничение есть, но всё-таки если спектральная библиотека обширная... Например, я сейчас пользуюсь для линии клеток HeLa библиотекой на 12 тысяч белков, которую взял из чужой статьи, и, наверное, в образцах я вряд ли что-то еще зарегистрирую. А тебе не надо такое?
— Мне не нравится этот подход для работы на клинических образцах, потому что если ты делаешь библиотеку из объединенных образцов, усредненную, ты можешь пропустить качественные масс-спектры какого-то биомаркера, который присутствует в образцах от пациентов.
— С этим я соглашусь, хотя это очень гипотетическая ситуация. Потому что если ты хочешь, например, заниматься тем же термопрофилированием , то такая глубокая библиотека вполне адекватна, потому что ты работаешь на конкретных клеточных линиях. И если ты постарался так, что у тебя есть спектральная библиотека на 12 тысяч генных продуктов, как результат интенсивного фракционирования большого количества материала, миллиграммов белка, то это будет хорошим решением.
Термальное профилирование протеома — набор методов, где протеомы подвергают воздействию серии возрастающих температур [7]. При этом не анализируют денатурировавшие белки. Оказалось, что связывание лиганда может стабилизировать белок при высоких температурах. Исследуя термальные профили протеомов в присутствии низкомолекулярного лекарства и в его отсутствие, можно обнаружить неизвестные белки-партнеры, связывающие это соединение. Термальное профилирование протеома в настоящее время широко используется при разработке и перенаправлении лекарственных средств.
— В таком случае, да.
— Потому что белковые биомаркеры, которые существуют по принципу «всё или ничего», это, к сожалению, похоже, утопия. Исключением могут быть орфанные заболевания (где белковые маркеры, по сути, и не нужны); может быть, мутантные белки при некоторых злокачественных опухолях.
— Ну понятно. Мечты, мечты.
— Скажи, а применить-то на чем удалось твой метод? Если статьи уже вышли, значит, ты как-то ты доказал людям, что он работает? Какие у тебя были модели?
— Большая часть статей как раз и была вначале для подтверждения того, что всё работает — для этого использовали такие стандартные образцы с заведомо известным составом. И на них мы показываем, как мы нашим методом видим количественные изменения конкретных продуктов.
— А что это за образцы?
Ну, например, смешивают белки E. coli и стандарт человеческих белков, синтетических, покупных. Там 48 белков. И эти белки смешивают с протеомом кишечной палочки в разных количествах, притом что бактериальные белки остаются в одинаковой концентрации. Полученные смеси вносят в хромато-масс-спектрометр и сравнивают (рис. 2).
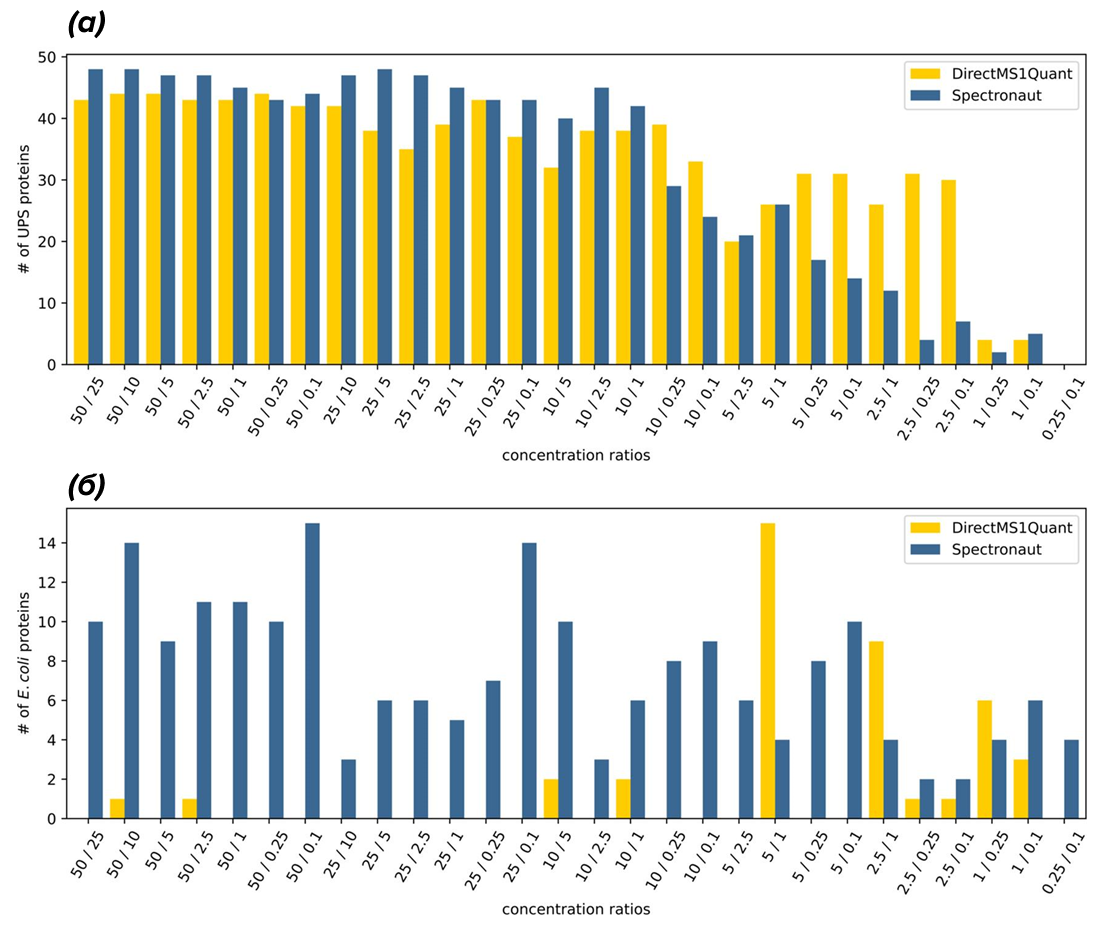
Рисунок 2. Сравнение методов DirectMS1 (программа DirectMS1Quant) и DIA (программа Spectronaut) с использованием смеси белков E. coli и 48 синтетических человеческих белков (UPS), где белки UPS добавляли к смеси в различной концентрации от 0,1 до 50 фмоль. (a) — достоверно обнаруженные дифференциально экспрессированные белки в зависимости от попарно сравниваемых концентраций; (б) — белки, ложно репортируемые как изменившиеся между образцами. Видно, что на использованных данных DIrectMS1Quant дает гораздо меньше ложных результатов.
— То есть, делаются искусственные смеси. А естественные смеси потом какие-то использовали?
— Да, например, в статье про количественный анализ [4] были образцы с воздействием интерферона I типа на клетки глиобластомы. У нас были данные по количественному анализу DDA на длинных хроматографических градиентах (около 2 ч на запуск) из ранее опубликованной работы. К счастью, от этого проекта сохранились замороженные образцы, и для сравнения на них же мы еще сделали эксперименты по нашему методу DirectMS1, где каждый анализ длился всего пять минут. Кроме того, мы дополнили эти данные результатами анализа с изотопными метками TMT , которые до сих пор служат золотым стандартом количественного анализа в протеомике. В общем, результаты по дифференциально продуцированным белкам получились сопоставимыми для всех методов. Где-то поменьше их выявляется, где-то побольше шума, но в целом очень похоже (рис. 3).
Тандемные массовые метки (TMT) — химические метки, которые присоединяются к трипсиновым пептидам протеома в конце пробоподготовки. Набор меток при помощи введения стабильных изотопов сконструирован таким образом, что при фрагментации они дают различные массы, при том, что в целом виде они характеризуются сходной массой. Пометив, например, опыт и контроль разными TMT, а потом смешав их, мы можем сравнить интенсивность стоящих недалеко друг от друга фрагментов в масс-спектрах и достаточно точно определить количественное соотношение одних и тех же пептидов (и белков) в опыте и контроле.
Дифференциально продуцированные (или экспрессированные) белки имеют различные концентрации при сравнении состояний. В англоязычных источниках используется термин «дифференциально экспрессированные», хотя, строго говоря, экспрессируются гены, а белки продуцируются или синтезируются.
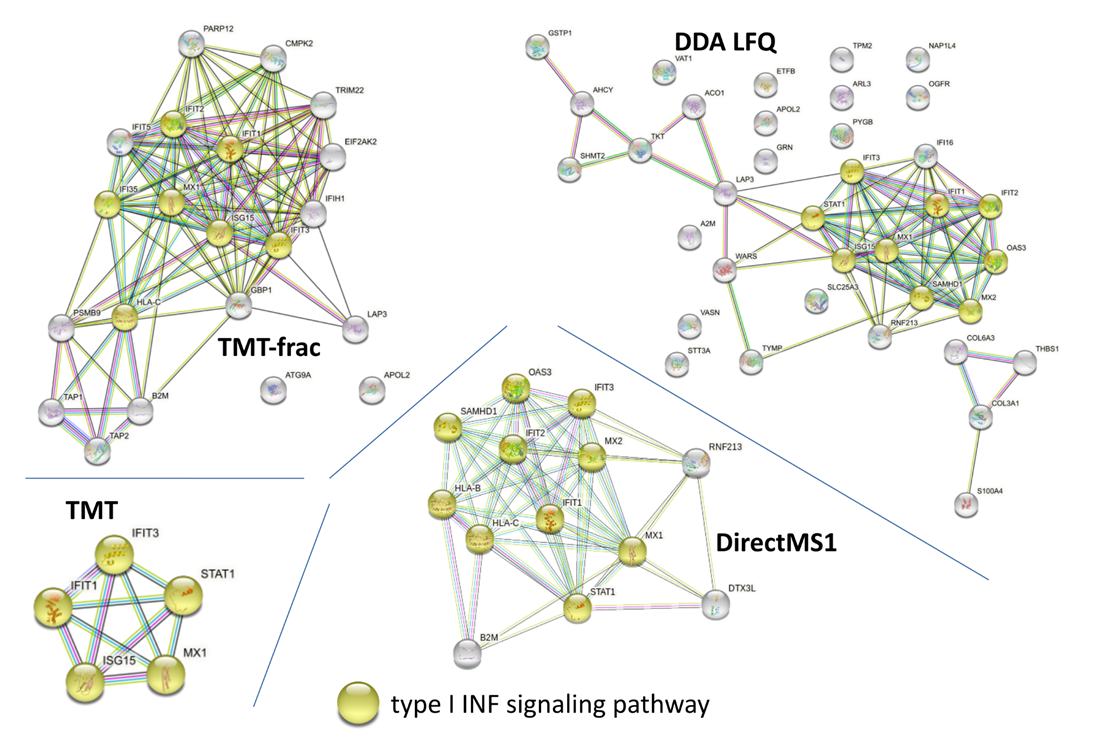
Рисунок 3. Сравнение различных протеомных методов по обнаружению отклика на воздействие интерферона I типа на клетки глиобластомы. Визуализированы сети взаимодействий дифференциально экспрессированных под воздействием интерферона I типа белков, идентифицированным различными методами. Желтым цветом показаны белки каскада интерферона I типа.
— На рынок вышел передовой масс-спектрометр фирмы Thermo — Orbitrap Astral. Там ионная ловушка Orbitrap присоединена в одной вакуумной системе к масс-спектрометру, похожему на времяпролетный (в технические детали сейчас вдаваться не будем), что обеспечило прибавку к чувствительности. Помимо статей, которым мы верим, я сам работал недавно с такими данными, и, действительно, результаты впечатляющие. Какие у твоего метода отношения с Astral?
— Я уже выпустил статью полгода назад в JASMS на эту тему [8]. Мы как раз взяли данные одной из первых работ по Astral, которая вышла в Nature Biotechnology [9]. Astral как раз для нашего метода хорош тем, что он снимает первичные масс-спектры в Orbitrap, а MS/MS — в остальной части прибора. И если мы обычно повторно анализируем чьи-то доступные данные нашим методом, приходится говорить, что вот мы получили такие результаты, но они были бы лучше, если бы эти люди сразу снимали масс-спектры в нашем режиме без фрагментации. А в Astral, получается, вообще никакой разницы. Причем там это реальные образцы были, образцы мозга.
— Как убедить людей пользоваться твоим методом?
— Первые статьи мы выпускали общие, демонстрировали на всяких стандартах, что мы не врём, показываем нужный уровень ложноположительных идентификаций (FDR), и результаты сопоставимы с классическим методом. Но пока этот путь не работает. Остается только продолжать — мы берем задачу, сами делаем своим методом, получаем что-то хорошее и доказываем, что его действительно можно тут использовать. Конечно, если интересуют, как я уже говорил до того, мутации и посттрансляционные модификации, то тут наш подход не годится, нужны классические методы с фрагментацией.
Для чего протеомике искусственный интеллект?
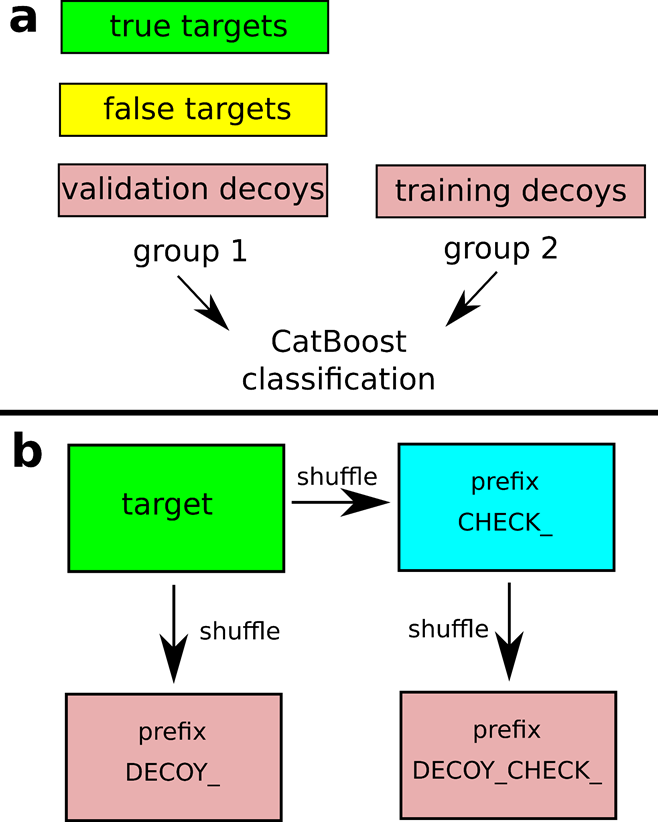
Рисунок 4. Предложенная схема для адаптации традиционного подхода target-decoy и моделей машинного обучения в задачах определения достоверности идентификации пептидов по масс-спектрам фрагментации.
— А теперь давай поговорим об этом AI знаменитом. Он-то где у тебя вообще и зачем?
— Самая большая проблема в протеомике, в целом, даже не только в нашем методе, что нужно отличать что-то ложное от чего-то достоверного. То есть масс-спектров много, они приписываются к реально ожидаемым фрагментам белков, но это всегда гипотеза, где ложное предсказание нужно отделить от правильного. Это, в первую очередь, точка приложения искусственного интеллекта. Повторюсь, у масс-спектрометрии пептидов есть особенность, что смеси очень сложные — много пептидов, много сигналов, и мы пытаемся что-то сопоставить с базой данных белков, теоретически расщепленных на пептиды.
Где-то сопоставление работает очень хорошо, очевидно. Где-то заведомо ясно, что результат ложноположительный, то есть совпадение плохое. Но при этом много и пограничных случаев, в которых хочется разобраться. Необходимо как можно дальше отодвинуть уровень отсечения, чтобы выгрести как можно больше правильных идентификаций. Стандартные статистические методы с этим не справляются, а вот машинное обучение при этом помогает.
— То есть это обучение с учителем, да? Ты ведь заранее алгоритму показываешь достоверные и ложные идентификации?
— Да. Пусть это будет неточно, топорно, но мы берем самые достоверные и самые недостоверные, на них тренируем модель, а потом ей же скармливаем уже всё остальное. Там нужны усложнения, перекрестные валидации, чтобы никакого переобучения не было. Некоторые тонкости касаются, как встроить в работу машинного обучения традиционный подход target-decoy , чтобы всё правильно работало (рис. 4) [10].
Метод контроля ложных идентификаций в панорамной протеомике, основанный на введении в сопоставление масс-спектров и ожидаемых в образце пептидов и белков (target) ложных, заведомо не присутствующих в нем последовательностей-обманок (decoy) [11]. При ранжировании скорингов сопоставления масс-спектров и пептидов от лучшего к худшему появление «обманок» дает сигнал, что нужно провести отсечение.
Другая задача, которую решает машинное обучение, — например, предсказание времен удерживания пептидов на хроматографических колонках. Это ценная информация о пептидах, об их структуре, дополнительная к измеряемой точно молекулярной массе, можно сказать, ортогональная информация.
— А такое предсказание — это не решенная задача? Я слышал об алгоритме DeepLC [12].
— Стандарта предсказания в этой области как такового пока нет, но DeepLC мы любим и используем. Он входит в состав алгоритма, работающего для нашего метода DirectMS1. Достоверность идентификации пептидов мы предсказываем своей моделью, но для хроматографических времен удерживания у нас DeepLC. Мы его немного адаптировали, снабдив более точной калибровкой. Это модель глубокого машинного обучения, она натренирована на разных датасетах, сотнях тысяч пептидов. При этом у нас в группе есть своя модель, но это не машинное обучение. Она сделана на основе статфизики еще задолго до того, как я пришел в лабораторию. Эта модель тоже предсказывает времена удерживания пептидов, но менее эффективно.
В общем, у нас нет цели сделать всё свое полностью; если есть готовые хорошие решения, то лучше их встроить.
— Теперь вопрос от человека, поверхностно понимающего тему. Давно хотел спросить. Почему глубинное, или глубокое обучение? У него есть глубина, что ли, какая-то?
— Модели глубокого обучения используют множество слоев преобразования данных. Каждый слой получает входные данные и передает дальше уже преобразованные данные, и это очень похоже на упрощенную работу человеческого мозга. И вот число используемых слоев характеризует глубину. Важная особенность моделей глубокого обучения — что они могут сами выделять признаки для обучения. При этом их существенный недостаток — данных для обучения нужно очень много . Обычное машинное обучение попроще, сами признаки для обучения должны быть придуманы исследователем. Тут есть плюс — данных для обучения тогда нужно уже меньше, как и вычислительных ресурсов; но есть и минус — от используемых признаков будет зависеть эффективность созданных моделей. Как правило, глубокое машинное обучение применяется для задач по распознаванию речи, изображений или текстов, а вот для данных околотабличного вида берут методы попроще.
Мы рассматривали глубокое обучение в контексте естественных наук в статье «История развития искусственного интеллекта и его пришествия в биологию» [13].
— Понятно. Насколько я понимаю, при использовании более простых методов стараются уменьшить размерность, снизить число этих параметров, который берет в расчет модель?
— Да, скорее так.
— Я еще помню, на заре туманной юности мы использовали алгоритм опорных векторов (Support Vector Machine ) для выбора пиков на масс-спектре плазмы крови, по которым отличали пациентов и здоровых доноров [14], там мы тоже размерность снижали.
Метод опорных векторов — один из инструментов машинного обучения, разработанный в 1995 году при участии советского и американского математика Владимира Вапника.
— Этот метод до сих пор широко используется. Например, в протеомике до сих пор популярна программа Percolator для валидации идентификации по тандемным спектрам (MS/MS). Она на основе SVM.
— Это ведь можно считать машинным обучением тоже, да? То есть машинное обучение — это классификаторы, да?
— Можно так сказать.
— Я тут пытаюсь уловить тонкую грань, когда мы стали называть эти классификаторы такими красивыми словами, и всё это стало какой-то магией. Или теперь это в основном черный ящик, где ты не можешь извлечь из него формулы, по которым он работает?
— Модели бывают интерпретируемые и неинтерпретируемые. В интерпретируемых потом, после обучения и тренировки, мы уже можем достать правила, по которым она что-то предсказывает. Какие из них использовать, зависит от задач. Если мы хотим разделить достоверные и ложные идентификации пептидов, нас не сильно волнует интерпретируемость. Мы просто эту модель берем, используем, и вот она что-то выдает, мы это используем как метод. А если мы изучаем какую-то условно биологическую проблему, например, что мы делали недавно, анализировали протеомы пяти сотен образцов мозгов, связанных с болезнью Альцгеймера. Тренировали модель различать образцы от бессимптомной болезни; болезни, развернутой клинически; и от контрольной группы. Пытались выяснить, по концентрациям каких белков, согласно этой модели машинного обучения, можно распознать три клинические группы. В такой задаче модель как классификатор сама по себе не нужна (задача постановки диагноза по образцам мозга, полученным уже после смерти при вскрытии, не особо актуальна), но важна ее интерпретируемость и извлечение этих релевантных для обучения белков.
— Интерпретация дифференциально продуцированных белков — это вообще сложный момент в протеомике. Тут, конечно, новые инструменты пригодились бы.
Мой следующий вопрос тоже, мягко говоря, общеобразовательный. Ты обучил систему, и, если вдруг у тебя компьютер сгорит, на котором эта обученная система (и ты забыл ее зарезервировать, ха-ха!), то всё, ты ее теряешь. То есть это как индивидуальность, личность, что ли?
— Это зависит от того, о чем мы именно говорим. В нашем DirectMS1, когда происходит разделение достоверных и недостоверных идентификаций, модель обучается «на лету». Мы как раз специально используем не нейронные сети или глубокое обучение, а простой градиентный бустинг (gradient boosting), способ попроще. Нам каждый раз под каждый новый набор данных надо обучаться заново. В одних данных у вас будет один систематический сдвиг по массе из-за настроек масс-спектрометра, и, например, там чуть более длинные пептиды из-за специфики пробоподготовки. Тут работает только обучение на лету, и тут модель, которая использовалась, каждый раз забывается.
— То есть в этом случае обучение — это результат, а не инструмент.
— Да. Но, наоборот, если говорим про предсказание времен удерживания, там есть натренированные модели, и они обучены один раз.
Что дальше?
— Я так понимаю, что DirectMS1 в общих чертах сформирован, остается только его использовать самим и как-то попытаться убедить в его полезности других. А что дальше будешь делать, кроме этого? Какие-то новые идеи есть?
— Я наконец освободился от подачи грантов, переподачи статей и прочего, и вернулся к нескольким идеям. Например, что еще не решено в протеомике — это предсказание интенсивности пиков нефрагментированных пептидов. Даже у Матиаса Манна в презентациях про машинное обучение в масс-спектрометрии пептидов говорится, что его применяют почти на всех стадиях, кроме попыток предсказать эту интенсивность.
Маттиас Манн — один из основоположников масс-спектрометрической протеомики, член совета директоров Института биохимии Макса Планка в Германии. В его лаборатории активно используют AI для различных решений в масс-спектрометрии.
— От чего может зависеть эта интенсивность, от ионизации?
— В том-то и проблема, что на нее влияет не только ионизация, но и концентрация белка, взаимодействия при хроматографии с компонентами элюента и многое другое, в том числе непредсказуемые модификации при пробоподготовке, в источнике ионизации. Поэтому так сложно это предсказать. Пока задача совсем не проработана, и я не знаю, есть ли смысл в ее решении, но об этом я сейчас думаю.
Прототип решения у меня есть, но пока я зашел в тупик на том, что не уверен, что в этом в итоге есть смысл, потому что протeоформы белков тоже вносят вклад в то, как именно пептиды «летят», какая у них должна быть интенсивность. Комбинация концентрации протeоформ белков, естественно, будет меняться от образца к образцу, и это мы не можем учесть.
— На первое время хватит. А ведь вы еще какими-то клиническими образцами занимаетесь, как я знаю? Это тоже полезная вещь.
— Да, анализируем большой массив протеомных данных от пациентов с болезнью Альцгеймера, пытаемся найти что-то интересное, то, что не нашли другие. Идентифицировали каскад ферроптоза, который может играть некую роль в клиническом проявлении заболевания. Там, конечно, нужно как-то подступиться к тому, как это дальше исследовать. Отдельный интерес у меня к эффектам противораковых средств в отношении протеома. Написал на эту тему заявку на грант. В общем, найдем, чем себя занять, направлений работы у нас много.
Литература
- «Омики» — эпоха большой биологии;
- 12 методов в картинках: протеомика;
- 12 методов в картинках: очистка молекул и разделение смесей;
- Mark V. Ivanov, Julia A. Bubis, Vladimir Gorshkov, Irina A. Tarasova, Lev I. Levitsky, et. al.. (2022). DirectMS1Quant: Ultrafast Quantitative Proteomics with MS/MS-Free Mass Spectrometry. Anal. Chem.. 94, 13068-13075;
- Mark V. Ivanov, Leyla A. Garibova, Valeriy I. Postoenko, Lev I. Levitsky, Mikhail V. Gorshkov. (2024). On the excessive use of coefficient of variation as a metric of quantitation quality in proteomics. Proteomics. 24;
- Vadim Demichev, Christoph B. Messner, Spyros I. Vernardis, Kathryn S. Lilley, Markus Ralser. (2020). DIA-NN: neural networks and interference correction enable deep proteome coverage in high throughput. Nat Methods. 17, 41-44;
- Cecile Le Sueur, Henrik M. Hammarén, Sindhuja Sridharan, Mikhail M. Savitski. (2022). Thermal proteome profiling: Insights into protein modifications, associations, and functions. Current Opinion in Chemical Biology. 71, 102225;
- Mark V. Ivanov, Anna S. Kopeykina, Mikhail V. Gorshkov. (2024). Reanalysis of DIA Data Demonstrates the Capabilities of MS/MS-Free Proteomics to Reveal New Biological Insights in Disease-Related Samples. J. Am. Soc. Mass Spectrom.. 35, 1775-1785;
- Ulises H. Guzman, Ana Martinez-Val, Zilu Ye, Eugen Damoc, Tabiwang N. Arrey, et. al.. (2024). Ultra-fast label-free quantification and comprehensive proteome coverage with narrow-window data-independent acquisition. Nat Biotechnol. 42, 1855-1866;
- Mark V. Ivanov, Lev I. Levitsky, Julia A. Bubis, Mikhail V. Gorshkov. (2019). Scavager: A Versatile Postsearch Validation Algorithm for Shotgun Proteomics Based on Gradient Boosting. Proteomics. 19;
- Joshua E Elias, Steven P Gygi. (2007). Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Meth. 4, 207-214;
- Robbin Bouwmeester, Ralf Gabriels, Niels Hulstaert, Lennart Martens, Sven Degroeve. (2021). DeepLC can predict retention times for peptides that carry as-yet unseen modifications. Nat Methods. 18, 1363-1369;
- История развития искусственного интеллекта и его пришествия в биологию;
- Sergei A. Moshkovskii, Maria A. Vlasova, Mikhail A. Pyatnitskiy, Olga V. Tikhonova, Metanat R. Safarova, et. al.. (2007). Acute phase serum amyloid A in ovarian cancer as an important component of proteome diagnostic profiling. Prot. Clin. Appl.. 1, 107-117.






