Виртуальные битвы с бактериями: использование вычислительной биологии для поломки белка RpfB и борьбы с Stenotrophomonas maltophilia
08 апреля 2025
Виртуальные битвы с бактериями: использование вычислительной биологии для поломки белка RpfB и борьбы с Stenotrophomonas maltophilia
- 55
- 0
- 0
На этой картинке представлена сцена кворум-сенсинга между бактериями, где зеленые звездочки — это молекулы кворум-сенсинга.
Рисунок в полном размере.
коллаж автора
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: Если я скажу, что бактерии между собой разговаривают, возможно, вы мне не поверите. Но на самом деле у них есть целый процесс «переговоров», называемый кворум-сенсингом или чувством кворума. Этот процесс позволяет им работать вместе и таким образом еще сильнее атаковать организм. В этом исследовании представлен стратегический план действий по прерыванию переговоров между бактериями Stenotrophomonas maltophilia, используя биоинформатику. Эти бактерии общаются с помощью особой молекулы — диффузионного сигнального фактора (DSF), регулируемого белком RpfB. Вот этот белок мы и попробуем вывести из строя, собрав свою команду молекул, которые могут тормозить его действие.
Конкурс «Био/Мол/Текст»-2024/2025
Эта работа опубликована в номинации «Школьная» конкурса «Био/Мол/Текст»-2024/2025.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Данная работа была выполнена в Лаборатории вычислительной биологии Института Биотехнологии и Биомедицины под руководством Хавьера Дауры. Это мой первый опыт участия в научном исследовании , который я смогла реализовать благодаря программе Joves i Ciència. Хотя это только начало моего пути в науке, давайте я, как в настоящей статье, расскажу вам о проделанной работе, методах и полученных результатах, а также о том, какие выводы можно из этого сделать.
У «Биомолекулы» есть принцип: посвящать популярные материалы только тем темам, по которым уже вышли научные статьи в рецензируемых журналах с импакт-фактором — в норме мы отказываем в публикации «оригинальных» результатов. Но тут случай особый, потому что автор — школьница, сделавшая научную работу в рамках программы международных стажировок, и главная цель этой статьи — не научные результаты как таковые (они требуют подтверждения и рецензии профессионалами), а скорее популяризация научной деятельности как таковой. — Ред.
Кворум-сенсинг : мессенджер для S. maltophilia
Бактерии общаются между собой примерно так же, как мы в мессенджерах. Они тоже обмениваются сообщениями, только в их случае это не стикеры, а сигнальные молекулы. Через процесс кворум-сенсинга они выделяют аутоиндукторы, которые можно сравнить с приглашением на вечеринку. Чем больше бактерий, тем выше концентрация этих молекул, и в определенный момент срабатывает сигнал: пора переходить к действиям [1], [2]! Благодаря такому сговору бактерии координируют сложные действия, такие как формирование биопленок, выработку устойчивости к антибиотикам и усиление своей «атаки». К сожалению, всё это сильно усложняет нашу задачу в борьбе с инфекциями [3–5].
Для погружения в контекст кворум-сенсинга рекомендуем прочесть статью «Бактерии, молчать! Как и зачем вносить помехи в межклеточное общение» [6]. — Ред.
Stenotrophomonas maltophilia (или просто S. maltophilia) — одна из самых хитрых бактерий. Она использует чувство кворума (QS), чтобы организовать свои «злые» планы по захвату организма, включая образование биопленки, подвижность и устойчивость к антибиотикам. S. maltophilia на первый взгляд может показаться безобидным, так как в основном он не наносит вреда здоровым людям. Но в случаях пониженного иммунитета S. maltophilia превращается из безопасного микроба в смертельно опасного воина. Особенно это касается пациентов в больницах, где он существует, и учится становиться устойчивым ко многим препаратам. Всемирная организация здравоохранения считает S. maltophilia одним из самых сложных для лечения больничных патогенов [7]. Более того, S. maltophilia является одной из основных причин инфекции у пациентов с муковисцидозом, что является дополнительной проблемой [8].
У S. maltophilia кворум-сенсинг управляется специальным химическим «сигналом» — жирной кислотой с замысловатым названием цис-11-метил-2-додеценовая кислота, или просто диффузионный сигнальный фактор (DSF). Впервые он был обнаружен у бактерии Xanthomonas campestris [9], [10]. Так как эта молекула играет важную роль в кворум-сенсинге, возможность «заглушить» DSF — перспективная стратегия, которая может ослабить бактерию и сделать её более уязвимой [11–15].
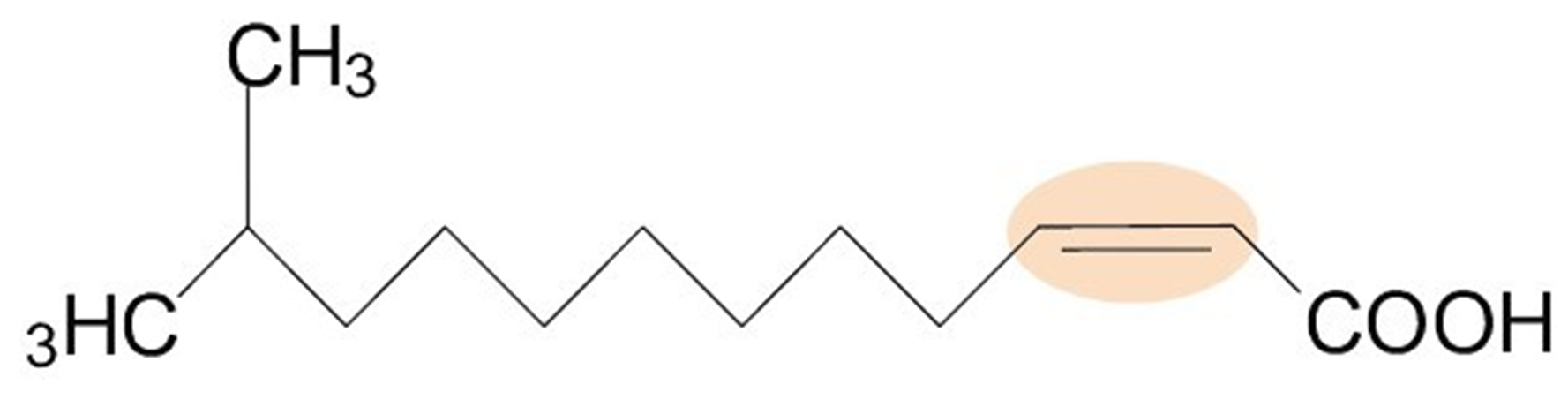
Рисунок 1. Структура DSF. Оранжевым выделена двойная цис-связь.
DSF — продукт работы генного кластера rpf (регуляция факторов патогенности). У S. maltophilia этот кластер имеет два варианта: rpf1 и rpf2. Если в штамме доминирует rpf1, бактерия может активно производить DSF. Кластер rpf разделен на два «операционных центра»: rpfBF и rpfCG. Первый включает гены, отвечающие за производство RpfB — это фермент, играющий ключевую роль в обработке DSF [16], [17]. Именно белок RpfB и является центральной частью этого исследования.
RpfB — это своего рода «фильтр», который предотвращает накопление слишком большого количества DSF в клетке. Как же он это делает? С помощью кофермента А (CoA) он помечает DSF, чтобы тот мог быть уничтожен с помощью β-окисления — процесса, который расщепляет жирные кислоты. Хотя в клетке есть и другие ферменты, способные работать с CoA, уникальная структура DSF (например, двойная цис-связь) требует особого специалиста — RpfB. Без этого фермента вся система кворум-сенсинга может выйти из-под контроля. Поэтому, учитывая, насколько важен DSF для регулирования вирулентности, это исследование сосредоточено на том, чтобы белок RpfB перестал работать [16], [17]. Для этого нужно найти молекулы, которые могли бы ингибировать RpfB, нарушив работу кворум-сенсинга. Как раз тут в дело идет биоинформатика. С помощью баз данных и вычислительных программ можно найти такие молекулы.
Методы, или краткий гайд по поломке RpfB, используя вычислительную биологию
После нескольких проб и ошибок была разработана методология, состоящая из пяти этапов: пять шагов, чтобы «сломать» RpfB. Сначала мы предсказали структуру этого белка с помощью AlphaFold и подготовили базу данных молекул-лигандов, которые могли бы с ним взаимодействовать. Затем мы идентифицировали, где именно на белке располагается сайт связывания АТФ — основная «мишень». После этого мы провели фармакофорный поиск (о нем чуть позже) и завершили работу виртуальным докингом, чтобы увидеть, какие молекулы могут соединиться с этим сайтом и эффективно ингибировать RpfB.
Немного магии: предсказание структуры RpfB
До недавнего времени определение трехмерных структур белков без долгих и затратных экспериментов казалось невозможным — пока не появился AlphaFold в 2021 году [18–21] . Эта программа использует искусственный интеллект и глубокое обучение, чтобы предсказывать структуры белков. Столь революционное открытие даже удостоилось Нобелевской премии по химии в 2024 году [22].
На самом деле, это не совсем так: предсказание 3D-структур имеет почти столь же длительную историю, как и сама структурная биология [23], только раньше для этого использовался почти исключительно принцип гомологии (см.: «Торжество компьютерных методов: предсказание строения белков» [24]), а сейчас накопившиеся «большие данные» и мощные алгоритмы глубокого обучения научились делать такие предсказания даже в случае, если гомологи неизвестны (см.: «История развития искусственного интеллекта и его пришествия в биологию» [21], «Как языковые модели покорили мир белков» [25]). — Ред.
Было решено самостоятельно использовать AlphaFold для прогнозирования структуры RpfB, хотя высоконадежная модель уже была доступна в базе данных AlphaFold. Это решение было принято по двум основным причинам. Во-первых, мы хотели посмотреть, сможет ли AlphaFold обеспечить еще более высокую оценку pLDDT (predicted Local Distance Difference Test), особенно в важных областях, таких как сайт связывания АТФ. Во-вторых, поскольку это был мой первый опыт работы с инструментами вычислительной биологии, он предоставил прекрасную возможность получить практический опыт работы с AlphaFold.
Последовательность аминокислот RpfB можно найти на Uniprot [26]. Белок RpfB с кодом последовательности B2FQ88 состоит из 558 аминокислот.
Итак, первым шагом в этом исследовании стало предсказание трехмерной (3D) структуры белка RpfB с помощью AlphaFold. Для работы AlphaFold нужна мощная техника, поэтому был использован графический процессор Nvidia, который значительно ускорил процесс. Мы загрузили AlphaFold с помощью специального bash-сценария (назовем его Скрипт 1), который помог автоматизировать создание файлов в формате PDB — 3D-моделей белков. Но было важно не только построить структуру, но и оценить ее точность. Для этого программа рассчитала две ключевые метрики:
- pLDDT (тест прогнозируемой локальной разницы расстояний): это показатель уверенности AlphaFold в том, как точно он предсказал каждую часть белка;
- PAE (прогнозируемая ошибка выравнивания): этот тест измеряет, насколько близко находятся различные части белка в пространстве.
Чтобы всё это заработало, мы создали виртуальную среду Python 3.9 и добавили необходимые «инструменты» — библиотеки Jax, matplotlib и numpy c помощью нескольких команд:
pip3.9 install matplotlib numpy
pip3.9 install --upgrade "jax[cpu]==0.4.1"
Затем мы подключили среду Python и выполнили Скрипт 2.
Подготовка базы данных
Чтобы найти идеальные кандидаты для взаимодействия с белком RpfB, нужна база данных лигандов. Для этого я использовала коллекцию Enamine Hit Locator (выпуск от февраля 2022 года) — это словно огромная библиотека, состоящая из 460 160 молекул. Эта библиотека была выбрана среди других более крупных коллекций, таких как ZINC, поскольку эта коллекция предварительно отфильтрована на предмет сходства с лекарствами, что гарантирует, что молекулы будут соответствовать желаемым физико-химическим свойствам. Но не все молекулы в этой библиотеке идеальны: некоторые из них с «опечатками», а другие нуждаются в «редактировании». Поэтому сначала я провела «чистку» базы данных, используя программу Molecular Operating Environment (MOE).
Я использовала специальный скрипт (назовем его Скрипт 3), чтобы автоматизировать процесс очистки. Вот как это выглядело:
- Чистка структур: Очистка данных включает удаление нечитаемых записей, приведение структур к единому виду и устранение ненужных элементов. Например, соли представляют в виде ионных форм, молекулы с илидными связями записывают в стандартной форме, а структуры, содержащие редкие или неорганические элементы, исключаются из базы.
- Фильтрация по тесту Опреа: В отличие от правила Липинского [27], этот тест используется на ранних этапах процесса разработки возможных лекарств- молекул, которые можно оптимизировать для превращения в потенциальные лекарственные препараты [28], [29]. Только молекулы, которые являются возможными лекарствами, прошли этот этап. Остальные были удалены.
- Избавление от таутомеров: Молекулы с более чем 25 таутомерами или состояниями протонирования также исключались.
- Генерация стереоизомеров: Мы добавили варианты молекул с разной пространственной конфигурацией.
- Создание кодов SMILES: Эти коды описывают структуру молекул в текстовом виде, упрощая дальнейшую обработку.
После выполнения всех пяти шагов было обнаружено, что выходные данные распределились по двум форматам: molfile V2000 и molfile V3000. MDL Molfile — это формат файла для хранения информации об атомах, связях и координатах молекулы. Обычно, если число атомов или связей превышает 999, используется расширенный формат molfile V3000. Текущей версией является molfile V2000, хотя в последнее время формат V3000 стал достаточно распространен, однако некоторые приложения как MOE не до конца поддерживают этот формат. Инструменты MOE SD лучше работают с форматом V2000, так как он предотвращает потерю информации. Поэтому я использовала еще один скрипт (Скрипт 4), чтобы отделить молекулы формата V2000. Это помогло сохранить качество базы данных.
более мелкие блоки, используя Скрипт 5 (написанный на Perl). В итоге получилось 128 мини-баз, каждая из которых включала 3000 молекул. Это позволило работать быстрее и без риска ошибок.
Завершающий этап — создание энергетически стабильных форм молекул, называемых конформациями. Мы использовали Скрипт 6, чтобы разделить работу между тремя процессами MOE. Такой подход не только ускорил процесс, но и сделал его более надежным, благодаря параллельному скринингу.
В итоге мы получили чистую, готовую к докингу базу данных, которая стала основой для последующих этапов исследования. Теперь можно было начинать поиск молекул, которые могли бы противостоять белку RpfB!
Поиск сайта связывания АТФ: где прицепится молекула?
Следующим шагом стало определение АТФ-сайта на белке RpfB — главной мишени для наших будущих ингибиторов. Обычно ингибируется активный сайт белка, однако для RpfB, как для и других лигаз жирных кислот, субстратом, который присоединяется к активному сайту, являются длинноцепочечные жирные кислоты, которые обычно не подходят для разработки лекарств. Именно поэтому было решено сосредоточиться на сайте АТФ RpfB. Наша цель — найти молекулу, которая сможет занять место АТФ в этом участке — таким образом она будет препятствовать присоединению настоящего АТФ, нарушая работу фермента.
Так как у RpfB нет документированного сайта связи АТФ, я воспользовалась инструментом NCBI BLAST для белков, который позволяет находить «родственников» белка в огромной базе данных [30]. Это делается, сравнивая последовательность аминокислот RpfB с аминокислотами других белков.
Загрузив последовательность аминокислот RpfB в BLAST, нужно было сфокусироваться на ферментах-лигазах с уже известным сайтом АТФ. Для большей точности я ограничила поиск только структурными белками из базы PDB (Protein Data Bank). Сохранив остальные настройки по умолчанию, мы смогли выявить участок, где RpfB взаимодействует с АТФ. Именно на этот участок и будут направлены лиганды.
Фармакофорный поиск: как найти правильные молекулы?
Теперь, когда у нас есть точная информация о сайте связывания АТФ, я перешла к созданию фармакофорного запроса. Фармакофор — это как шаблон, который описывает ключевые свойства молекулы, позволяющие ей идеально вписаться в нужный участок белка.
- Использование MOE Site Finder: эта программа посчитала потенциальные активные участки на RpfB. Я выделила участок, соответствующий АТФ-сайту, и изолировала его, чтобы сосредоточиться только на ключевой области.
- Выбор признаков: основываясь на аминокислотах, окружающих АТФ-сайт, мы создали запрос с шестью основными признаками:
- один донор водородной связи;
- три акцептора водородной связи;
- два гидрофобных центра.
- При настройке фармакофорных признаков в MOE можно задать средний радиус для фармакофора в Å. Для гидрофобных признаков был задан радиус чуть больше, чем для остальных (1,1 против 1 Å), чтобы молекулы могли лучше взаимодействовать с активным участком.
- Фильтрация молекул: мы обозначили все шесть признаков как обязательные. Это значит, что, если молекула не соответствует хотя бы одному из них, она сразу исключается.
С помощью Скрипта 7, который выполняется на MOE, был запущен поиск в базе данных лигандов, подготовленной ранее. Этот процесс помог отобрать только те молекулы, которые могли бы эффективно взаимодействовать с сайтом связывания АТФ на RpfB. Таким образом, мы получили точную выборку кандидатов для следующего этапа — докинга.
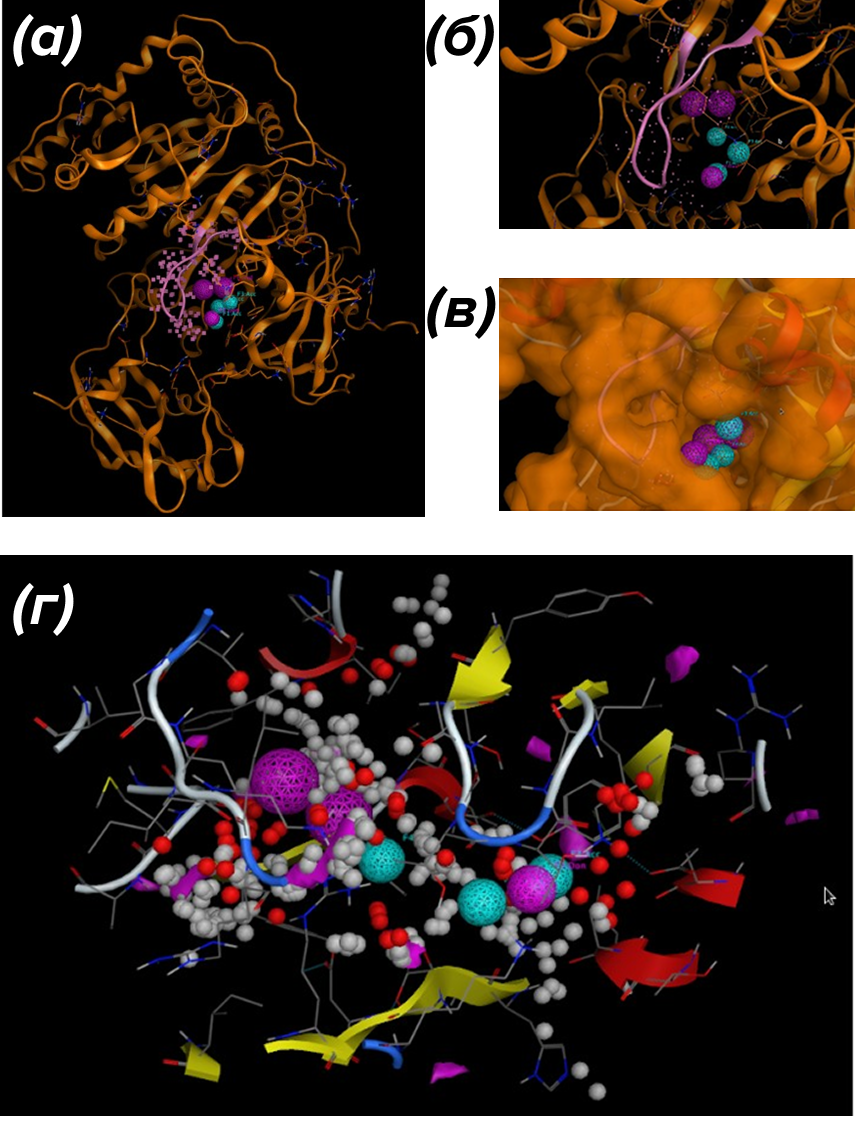
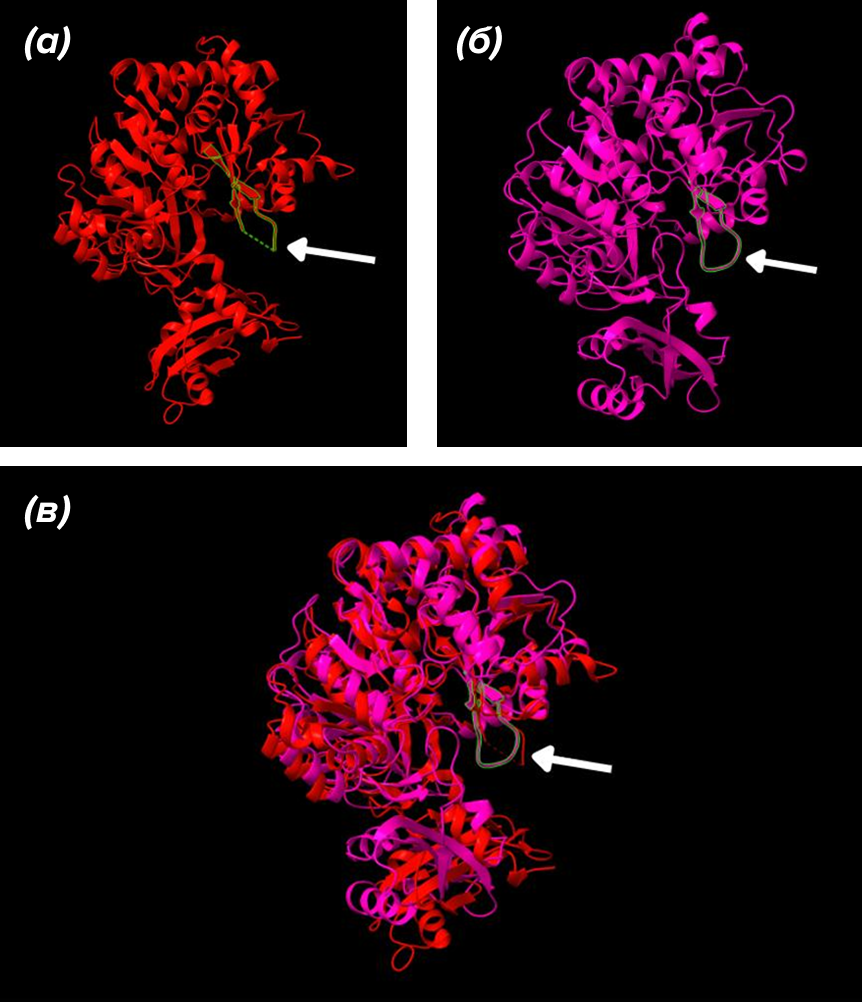
Рисунок 2. Расположение фармакофорных признаков в RpfB. (а) и (б) — фармакофорные признаки, включающие шесть основных признаков: доноры и акцепторы водородной связи, и гидрофобные центры. Гидрофобные признаки можно отличить от доноров, поскольку они имеют больший радиус и находятся дальше от участка связывания АТФ. (в) — молекулярная поверхность RpfB с фармакофорными признаками внутри полости связывания АТФ. (г) — изолированная полость связывания АТФ. Белые и красные точки созданы MOE Site Finder. Оранжевым выделена двойная цис-связь.
рисунок создан с помощью программного обеспечения MOE, лицензированного Universitat Autònoma de Barcelona
Докинг: соединение лиганда и белка
Докинг — это словно игра в молекулярный пазл, где задача программы — точно разместить молекулу в нужном участке белка [31]. Этот метод помогает предсказать, как химические соединения (лиганд) взаимодействуют с биологической мишенью (белком). В разработке лекарств это один из ключевых этапов, который позволяет понять, насколько хорошо молекула подходит для блокировки активности белка. В данном случае мы ищем лиганд, способный заблокировать АТФ в белке RpfB.
Прежде чем приступить к процессу, мы провели несколько важных подготовительных шагов:
- Добавление атомов водорода: с помощью MOE Protonate 3D мы добавили к белку и лигандам водороды — это необходимо для более точного учета таких важных взаимодействий как водородные связи.
- Применение MOE QuickPrep — дополнительная стандартизация и подготовка молекул к расчетам.
Теперь, когда белок готов, мы приступили к самому докингу:
- Выбор базы лигандов: для докинга мы использовали молекулы, которые прошли отбор на этапе фармакофорного поиска. Это были лучшие кандидаты, идеально подходящие к нашему АТФ-связывающему сайту. После запуска фармакофорного поиска мы получили базу данных с 1407 молекулами или 3065 с различными конформациями.
- Определение места для докинга: были использованы фиктивные атомы, созданные с помощью инструмента Site Finder, чтобы определить, куда именно лиганды должны прикрепляться. Обычно Site Finder предлагает несколько возможных участков связывания молекул, но мы заранее нашли участок на RpfB, который взаимодействует с АТФ. Таким образом, из всех возможных участков мы сосредоточились на том, который содержит сайт АТФ. Именно в эту область будет присоединяться молекула, способная ингибировать активность АТФ-сайта. Этот шаг позволил программному обеспечению с высокой точностью определить нужное место для связывания.
- Методы размещения и подсчета: использовались стандартные параметры MOE для подсчёта энергетических взаимодействий между лигандом и белком. Это дало возможность определить, какие молекулы подходят лучше всего.
Докинг позволил увидеть, какие из отобранных лигандов действительно могут связываться с белком RpfB. Это важный этап на пути к созданию потенциальных лекарств, которые смогут подавлять активность бактерий.
Результаты
Предсказание структуры белка RpfB в AlphaFold
Модель, предсказанная AlphaFold ранее, имела достаточно высокий средний pLDDT (predicted Local Distance Difference Test). В данном случае средний показатель pLDDT (тест прогнозируемой локальной разницы расстояний) составил 91,5 из 100. Для понимания: чем ближе значение pLDDT к 100, тем увереннее AlphaFold в точности предсказания.
Хотя эта структура выглядела впечатляюще, мы решили проверить, можно ли улучшить ее точность. Мы запустили собственный расчет (Скрипт 1 и Скрипт 2), чтобы получить пять независимых прогнозов структуры RpfB. Это было сделано, чтобы посмотреть, будет ли в одной из структур более высокий показатель pLDDT, чем в базе AlphaFold DB, хотя это маловероятно. Все эти прогнозы показали высокий уровень достоверности, со средним pLDDT 92,4, что выше исходного значения UniProt. Однако у всех шести вариантов, включая Uniprot, очень похожий pLDDT, и в целом их 3D структуры практически идентичны (рисунок 3в), как и можно было ожидать, хотя это необязательно говорит об их правильности.
Модель номер 3 оказалась лучшей среди пяти (хотя разница между ними незначительная и можно было бы использовать любую модель из пяти), с максимальным средним pLDDT (92,9). Именно она стала основой для дальнейшего исследования (рис. 3). B модели №3 были отмечены некоторые неточности:
- Проблемные зоны: аминокислоты 217–220 и C-конец белка.
- Причина: эти области относятся к неупорядоченным сегментам, которые сложнее предсказать из-за их гибкости и отсутствия четкой структуры.
Как можно увидеть на рисунке 3, RpfB имеет два структурных домена.
Анализ точности с использованием метрики PAE
Помимо метрики pLDDT, AlphaFold предоставляет еще один важный инструмент для оценки структуры белка — Predicted Aligned Error (PAE). Эта метрика помогает понять, насколько точно выровнены могут быть предсказанная и реальная структуры белка. PAE измеряется в ангстремах (Å), где чем меньше значение, тем точнее предсказание. Максимальный предел PAE установлен на уровне 31,75 Å [32].
В целом, PAE для модели RpfB оказалось низким, что указывает на высокую точность пространственного расположения аминокислот (рис. 3г). Однако были обнаружены области с более высокими значениями PAE, что сигнализирует о возможных ошибках:
- Ключевая зона с повышенным PAE: область между двумя доменами белка.
- Причина: скорее всего, это связано с гибкостью линкерной области, соединяющей два домена. Эта подвижность делает предсказание пространственного расположения сложнее.
Высокие значения PAE в этих зонах — нормальное явление для белков, имеющих подвижные области. Это не снижает общую точность модели, но требует более осторожной интерпретации в отношении пространственного распределения доменов.
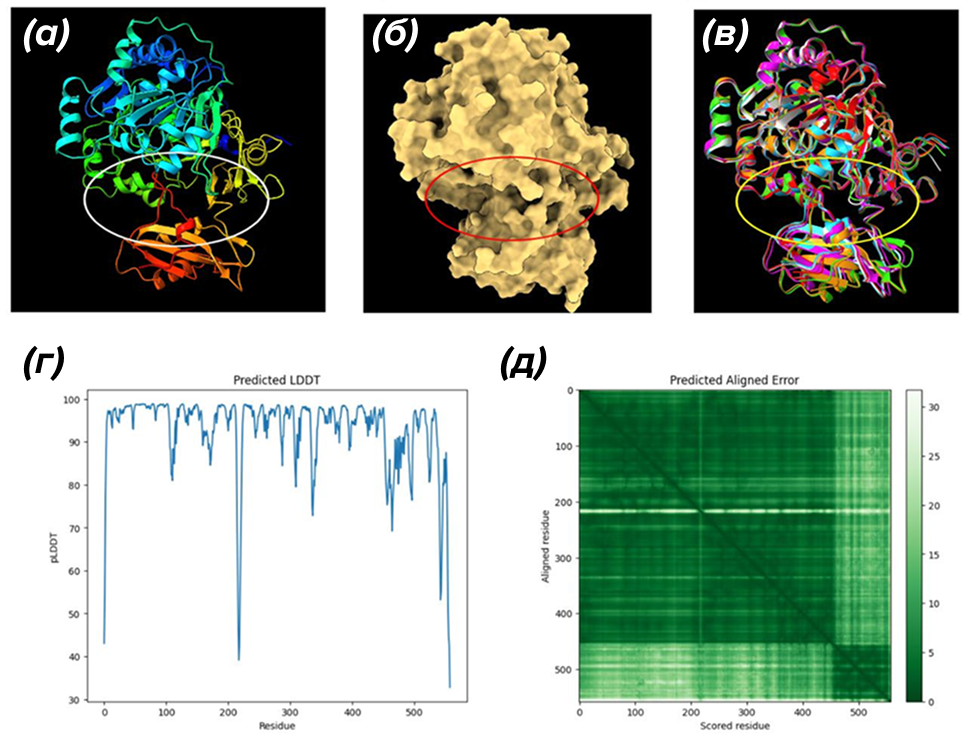
Рисунок 3. Прогноз AlphaFold трехмерной конформации белка RpfB, визуализированный с помощью UCSF ChimeraX [33] в виде (а) ленточной диаграммы или (б) молекулярной поверхности. (в) — шесть совмещенных структур RpfB (одна — загружена из AlpfaFold DB и пять — построены самостоятельно) с помощью ChimeraX Matchmaker. (г) — прогнозируемая LDDT для остатков RpfB. (д) — прогнозируемая ошибка выравнивания для остатков RpfB. На рисунках (а), (б), (в) выделена проблемная зона соединения между двумя доменами белка.
рисунок создан с помощью программного обеспечения MOE, лицензированного Universitat Autònoma de Barcelona
Сайт связывания АТФ
Чтобы найти сайт связывания АТФ в RpfB, мы провели поиск гомологов этого белка с помощью программы BLASTp, потому что у родственных белков он может быть уже обнаружен, что поможет и нам. И вот мы обнаружили, что наиболее близким результатом к последовательности RpfB оказалась жирная ацил-КоА-синтаза FadD13 из Mycobacterium tuberculosis с номером доступа PDB 3R44_A [34].
В этом белке сайт связывания АТФ находится между аминокислотами 163–174. После выравнивания FadD13 и RpfB (рис. 4) мы смогли понять, что в S. maltophilia сайт связывания находится между аминокислотами 214 и 225 — и вот его последовательность: YTGGTTGVAKGA (рис. 5).
Результаты докинга
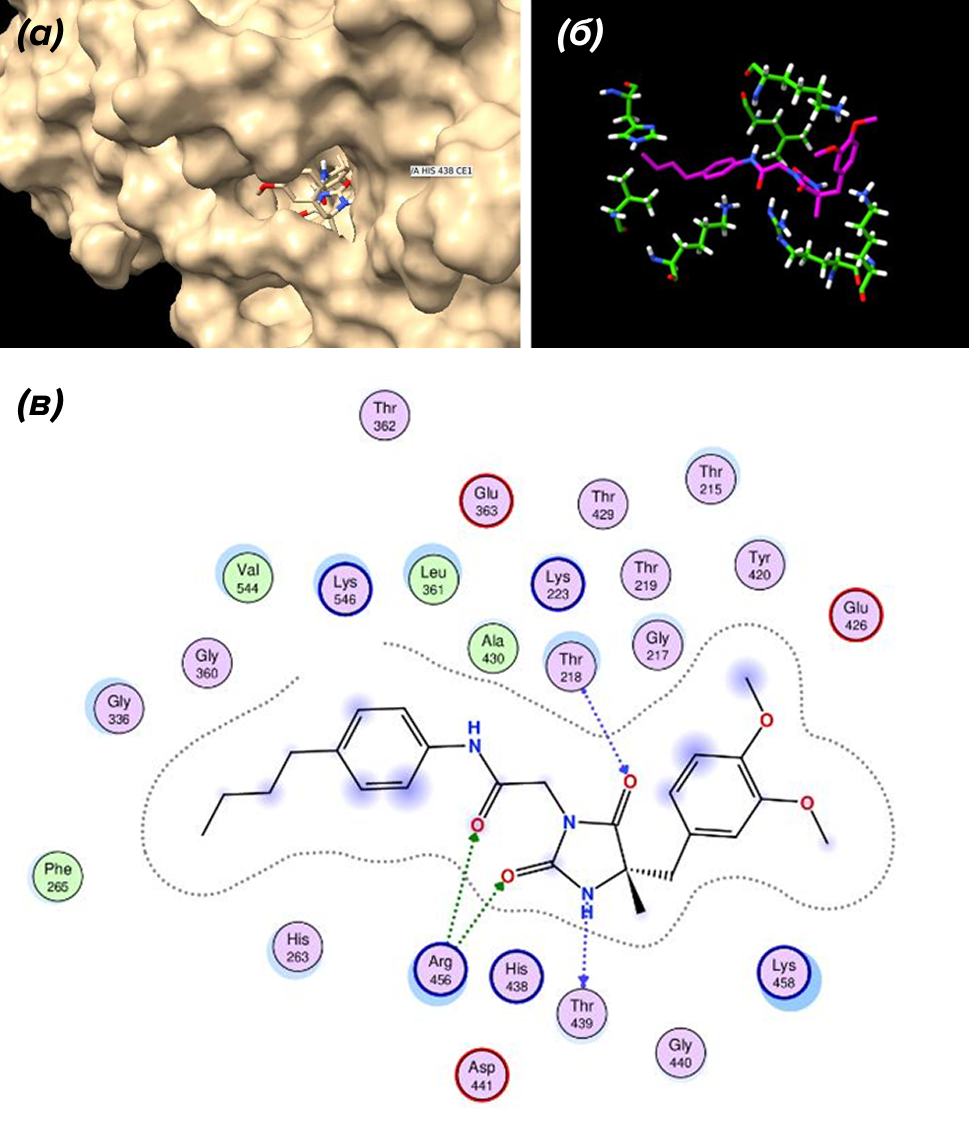
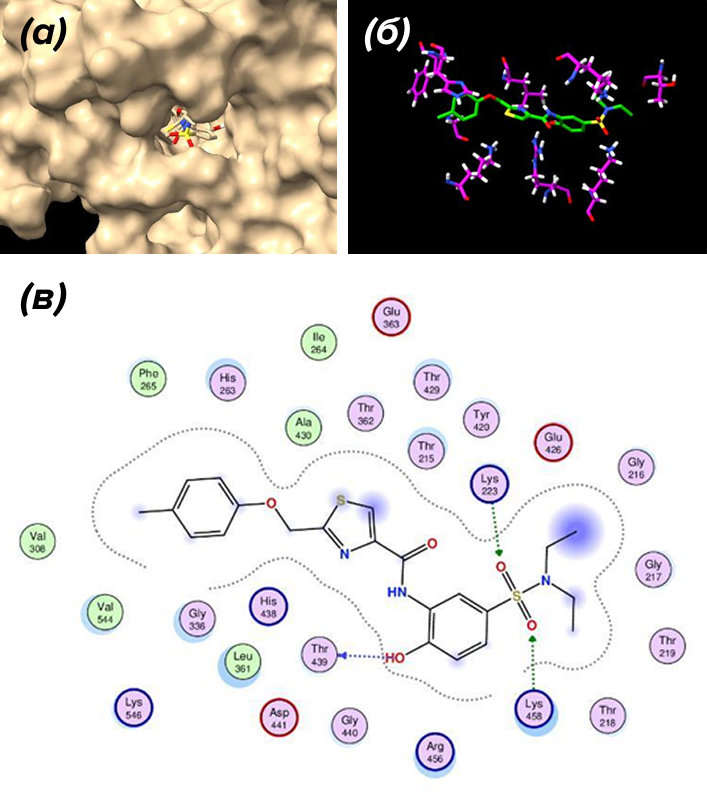
Докинг в MOE выявил 3065 лигандов с отрицательными значениями скоринговой функции, призванной отражать свободную энергию связывания . Из всех этих молекул мы выбрали два лиганда с наиболее отрицательными (то есть — лучшими) скорингами. Оба лиганда продемонстрировали взаимодействие с аминокислотами сайта связывания АТФ, участвуя в водородных связях и гидрофобных взаимодействиях. Примечательно, что ни один из комплексов не продемонстрировал никаких неблагоприятных взаимодействий. Таким образом, эти лиганды вероятно способны ингибировать сайт АТФ в RpfB , прерывая работу белка и впоследствии, коммуникацию между бактериями.
В реальности такого никогда не происходит, потому что у программ докинга в приоритете быстрый расчет скор-функций, а потому делается это физически ненадежными методами, имеющими крайне отдаленное отношение к реальности. Именно поэтому выдаваемые программами докинга значения ΔG не стоит принимать за настоящую свободную энергию Гиббса — это лишь очень грубый способ отсечь случайные результаты от совсем уж невероятных. — Ред.
Но это не точно. — Ред.
Комплекс 1 (рис. 5) имел наилучший балл: −8,7 ккал/моль. Рисунок 5в подчеркивает взаимодействие лиганда с ключевыми остатками сайта связывания АТФ, в частности с треонином 229 и 439, а также аргинином 456. Эта молекула, судя по всему, нейтральна. Водородные связи, показанные на схеме стрелками (зеленые — акцепторы боковых цепей, синие — акцепторы основной цепи), не означают, что молекула заряжена или ионизирована. Такие связи формируются благодаря полярным группам, например карбонильным или гидроксильным, которые могут «принимать» или «отдавать» водородные связи, но сами по себе не несут заряд . На диаграмме видно, что эта молекула не имеет заряженных групп, вроде -COO⁻ или -NH₃⁺, которые обычно придают заряд при нейтральном уровне pH.
Кто хочет разобраться подробнее, милости просим: «Роль слабых взаимодействий в биополимерах» [35]. — Ред.
В случае комплекса 2 (рис. 5Б — он в крутилке после предыдущего) он получил вторую лучшую оценку: −7,6 ккал/моль. Этот комплекс также установил взаимодействия с некоторыми аминокислотами внутри полости связывания АТФ, включая лизин 223, 458 и треонин 439, как визуально показано на рисунке 5Б. Похоже, что эта молекула также не заряжена. Молекула АТФ имеет заряд −3, что поднимает вопрос о том, почему оба лиганда нейтральны. Более того, эти молекулы не являются аналогами АТФ, что было бы наиболее логичным результатом. Это могло произойти, потому что молекулы могут ингибировать связывание АТФ, конкурируя стерически, а не имитируя АТФ напрямую. Это означает, что они занимают сайт, но не повторяют специфические электростатические или водородные связи АТФ. Кроме того, скоринговая функция докинга может отдавать предпочтение гидрофобным или незаряженным взаимодействиям, которые могут иметь сильную энергию связывания, даже если молекула не имитирует АТФ. Однако также возможно, что они не будут работать, в чем можно будет убедиться только с помощью экспериментального анализа.
Обсуждение: как эти находки проливают свет на борьбу с S. maltophilia
С помощью AlphaFold получилось предсказать структуру белка RpfB. Хотя некоторые участки в зоне связывания АТФ вызывали сомнения из-за небольших отклонений, большая часть модели показала высокие результаты pLDDT и PAE. Правда, в зоне соединения двух доменов белка наблюдалась достаточно сильная ошибка, что связано с гибкостью этой области [36]. Такие особенности типичны для белков, и в будущем точность можно улучшить, добавив экспериментальные данные из похожих структур, например белка FadD13 из Mycobacterium tuberculosis [37].
После этапа докинга мы выделили два лиганда, которые могут стать кандидатами для ингибирования RpfB. Эти молекулы демонстрируют устойчивое и энергетически выгодное связывание с целевым участком белка. Однако важно помнить, что предсказания AlphaFold не дают абсолютной уверенности в правильности предсказанной структуры, и некоторые зоны белка в реальности могут отличаться. Tолько проверка экспериментальным методом даст более надежные результаты.
Значение DSF
Кворум-сенсинг (QS) в S. maltophilia — это система, которая управляет ее вирулентностью, позволяя бактерии становиться устойчивее и опаснее. DSF, сигнальная молекула этой системы, играет ключевую роль. Хотя ранее считалось, что белок RpfB помогает синтезировать DSF, позже выяснилось, что он, наоборот, предотвращает его накопление. Это делает RpfB важным регулятором баланса внутри клетки [38], [39].
Интересно, что в экспериментах с другими похожими бактериями, такими как Xanthomonas oryzae pv. oryzae, удаление RpfB вызывало рост уровня DSF, ослабление вирулентности и изменение поведения бактерий. Однако это почти не влияло на их рост, что дает основание полагать: ингибирование RpfB вместе с другими белками кластера может быть еще более эффективным способом борьбы с инфекцией [16]. Несмотря на то, что функция RpfB в S. maltophilia еще не выяснена, похоже, что большинство генов в кластере rpf высококонсервативны среди видов, тесно связанных с семейством Xanthomonadaceae [39]. Поэтому предполагается, что RpfB в S. maltophilia имеет схожую функцию.
Новый подход к лечению бактериальных инфекций
Снижение активности пути DSF — это перспективная мишень для новых методов лечения [40]. В отличие от обычных антибиотиков, данная стратегия направлена на ослабление бактерии, а не на ее уничтожение. Такой подход уменьшает вероятность того, что бактерия со временем выработает устойчивость к антибиотикам.
Проблема в том, что исследований успешного ингибирования DSF у S. maltophilia пока мало. Это подчеркивает необходимость дальнейших исследований в этой области [40].
Ограничения и что делать дальше
Как и любое вычислительное исследование, наша работа имеет свои ограничения. Например, условия в лаборатории и реальные клинические ситуации могут сильно отличаться. Поэтому, несмотря на перспективные результаты, нам нужны дополнительные эксперименты:
- Первый шаг — исследовать взаимодействия рекомбинантного белка и молекул-мишеней для подтверждения связывания.
- Второй шаг — провести исследования in vitro с бактериями, чтобы понять, как наши соединения влияют на их вирулентность и способность формировать биопленки.
- Далее — эксперименты in vivo на модели Galleria mellonella (личинка большой восковой моли). Этот организм часто используют для проверки антимикробных соединений [41].
- Финал — клинические испытания с реальными пациентами, чтобы подтвердить, насколько эффективны наши подходы.
К сожалению, в настоящее время я не могу закончить экспериментальную часть, поскольку моя стажировка в этой лаборатории подошла к концу. Помимо этого, группа, в которой был выполнен этот проект, специализируется исключительно на биовычислительной работе. Однако в рамках третьего года Joves i Ciència я планирую пройти еще одну стажировку в лаборатории Bacterial Molecular Genetics, которая изучает устойчивость к антибиотикам у S. maltophilia, также в Автономном Университете Барселоны. Там я, возможно, смогу продолжить этот проект и завершить эксперименты in vivo.
Итог
В данном исследовании получены два возможных соединения анти-QS, которые могут быть полезны для лечения инфекций S. maltophilia, хотя, как отмечалось ранее, потребуется провести множество дополнительных исследований, прежде чем эти соединения можно будет считать фармацевтическими препаратами. Кроме того, это исследование может быть полезным для других исследований в этой области, давая представление о возможных ингибиторах RpfB.
Заключение
В этом исследовании мы стремимся использовать вычислительные методы для ингибирования RpfB — важного белка в кворум-сенсоре S. maltophilia на основе DSF. В конце нашего исследования мы получили два потенциальных ингибитора, нацеленных на сайт связывания АТФ RpfB. Экспериментальная проверка данного исследования имеет потенциальные последствия в области медицины, предлагая перспективу смягчения инфекций, вызванных S. maltophilia. Несмотря на то, что вычислительные методы не соответствуют точности экспериментальных исследований, мы выбрали эту методологию из-за ее преимуществ с точки зрения времени и экономических ресурсов. Вычислительное исследование не только служит ценной основой для последующих экспериментов, но и сокращает ресурсы, необходимые для их реализации. Существует дефицит исследований, сосредоточенных на белке RpfB в контексте S. maltophilia. С помощью данного исследования мы намерены создать отправную точку для будущих исследований в этой неизученной области.
Благодарность
Прежде всего, я выражаю свою благодарность проекту Joves i Ciència предоставленного организацией Catalunya la Pedrera за предоставление уникальной возможности, которая позволяет ученикам старших классов исследователям заниматься научными исследованиями и личностным ростом. Я также признательна Хавьеру Дауре за исключительную возможность провести мой проект в его лаборатории в Автономном университете Барселоны, а также за предоставление всех скриптов, необходимых для этого исследования. Наконец, я выражаю признательность Оскару Кончилло за помощь в аспектах информатики этого проекта и за предоставление двух скриптов, необходимых для работы с AlphaFold.
Литература
- Xavier Coves, Marc Bravo, Pol Huedo, Òscar Conchillo-Solé, Andromeda-Celeste Gómez, et. al.. (2023). A Stenotrophomonas maltophilia TetR-Like Transcriptional Regulator Involved in Fatty Acid Metabolism Is Controlled by Quorum Sensing Signals. Appl Environ Microbiol. 89;
- Mihael Spacapan, Cristina Bez, Vittorio Venturi. (2023). Quorum sensing going wild. iScience. 26, 108000;
- Wai-Leung Ng, Bonnie L. Bassler. (2009). Bacterial Quorum-Sensing Network Architectures. Annu. Rev. Genet.. 43, 197-222;
- S. T. Rutherford, B. L. Bassler. (2012). Bacterial Quorum Sensing: Its Role in Virulence and Possibilities for Its Control. Cold Spring Harbor Perspectives in Medicine. 2, a012427-a012427;
- Kai Papenfort, Bonnie L. Bassler. (2016). Quorum sensing signal–response systems in Gram-negative bacteria. Nat Rev Microbiol. 14, 576-588;
- Бактерии, молчать! Как и зачем вносить помехи в межклеточное общение;
- Joanna S Brooke. (2014). New strategies againstStenotrophomonas maltophilia: a serious worldwide intrinsically drug-resistant opportunistic pathogen. Expert Review of Anti-infective Therapy. 12, 1-4;
- Reshma Amin, Valerie Waters. (2016). Antibiotic treatment forStenotrophomonas maltophiliain people with cystic fibrosis. Cochrane Database of Systematic Reviews;
- C. E. Barber, J. L. Tang, J. X. Feng, M. Q. Pan, T. J. G. Wilson, et. al.. (1997). A novel regulatory system required for pathogenicity of Xanthomonas campestris is mediated by a small diffusible signal molecule. Molecular Microbiology. 24, 555-566;
- Lisa Crossman, J.Maxwell Dow. (2004). Biofilm formation and dispersal in Xanthomonas campestris. Microbes and Infection. 6, 623-629;
- Pol Huedo, Daniel Yero, Sònia Martínez-Servat, Iratxe Estibariz, Raquel Planell, et. al.. (2014). Two Different rpf Clusters Distributed among a Population of Stenotrophomonas maltophilia Clinical Strains Display Differential Diffusible Signal Factor Production and Virulence Regulation. J Bacteriol. 196, 2431-2442;
- 10.1186/S13104-018-3690-1/FIGURES/3;
- Eliana Alcaraz, Carlos García, Laura Friedman, Beatriz Passerini de Rossi. (2019). The rpf/DSF signalling system of Stenotrophomonas maltophilia positively regulates biofilm formation, production of virulence-associated factors and β-lactamase induction. FEMS Microbiology Letters. 366;
- Daniel Yero, Pol Huedo, Oscar Conchillo-Solé, Sònia Martínez-Servat, Uwe Mamat, et. al.. (2020). Genetic Variants of the DSF Quorum Sensing System in Stenotrophomonas maltophilia Influence Virulence and Resistance Phenotypes Among Genotypically Diverse Clinical Isolates. Front. Microbiol.. 11;
- Tian Ye, Tian Zhou, Qiting Li, Xudan Xu, Xinghui Fan, et. al.. (2019). Cupriavidus sp. HN-2, a Novel Quorum Quenching Bacterial Isolate, is a Potent Biocontrol Agent Against Xanthomonas campestris pv. campestris. Microorganisms. 8, 45;
- Xing-Yu Wang, Lian Zhou, Jun Yang, Guang-Hai Ji, Ya-Wen He. (2016). The RpfB-Dependent Quorum Sensing Signal Turnover System Is Required for Adaptation and Virulence in Rice Bacterial Blight Pathogen Xanthomonas oryzae pv. oryzae. MPMI. 29, 220-230;
- Lian Zhou, Lian-Hui Zhang, Miguel Cámara, Ya-Wen He. (2017). The DSF Family of Quorum Sensing Signals: Diversity, Biosynthesis, and Turnover. Trends in Microbiology. 25, 293-303;
- John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, et. al.. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. 596, 583-589;
- Белковые галлюцинации: как справляется AlphaFold?;
- AlphaFold: нейросеть для предсказания структуры белков от британских ученых;
- История развития искусственного интеллекта и его пришествия в биологию;
- Несуществующие в природе белки́ — за что вручили Нобелевскую премию по химии (2024);
- 12 методов в картинках: структурная биология;
- Торжество компьютерных методов: предсказание строения белков;
- Как языковые модели покорили мир белков;
- The UniProt Consortium, Alex Bateman, Maria-Jesus Martin, Sandra Orchard, Michele Magrane, et. al.. (2023). UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, D523-D531;
- Драг-дизайн: как в современном мире создаются новые лекарства;
- Christopher A Lipinski, Franco Lombardo, Beryl W Dominy, Paul J Feeney. (2001). Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings 1PII of original article: S0169-409X(96)00423-1. The article was originally published in Advanced Drug Delivery Reviews 23 (1997) 3–25. 1. Advanced Drug Delivery Reviews. 46, 3-26;
- Tudor I. Oprea. (2000). . Journal of Computer-Aided Molecular Design. 14, 251-264;
- Stephen F. Altschul, Warren Gish, Webb Miller, Eugene W. Myers, David J. Lipman. (1990). Basic local alignment search tool. Journal of Molecular Biology. 215, 403-410;
- Виртуальные тропы реальных лекарств;
- Mihaly Varadi, Stephen Anyango, Mandar Deshpande, Sreenath Nair, Cindy Natassia, et. al.. (2022). AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models. Nucleic Acids Research. 50, D439-D444;
- Elaine C. Meng, Thomas D. Goddard, Eric F. Pettersen, Greg S. Couch, Zach J. Pearson, et. al.. (2023).
UCSF ChimeraX : Tools for structure building and analysis. Protein Science. 32; - Charlotta S. Andersson, Camilla A.K. Lundgren, Auður Magnúsdóttir, Changrong Ge, Åke Wieslander, et. al.. (2012). The Mycobacterium tuberculosis Very-Long-Chain Fatty Acyl-CoA Synthetase: Structural Basis for Housing Lipid Substrates Longer than the Enzyme. Structure. 20, 1062-1070;
- Роль слабых взаимодействий в биополимерах;
- Alessia David, Suhail Islam, Evgeny Tankhilevich, Michael J.E. Sternberg. (2022). The AlphaFold Database of Protein Structures: A Biologist’s Guide. Journal of Molecular Biology. 434, 167336;
- Thomas C. Terwilliger, Billy K. Poon, Pavel V. Afonine, Christopher J. Schlicksup, Tristan I. Croll, et. al.. (2022). Improved AlphaFold modeling with implicit experimental information. Nat Methods. 19, 1376-1382;
- Hongkai Bi, Yonghong Yu, Huijuan Dong, Haihong Wang, John E. Cronan. (2014).
X anthomonas campestrisRpfB is a fattyAcyl ‐CoA ligase required to counteract the thioesterase activity of theRpfF diffusible signal factor (DSF ) synthase. Molecular Microbiology. 93, 262-275; - Lian Zhou, Xing‐Yu Wang, Shuang Sun, Li‐Chao Yang, Bo‐Le Jiang, Ya‐Wen He. (2015). Identification and characterization of naturally occurring
DSF ‐family quorum sensing signal turnover system in the phytopathogenX anthomonas. Environmental Microbiology. 17, 4646-4658; - Pol Huedo, Xavier Coves, Xavier Daura, Isidre Gibert, Daniel Yero. (2018). Quorum Sensing Signaling and Quenching in the Multidrug-Resistant Pathogen Stenotrophomonas maltophilia. Front. Cell. Infect. Microbiol.. 8;
- Athina Andrea, Karen Angeliki Krogfelt, Håvard Jenssen. (2019). Methods and Challenges of Using the Greater Wax Moth (Galleria mellonella) as a Model Organism in Antimicrobial Compound Discovery. Microorganisms. 7, 85.