
Найдены системы CRISPR, использующие обратную транскрипцию
09 марта 2016
Найдены системы CRISPR, использующие обратную транскрипцию
- 1277
- 0
- 6
Система CRISPR/Cas — «ножницы» для генома
-
Автор
-
Редакторы
Темы
Некоторые бактерии способны сохранять фрагменты геномов инфекционных агентов, используя в качестве исходного материала не только ДНК, но и РНК. Такие бактерии могут развивать иммунитет к вирусам с РНК-геномами. Кроме того, благодаря CRISPR-системе, использующей РНК, бактериальный иммунитет учится реагировать на наиболее активные гены патогенов.
В 1987 году японские ученые обнаружили в геномах бактерий участки с регулярной структурой — короткие одинаковые последовательности чередовались с уникальными фрагментами, которые у разных бактерий не имели ничего общего. Такие участки назвали CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). Спустя 10 лет удалось доказать, что эти участки отвечают за адаптивный иммунитет бактерий, а в уникальных последовательностях — спейсерах — хранится информация о геномах вирусов, против которых бактерия умеет бороться. РНК, которая синтезируется с таких участков, в комплексе с белками Cas уничтожает соответствующую ДНК вирусов, если они атакуют клетку. А самое замечательное, что бактерия может пополнять свою библиотеку вирусной ДНК, когда встречается с незнакомыми патогенами [1]. Именно поэтому иммунитет CRISPR адаптивный — он совершенствуется и учится противостоять новым типам инфекции. CRISPR — это интересный пример эволюции по Ламарку: события жизни организма непосредственно влияют на его ДНК, изменяя ее так, что организм становится более приспособленным. С другой стороны, CRISPR интересны для прикладной науки — оказалось, что, снабдив белки Cas произвольной РНК, можно заставить их расщепить ДНК клеток в соответствующем месте . С помощью таких систем можно просто и точно редактировать геномы самых разных клеток (оказалось, что бактериальные CRISPR хорошо работают и в клетках эукариот). Возможности CRISPR настолько замечательны, что редкий номер авторитетного журнала сегодня обходится без статьи, разъясняющей новые подробности работы этих систем или рассказывающей о новом их применении.
Ранее «биомолекула» неоднократно писала о достижениях в области использования CRISPR/Cas-систем для редактирования генома: «CRISPR-системы: иммунизация прокариот», «Мутагенная цепная реакция: редактирование геномов на грани фантастики» и «Вылечить миодистрофию Дюшенна: конкуренция групп, единство методик» [2–4]. — Ред.
Хотя никто уже не сомневается, что CRISPR способны на многое, 26 февраля 2016 года в Science вышла статья, рассказывающая о типе этих систем с новыми, еще более невероятными возможностями [5]. Оказалось, что некоторые из CRISPR-систем умеют записывать в память информацию не только из ДНК, но и из РНК патогенов.
На мысль, что такая возможность у бактерий должна быть, ученых навели некоторые сделанные ранее наблюдения. Во-первых, было известно, что CRISPR помогают бактериям бороться и против вирусов с РНК-геномом, в жизненном цикле которых вообще не используется ДНК. Значит, должен быть способ переписывать в «память» иммунной системы информацию, записанную в молекулах РНК, а не только в ДНК. Кроме того, у некоторых бактерий среди CRISPR-ассоциированных генов, которые располагаются неподалеку от кассет с иммунной памятью, обнаруживались участки, кодирующие домены обратной транскриптазы. Иногда обратная транскриптаза была закодирована в виде отдельного белка, а иногда в виде химеры с белком Cas1, который обычно вставляет ДНК вируса в кассеты CRISPR. Поэтому напрашивалась мысль, что бактерии могут с помощью обратной транскриптазы переписывать информацию из РНК вируса в форме ДНК и сохранять ее в таком виде.
Чтобы проверить свои догадки, ученые выбрали CRISPR систему легко культивируемых бактерий Martelella mediterranea. В ее составе обнаружился химерный белок с функциями Cas1 и обратной транскриптазы. Чтобы за работой белка было удобнее наблюдать, исследователи получили трансгенные штаммы бактерий с дополнительной копией гена химерного белка, поставленной под сильный промотор. При этом белок с функциями Cas1 и обратной транскриптазы нарабатывался клетками в огромных количествах. В таких условиях в кассеты CRISPR начинали встраиваться новые фрагменты, совпадающие с собственным генетическим материалом бактерии (рис. 1). При этом чаще всего в кассеты встраивались фрагменты тех участков ДНК, что наиболее активно транскрибировались. Эти данные косвенно подтверждали участие РНК в сохранении информации в память CRISPR, но не могли считаться прямым доказательством роли РНК в образовании новых спейсеров. Например, система CRISPR могла предпочтительно использовать ДНК активно транскрибируемых генов, потому что такая ДНК очищается от белков, и компонентам CRISPR могло быть удобнее к ней подойти.
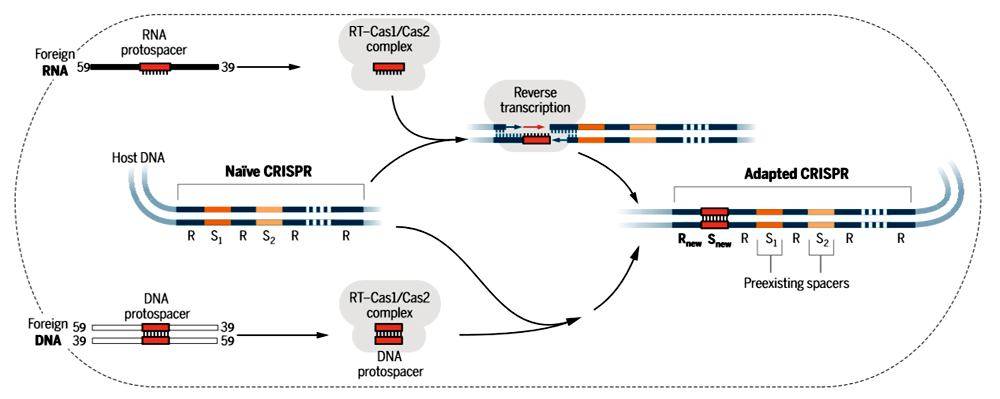
Рисунок 1. Схема встраивания новых спейсеров в CRISPR-кассеты ферментом, сочетающим функции белка Cas1 и обратной транскриптазы. В качестве исходного материала фермент может использовать как молекулы РНК (верхняя часть рисунка), так и молекулы ДНК (нижняя часть). В первом случае обратная транскриптаза достраивает к РНК, вставленной в кассету, комплементарную цепочку ДНК.
Чтобы прояснить вопрос о роли РНК в формировании новых вставок, ученые синтезировали специальную последовательность ДНК, кодирующую самосплайсирующуюся РНК. Такая РНК способна вырезать из самой себя определенный фрагмент. Поэтому вариант молекулы, записанный в ДНК, отличался от РНК-варианта, и можно было различить в CRISPR-кассетах информацию, записанную на базе информации ДНК или соответствующей РНК. Синтетическую ДНК скормили клеткам в больших количествах, чтобы она стала привлекательной мишенью для формирования новых вставок в кассеты CRISPR. Проанализировав последовательности кассет, ученые нашли в них спейсеры, которые могли образоваться только если бактерия записывала в память иммунной системы информацию из РНК-молекул.
Интересно, что при использовании гена со сломанным доменом обратной транскриптазы бактерии не прекращали вставлять в CRISPR-кассеты новые последовательности, но при этом пропадала их склонность вставлять туда фрагменты, соответствующие наиболее активно транскрибируемым генам. Это заставляло предположить, что химерный Cas1 может переписывать в память иммунной системы как информацию, записанную в РНК, так и информацию, записанную в ДНК. Для бактерий это было бы выгодно, потому что позволило бы атаковать все типы нуклеиновых кислот вируса сразу. Чтобы окончательно разобраться в механизме работы белка, ученые поставили чистые эксперименты в пробирке. При этом они использовали определенные типы матриц — только ДНК или только РНК. Эксперименты подтвердили, что оба типа молекул подходят как источник информации для записи в память бактериальной иммунной системы. Особенно интересным оказался механизм записи в кассеты информации из РНК — оказалось, что в ДНК кассет лигируется сама РНК, причем для лигирования необходим рабочий домен обратной транскриптазы. РНК может ориентироваться в любом направлении, и, по-видимому, не существует никаких механизмов, которые помогали бы располагать ее в правильной ориентации. Затем, если добавить в пробирку ДНК-нуклеотиды, на базе РНК достраивается комплементарная цепочка ДНК. Вероятно, в клетке РНК в составе нового фрагмента CRISPR уничтожается РНКазой H, распознающей гибриды РНК и ДНК, и затем заменяется второй цепочкой ДНК, так что в составе кассет не остается РНК-компонента.
Новый тип систем CRISPR можно считать первым обнаруженным примером адаптивного использования обратной транскрипции клеточными формами жизни. Интересно будет поставить эксперименты, которые позволят количественно оценить влияние такого типа иммунитета на выживаемость бактерий, противостоящих инфекции. Хотя система может помочь бактерии нацелиться именно на те гены, что работают у патогена активнее всего, у нее есть и недостаток — уже упомянутое отсутствие направленного ориентирования при встраивании фрагментов РНК в кассеты CRISPR. При этом получается, что около половины спейсеров, полученных на базе РНК, будут расположены задом наперед и будут направлять иммунную систему бактерий искать несуществующие мишени.
Как бы то ни было, новые данные дополнили наши знания о механизмах работы CRISPR-систем. Интересны и потенциальные практические приложения открытия: теперь у биоинженеров появится способ редактировать лишь те генетические варианты, что активно работают в определенной клетке.
Литература
- Sontheimer E.J. and Marraffini L.A. (2016). CRISPR goes retro. Science. 351, 920–921;
- CRISPR-системы: иммунизация прокариот;
- Мутагенная цепная реакция: редактирование геномов на грани фантастики;
- Вылечить миодистрофию Дюшенна: конкуренция групп, единство методик;
- Silas S., Mohr G., Sidote D.J., Markham L.M., Sanchez-Amat A., Bhaya D. et al. (2016). Direct CRISPR spacer acquisition from RNA by a natural reverse transcriptase-Cas1 fusion protein. Science. 351, aad4234.






