Залог успеха — большие данные в умелых руках
26 ноября 2021
Залог успеха — большие данные в умелых руках
- 2350
- 0
- 4
Сможем ли мы в ближайшем будущем совсем избавиться от использования животных в доКИ? Исследователи во всем мире разрабатывают всё более изощренные методы разработки и тестирования лекарств, используя омискные данные. Однако до переноса всего процесса в компьютер все еще далеко. Рисунок в полном размере.
иллюстрация Карины Илиопуло
-
Автор
-
Редакторы
-
Рецензент
-
Иллюстратор
Большие данные и омиксные технологии — модные термины, которыми биотех-стартапы пытаются заманить инвесторов на презентации своей идеи по разработке лекарств. Если же она еще и подкреплена внедрением алгоритмов машинного обучения и другими методами искусственного интеллекта, успех почти гарантирован. Или нет? В этой статье спецпроекта о доклинических исследованиях разберемся, что такое омиксные данные и какое применение они нашли в доклинических исследованиях.
Будущее доклинических исследований
Ни для кого не секрет, что биологические исследования и разработка лекарств — сложный и ответственный процесс. При этом в ходе отбора лучших молекул-кандидатов ученые должны заботиться не только о действенности, но и о безопасности новых препаратов. Так до того, как созданное лекарство получит человек, его изучают в других моделях: от компьютерных программ и клеточных культур до разных видов животных.
Спецпроект о доклинических исследованиях (доКИ) стал своеобразным приквелом к циклу статей о клинических испытаниях (КИ): что нужно сделать и изучить перед тем, как проводить клинические исследования с участием людей? Можно ли обойтись без экспериментов на животных? Почему правильные доКИ не менее значимы для разработки лекарства, чем КИ? Мы постараемся в доступной форме осветить сложные вопросы планирования и регулирования доклинических исследований; принципы этичности работы с лабораторными животными; узнаем, когда и чем можно их заменить; и заглянем в ближайшее будущее биомедицинской отрасли.
Партнер спецпроекта — российские аффилированные лица компании «Филип Моррис Интернэшнл», ФМИ (PMI Affiliates in Russia), которая активно ведет исследования в области биомедицины, системной биологии и биотехнологий.
В предыдущих статьях спецпроекта мы рассказали о самом принципе доклинических исследований (доКИ) [1] и об альтернативах использованию в них животных — культур клеток и гибридных микродевайсов, изображающих органы [2], [3]. Здесь мы рассмотрим, как омиксные данные позволяют еще больше отдалиться от использования живых систем, а также окунемся в более ранние фундаментальные стадии доКИ, где важную роль играют исследования in silico.
Омиксные данные — новый подход в драг-дизайне
Для начала разберем понятие «омиксные данные». Они относятся к так называемым большим данным (Big Data) — термину, знакомому многим, но при этом достаточно расплывчатому. Большие данные получили название за счет своих объемов и разнообразия, а описывают их часто тремя ключевыми V: volume (объем), velocity (скорость прироста данных и необходимость в быстрой обработке) и variety (разнообразие). Иногда к этим характеристикам добавляют еще и veracity (достоверность, качество данных), и value (ценность), что составляет 5 Vs, а порой и больше. Вне зависимости от того, сколько V мы выберем для описания больших данных, понятно, что этот термин охватывает разнообразные данные, которые производятся в огромных объемах и требуют специфических методов обработки. Ввиду своих особенностей, большие данные породили целый полк ученых, занимающихся их анализом, — Data Scientists. Про большие данные в бионауке «Биомолекула» еще напишет, а пока вернемся к их разновидности: омиксным данным, ведь именно они обещают вывести наше понимание патологических процессов и разработку лекарств на новый уровень.
Омиксные данные — большие массивы данных, собранные на разных уровнях биологических процессов. В целом «-омики» (от англ. omics) — это новые биологические дисциплины, объединенные своим подходом к методологии изучения биообъектов: изучить не один или несколько генов, а все гены сразу (геномика), или все белки (протеомика), транскрипты (транскриптомика), метаболиты (метаболомика) и так далее. Мы уже разбирали философию такого подхода в статье «“Омики” — эпоха большой биологии» [4]. К ней и адресуем читателя, который еще не совсем понял, что к чему.
Так, омиксные данные — это совокупность всех молекул определенного уровня, отражающая состояние организма или его части. Омиксные технологии используют высокопроизводительные (high-throughput) методы, что ведет за собой производство больших массивов данных. Для их анализа необходимы определенные компьютерные методы, что породило создание новой дисциплины на стыке биологии, статистики и компьютерной науки — биоинформатики [5]. Как правило, эти специалисты — биологи с прокаченными компьютерными навыками, которые не боятся командной строки UNIX/Linux и вовсю используют язык программирования R. Однако и этого уже становится недостаточно. Способность применения методов машинного обучения для решения биологических задач — требование-тренд, обещающий выжать все информационные соки из омиксных данных (подробнее — в следующей части статьи).
На данный момент основная сфера применения омиксных данных в контексте драг-дизайна — ранние фазы доКИ. Здесь выделяются два направления. Первое — оценка характеристик терапевтических молекул-кандидатов с помощью данных, получаемых из клеток. В этом случае клетки становятся источниками омиксной информации, которую мы используем для оценки характеристик потенциальных лекарств-кандидатов. Второе направление — поисковые доКИ. Здесь речь идет о получении «слепка» состояния организма при заболевании на определенном уровне (или нескольких сразу) и попытках воздействия на него лекарственными средствами. Классический пример применения омиксных данных одного уровня — сравнение транскриптома ткани больных людей и здоровых. Мы сравниваем экспрессию генов тех и других и делаем выводы о молекулярных путях, регуляция которых нарушена при заболевании.
Далее выбираются молекулы-мишени (например, белки, синтез которых значительно повышен при заболевании) [6] и подбираются молекулы-кандидаты, способные воздействовать на них. В последнее время, однако, все чаще исследователи прибегают к интеграции омиксных данных разных уровней — анализу мультиомиксных (multi-omics, или cross-omics) данных (рис. 1). Этому есть простое объяснение: любой процесс в клетке (в том числе патологический) охватывает различные уровни ее молекулярной организации, тем самым находя отражение во множестве типов омиксных данных. Поэтому, собрав и интегрировав данные сразу о последовательностях ДНК, РНК, пептидах, белках, метаболитах и т.д., можно воссоздать более полную картину заболевания и повысить вероятность успеха подбора терапевтических молекул.
Разберем подробнее оба направления применения омиксных данных в доКИ.
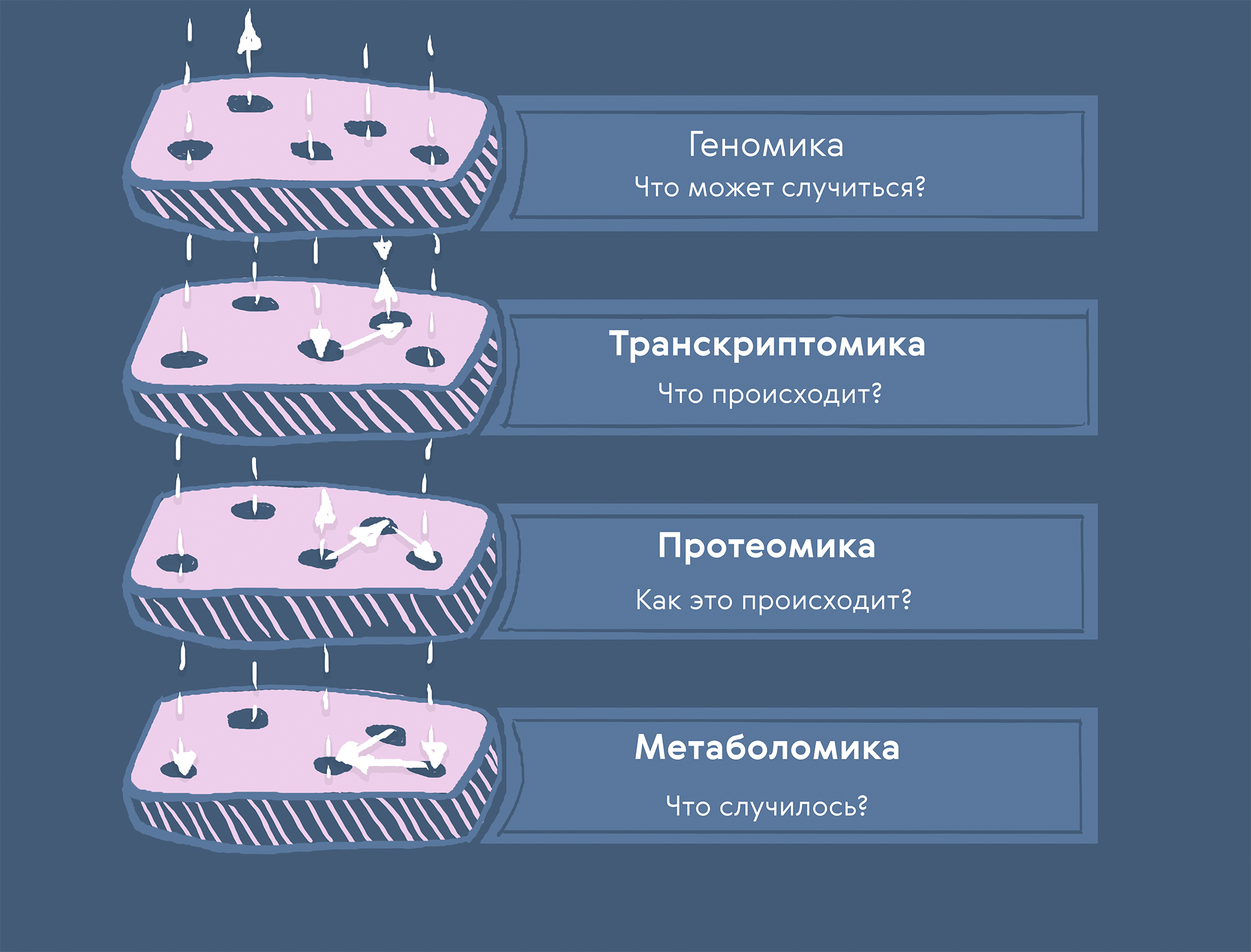
Рисунок 1. Цели мультиомиксных данных по уровням. Геномные данные отвечают на вопрос «что может случиться?»; транскриптомные — «что происходит?»; протеомные — «как это происходит?»; метаболомные — «что случилось?».
иллюстрация Карины Илиопуло по изображению Astra Zeneca
Клетки как платформы для анализа омиксных данных
Классическое применение клеточных моделей в доКИ, описанное в статье спецпроекта об исследованиях in vitro [2], сводится к тестированию отобранной молекулы-кандидата на клеточной линии, содержащей молекулярную мишень этой молекулы. Однако в последние годы были разработаны методики использования клеток в качестве платформ для оценки влияния молекул-кандидатов на полный протеом или транскриптом клеток. То есть в данном случае нет заранее сформированного представления о мишенях — оценивается, как молекула-кандидат влияет на все молекулы одного или нескольких уровней клетки сразу. Таким образом можно провести беспристрастный анализ: выявить как возможные мишени лекарства, так и побочные (off-target) взаимодействия. Это особенно актуально для некоторых терапевтических подходов (или молекул), у которых сразу много мишеней. К таким, в частности, относятся ингибиторы протеинкиназ. Есть даже специальный термин для совокупности всех протеинкиназ организма — кинóм. Используя киномные данные, возможно оценить специфичность уже одобренных ингибиторов протеинкиназ [7] или же подтвердить, что разрабатываемая молекула-ингибитор воздействует именно на мишень, заявленную в качестве основной.
Одним из ключевых примеров здесь выступают омиксные данные на уровне белков — протеома. Протеомное профилирование — многообещающее направление, меняющее представление о скрининге лекарственных веществ. Оно позволяет отслеживать непосредственное взаимодействие между лекарством и всем множеством клеточных белков, что обычно невозможно. Вместо этого вывод о связывании лекарства с мишенью делается на основе того, что можно измерить — клеточный ответ, в котором участвует мишень. Это может привести к провалу даже на уровне КИ, когда выяснится, что лекарство не взаимодействует с предполагаемой мишенью в клетке (а вероятно, с другой молекулой-участницей этого же сигнального каскада).
Чтобы исправить ситуацию, разработали методику температурного профилирования протеома (thermal proteome profiling, TPP), основанную на изменении термостабильности белков при взаимодействии с другими молекулами: если структура белка изменена (скажем, из-за связывания с лигандом), это повлияет на его стабильность при разных температурах. Клетки нагревают в контрольных условиях (без лекарства) и в экспериментальных (когда лекарство добавляют). Далее образцы охлаждают, белки растворимой фракции отделяют с помощью центрифуги и составляют кривую плавления каждого белка. Различие этой характеристики в условиях контроля и при наличии лекарства говорит об измененной стабильности белка, а значит, о его взаимодействии с лекарством. Этот метод получил название пробы клеточного термального сдвига (cellular thermal assay, CESTA) [8].
TPP же совмещает CESTA с масс-спектрометрией (MS) [9] и позволяет оценить изменения в стабильности сразу всех растворимых белков клеток под воздействием лекарства (рис. 2) [10]. Так возможно не только выявить мишени препарата, но и определить нежелательные off-target-взаимодействия между лекарством и белками, которые ответственны за побочные эффекты. Таким образом удалось определить более 50 мишеней ингибитора протеинкиназы стауроспорина, а также найти объяснение светочувствительности, вызываемой противоопухолевыми препаратами вемурафенибом и алектинибом [11].
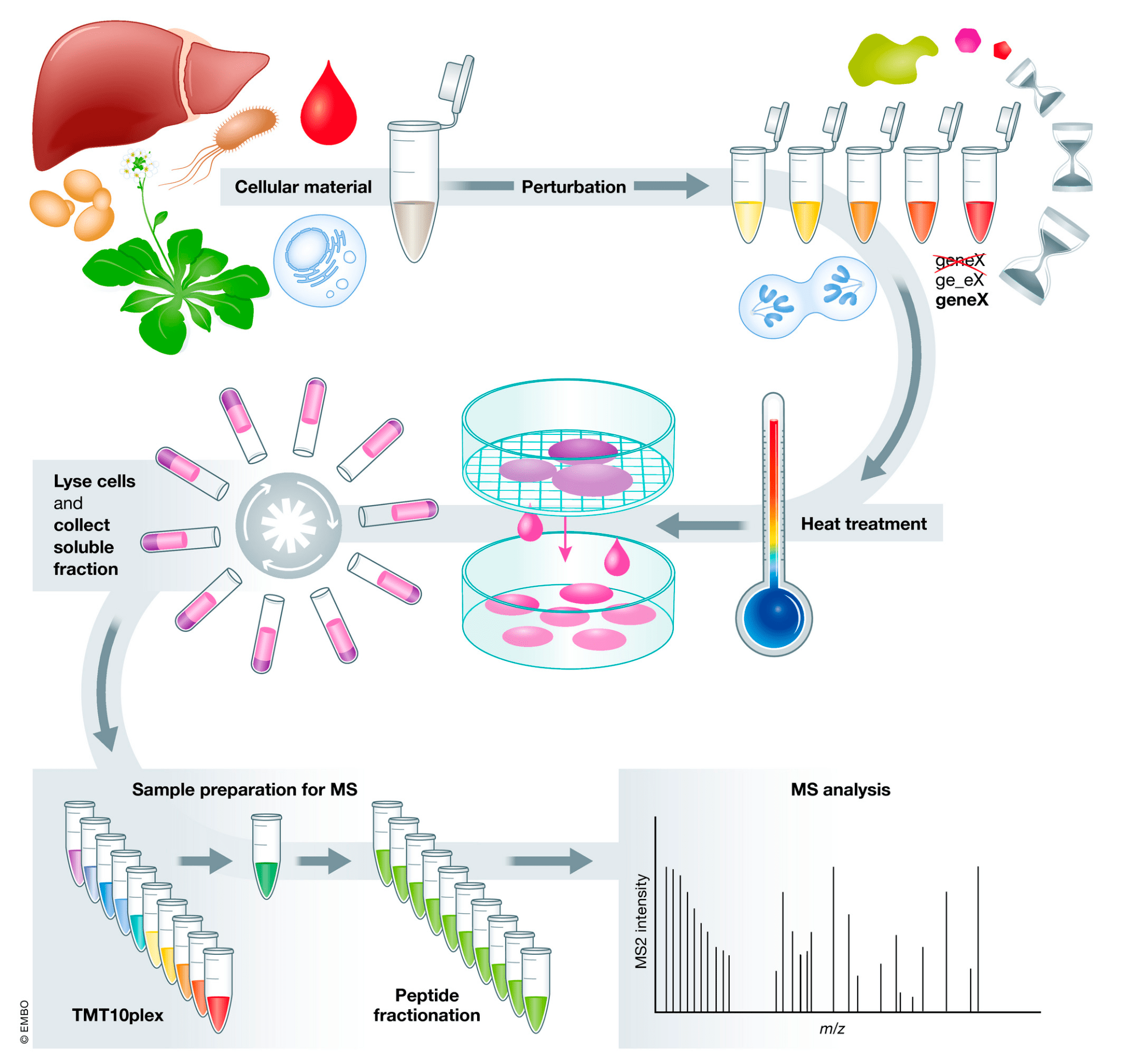
Рисунок 2. Температурное профилирование протеома (TPP). Биологический материал для анализа — клеточный экстракт, клетки, ткани или биологические жидкости, — подвергается воздействию (например, применению лекарства в разных дозах). Затем образцы нагревают, чтобы спровоцировать агрегацию белков. Оставшаяся жидкая фракция отделяется (обычно центрифугированием) и проводится протеомный анализ, чаще всего с помощью расщепления белков на пептиды трипсином и использования TMT-меток. Помеченные пептиды фракционируются и анализируются с помощью масс-спектрометрии.
Оказалось, несколько ингибиторов протеинкиназы (к которым относятся упомянутые лекарства) взаимодействуют с ферментом синтеза гема — феррохелатазой (FECH). Важно, что генетически обусловленный дефицит FECH вызывает порфирию — нарушение пигментного обмена, характеризующегося повышенной светочувствительностью. Соответственно, светочувствительность онкобольных, вызываемая этими лекарствами, объясняется взаимодействием препарата с FECH.
Интересно, что с помощью TPP можно определить, какие белки физически взаимодействуют с лекарством, а какие — действуют опосредованно, сравнив температурный профиль целых клеток и клеточного экстракта. В последнем случае мы узнаем только о прямых взаимодействиях лекарство–мишень. А вот при анализе клеток, в которых происходят активные процессы, TPP выявит и белки, вовлеченные в сигнальные пути белка-мишени.
Отметим, что TPP применимо не только к клеточным экстрактам и клеткам, но и тканям, органам, биологическим жидкостям человека и другим организмам in vitro, in situ или in vivo. Например, при помощи TPP можно изучить влияние антибиотиков на протеом бактерий [12]. Во времена нарастающей проблемы устойчивости к антибиотикам как никогда важными становятся эффективные методы тестирования новых молекул-кандидатов и понимания механизмов устойчивости к ним. Помимо прочего TPP как методология постоянно улучшается, и уже есть отчеты о ее применении для анализа мембранных белков (что изначально было невозможно) и даже для выявления мишеней лекарств прямо на целых органах животных [13].
Помимо протеомного анализа, описанного выше, транскриптомный профиль клеток также активно используется для оценки влияния лекарств. Так, например, исследователи оценивают разницу между применением нескольких лекарств отдельно или их комбинации, проводя РНК-секвенирование клеток, к которым применяются эти лекарства [13]. В последнее время все больший интерес вызывает синергетический эффект комбинаций препаратов, который, однако, сложно предсказать. С одной стороны, сочетание нескольких терапий для лечения одного заболевания может звучать как отличная идея. С другой, не всегда та комбинация, которая кажется очевидной, действительно сработает, как мы ожидаем. Беспристрастный анализ транскриптома клеток, подверженных воздействию каждого отдельного лекарства и их комбинаций, выявляет, какой из подходов эффективен. Здесь возникает проблема: тестирование многочисленных комбинаций лекарств с последующим секвенированием клеток каждого образца ведет за собой огромные затраты. Чтобы сократить потраченное время и средства, были разработаны способы компьютерного анализа, о которых речь в следующей главе.
Омиксные данные в поисковых исследованиях
Обратимся теперь к более фундаментальному использованию омиксных данных — самым ранним фазам доКИ. Здесь речь идет о том, чтобы собрать беспристрастные обширные данные для выявления потенциальных мишеней и подбора молекул-кандидатов. Подразумевается, что таким образом можно:
- обнаружить механизмы развития заболевания и выбрать мишени без заранее сформированного взгляда о тех сигнальных путях, которые задействованы в патологическом процессе;
- проводить валидацию мишеней, подтверждать механизм действия кандидата, а также вычислить его токсичность;
- сделать драг-дизайн в целом более быстрым и персонализированным процессом.
Раньше такие поисковые исследования назывались разработкой и, возможно, выносились за рамки доКИ. Это в основном исследования in silico, и на данный момент за ними все равно следует тестирование молекул-кандидатов на животных перед тем, как перейти в КИ. Однако амбициозность такого подхода позволяет значительно сократить стадию разработки лекарства, пропустить некоторые стадии доКИ, а иногда и КИ, стирает и без того расплывчатую грань между разработкой лекарств и непосредственно доКИ.
Разработка лекарств начинается с выбора мишени, что, как правило, основывается на уже сформированном взгляде о механизмах заболевания исходя из данных, полученных из предыдущих фундаментальных исследований [14], [15]. Новизна использования омиксных данных для поиска мишеней заключается в том, что никаких предположений о механизме развития заболевания у исследователей заранее как бы нет. Напротив, собирают все данные одного или нескольких уровней (например, транскриптом больных и здоровых людей), сравнивают и выявляют профиль экспрессии заболевания (набор генов, изменивших экспрессию). Далее подбирают вещества, способные вернуть экспрессию на уровень здоровых клеток. Такой подход еще называют системным (systems-based) — в отличие от подхода, основанного на мишени (target-based).
Разработан целый ряд методов оценки способности лекарства обратить вспять профиль экспрессии при заболевании. Некоторые исследователи используют специально созданный показатель — reverse gene expression score (RGES). Так, например, молекулы-кандидаты, обладающие наиболее высоким RGES, оказались эффективными при последующей проверке на раковых клетках печени [16].
Теоретически весь этот процесс (исключая валидацию на клетках и животных) можно провести, не отходя от экрана компьютера. Действительно, для некоторых заболеваний уже есть опубликованные данные экспрессии. Остается лишь сравнить их с профилем экспрессии здоровой ткани / людей, получив профиль экспрессии заболевания. Далее нужны данные о характеристиках лекарств, чтобы выбрать те, которые подходят в нашем случае . Это тоже не проблема, ведь существуют такие базы данных как ChEMBL. А в L1000 Viewer можно найти, на экспрессию каких генов влияют те или иные лекарства. Сравнив профиль экспрессии заболевания с профилем экспрессии, на который влияют лекарства, можно выбрать подходящие из них, рассчитав RGES. Следуя этому алгоритму, удалось предсказать эффективность 6 кандидатов для лечения рака желудка (рис. 3) [17].
Кстати, таким образом можно искать не только лекарства, но и мишени.
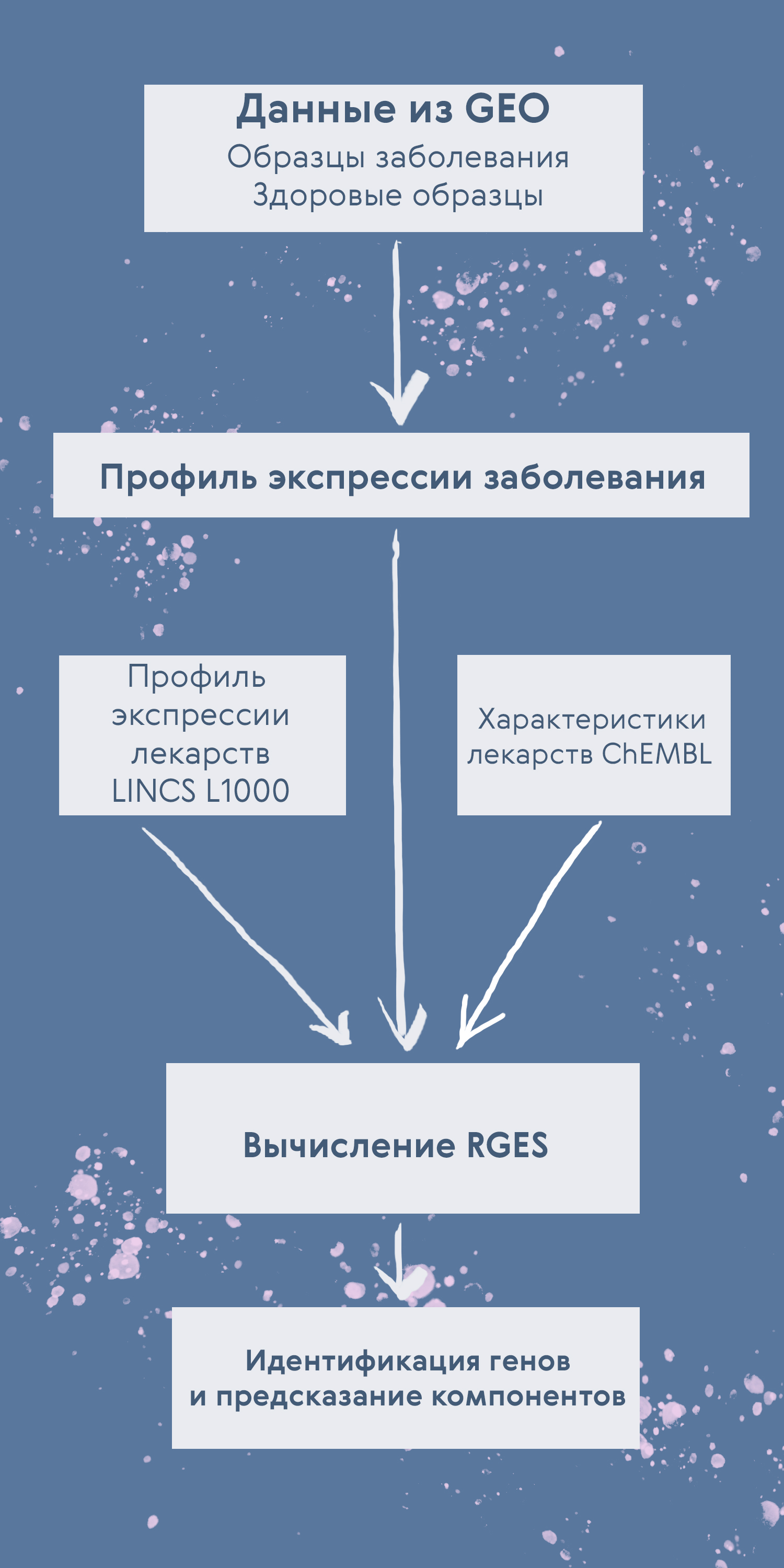
Рисунок 3. Алгоритм подбора лекарства путем расчета RGES. Сначала собирают данные экспрессии заболевания и здоровых образцов (база данных GEO) и вычисляют профиль экспрессии заболевания. Далее собирают данные о профилях экспрессии лекарств (база данных LINCS L1000) и характеристиках лекарств (база ChEMBL). Эти данные интегрируют и вычисляют RGES. Наконец, определяют гены, экспрессия которых изменена в результате применения лекарств, и предсказывают эффективность препарата.
иллюстрация Карины Илиопуло
А еще до появления RGES с помощью алгоритмов глубокого обучения исследователи классифицировали различные лекарства в несколько заранее известных терапевтических категорий на основе их транскрипционных профилей [18]. Здесь использовали данные о том, на какие сигнальные пути и важные гены (landmark genes) воздействуют лекарства при их применении в разных клеточных линиях и дозировках. В итоге показатель активации сигнальных путей (pathway activation score) оказался наиболее успешным в предсказании фармакологических свойств лекарств. Интересно, что такой анализ поместил некоторые лекарства в «неправильные категории», что может говорить о возможности их использования в новой роли. Например, алгоритм отнес антагонист мускариновых рецепторов отензепад в категорию лекарств для ЦНС, а он обычно используется только для сердечной аритмии, несмотря на важную роль этих рецепторов в функционировании мозга.
Такой system-based омиксный подход порой приводит к тому, что при заболевании обнаруживается сразу несколько мишеней, а соответственно, возникает необходимость подбора нескольких лекарств (polytherapy — политерапия). Для этого тоже есть определенные платформы (например, Drugbank). Политерапия — логичный подход, ведь многие заболевания — многофакторные, и в таких случаях лекарство, воздействующее на одну мишень, фактически обречено на провал. Но политерапия может сослужить добрую службу и при моногенных заболеваниях. Например, лекарство, на данный момент находящееся на фазе III КИ для редкой генетической болезни Шарко—Мари—Тута (БШМТ), состоит из трех малых молекул [19]. Они были подобраны с помощью беспристрастного анализа молекулярных сетей, вовлеченных в развитие заболевания, на основе GWAS (англ. genome-wide association studies — полногеномный поиск ассоциаций для определения снипов, связанных с заболеваниями).
Из библиотеки лекарств, уже доступных на рынке и одобренных для других заболеваний, изначально отобрали ~50 кандидатов, подходящих по профилю безопасности, токсичности, фармакокинетики и т.д. Такой процесс получил название drug repurposing, и позволил компании Pharnext пропустить фазу I КИ. Отметим, что в подборе компонентов на основе геномных данных компании помогли алгоритмы ИИ. В доКИ in vitro и in vivo скрининг перешли несколько комбинаций кандидатов, из которых потом одна вышла на стадию тестирования на людях. На данный момент компания вывела уже две комбинации в КИ — для БШМТ и болезни Альцгеймера.
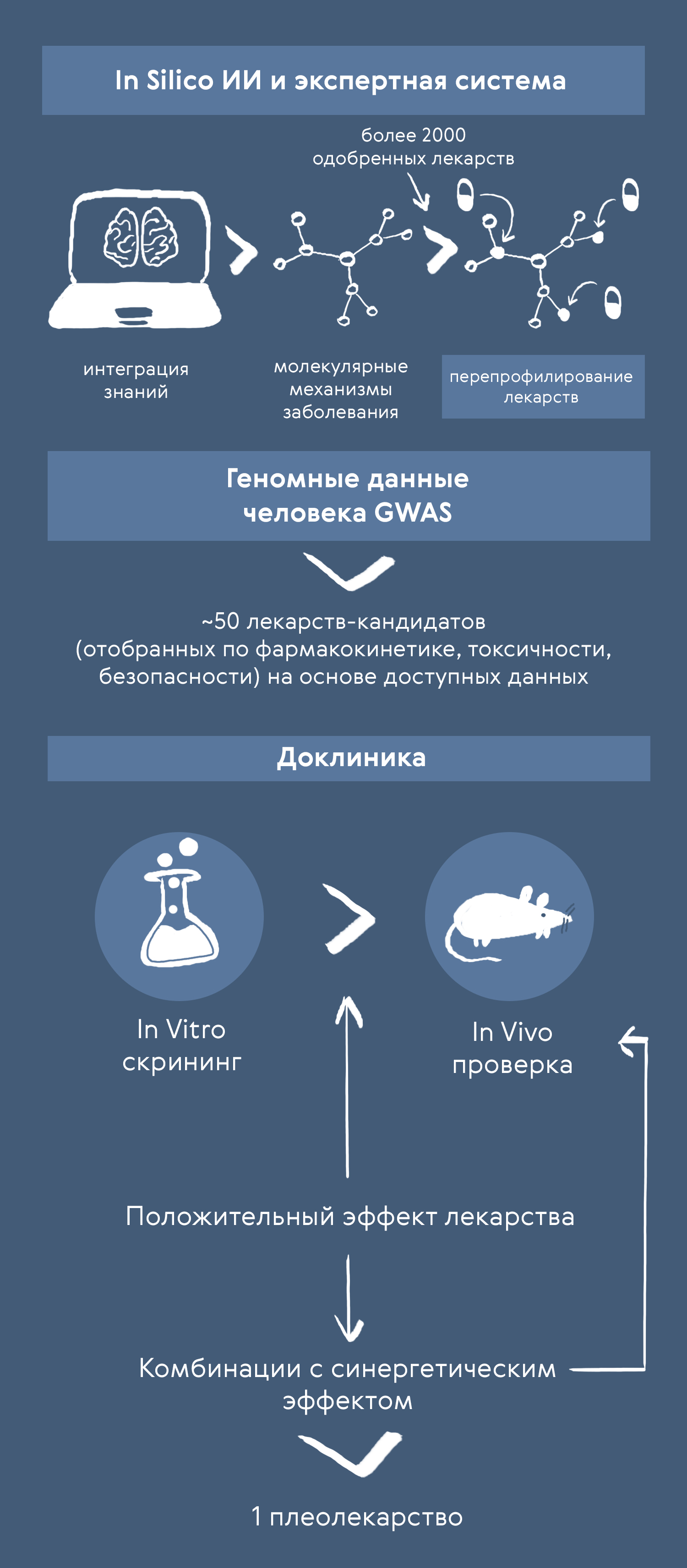
Рисунок 4. Подход Pharnext: интеграция опубликованных омиксных данных для поиска механизмов заболевания и подбора комбинации из уже одобренных для других заболеваний лекарств. На стадию доКИ выходит ~50 кандидатов, которые далее тестируются in vitro и in vivo. Одна успешная комбинация (плеолекарство) переходит на стадию КИ.
иллюстрация Карины Илиопуло по изображению Pharnext
Pharnext — не исключение, когда речь заходит о применении методов машинного обучения для подбора лекарств на основе омиксных данных. Это объясняется сложностью и огромными размерами таких данных, что требует специфических методов обработки. Еще один яркий пример — Insilico Medicine. Это ИИ-биотех-компания, разрабатывающая лекарства in silico, как очевидно следует из названия. Компания внедряет методы машинного обучения на каждом этапе для дизайна новых малых молекул с определенными характеристиками. Они работают с мультиомиксными данными, выявляют набор белков-мишеней и определяют, какие молекулы-кандидаты можно применить для лечения заболевания. Фактически, каждый шаг выполняется с помощью автоматизированных ИИ-платформ. С помощью PandaOmics исследователи могут интерпретировать мультиомиксные данные и выбрать сигнальные пути-мишени для подбора лекарств. Эта платформа также помогает исследовать, насколько подкреплена связь между определенными мишенями и заболеванием на основе опубликованных данных. Мишени фильтруются и ранжируются на основе того, насколько они податливы воздействию малых молекул, и не будет ли токсичным такое воздействие на них.
Например, не стоит воздействовать на гены, нокаут которых у мышей летален. Далее алгоритмы могут предсказать, какова вероятность выхода лекарства, направленного на определенную мишень, в I фазу клиники. Наконец, с помощью платформы Chemistry42 можно разработать молекулы-кандидаты с нуля, задав необходимые параметры, такие как структура (для взаимодействия с мишенью), стабильность, сложность синтеза молекулы, подобия лекарству (drug-likeness) и т.д. А наиболее успешные молекулы затем тестируются в биологических системах.
Основное преимущество такого хода работы — весь процесс от обработки омиксных данных до финального дизайна лекарства проходит в одном месте: нет необходимости комбинировать данные из разных баз данных, мучаясь с разными форматами и способами анализа (рис. 5). Успех подхода Insilico Medicine был в очередной раз продемонстрирован, когда компания объявила о доКИ молекулы-кандидата для лечения фиброза почек, которые показали желаемые фармакологические свойства, фармакокинетический профиль, а также обнадеживающие результаты in vitro и in vivo. Весь процесс уложился всего в 18 месяцев, а обычно занимает годы. Ранее же с помощью PandaOmics компания идентифицировала принципиально новую мишень для идиопатического фиброза легких и сейчас готовится к проведению первых в своей истории клинических испытаний.
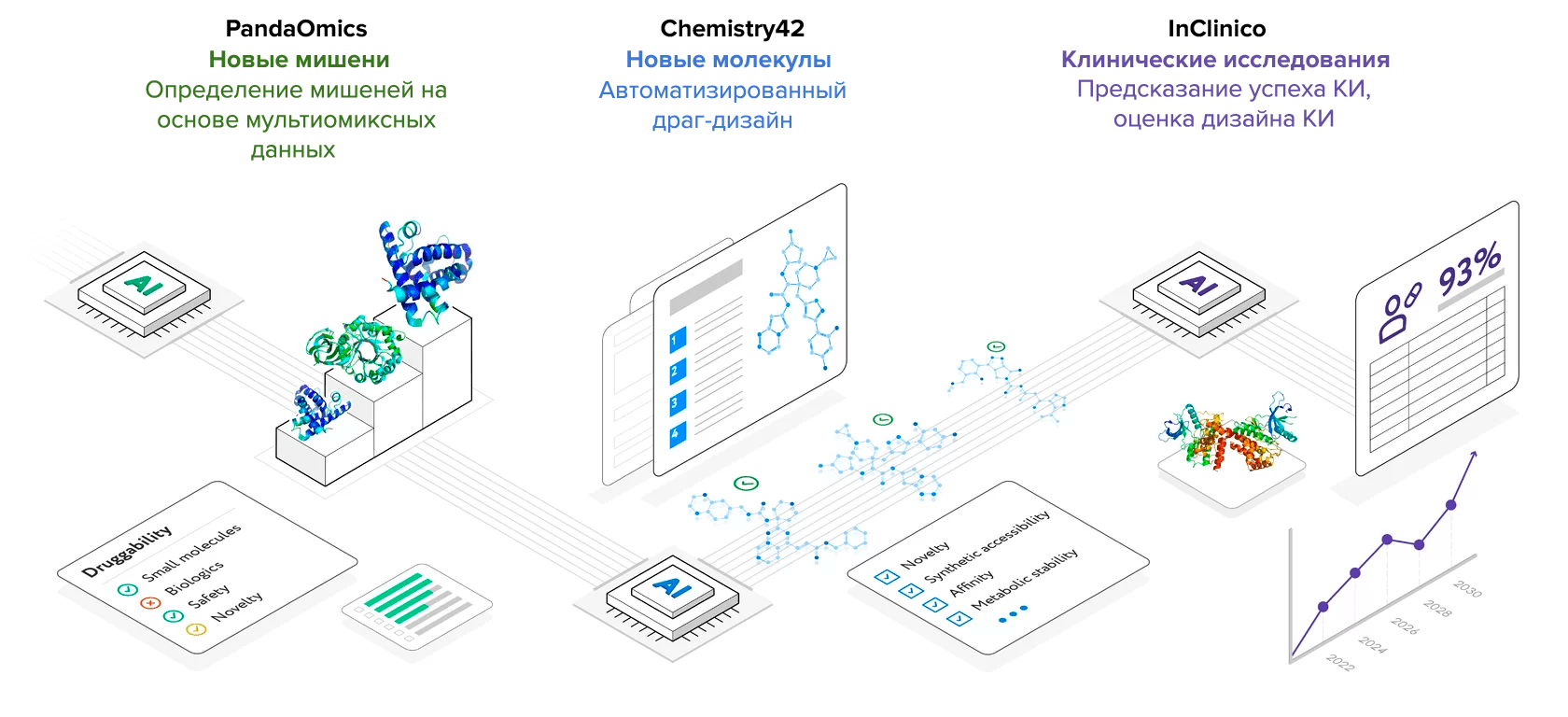
Рисунок 5. Подход Insilico Medicine заключается в ИИ-автоматизации всего процесса, начиная с анализа мультиомиксных данных для определения мишеней и заканчивая виртуальным синтезом молекул-кандидатов, а также предсказанием успеха КИ.
Персонализированная медицина и взгляд в будущее
Конечной целью разработки лекарств и доКИ является создание эффективного и безопасного лекарства. В последнее время все чаще приходит осознание того, что для достижения этой цели необходимо принимать во внимание индивидуальные особенности больных. Ведь одно и то же лекарство может показывать разную эффективность на разных людях с одним и тем же заболеванием. Кроме того, у кого-то лекарство вызывает побочные эффекты, а у кого-то — нет. Омиксные данные обладают потенциалом приблизить нас к внедрению персонализированной медицины и избежать подобной ситуации.
«Биомолекула» уже освещала тему фармакогеномики, когда учитывается вариативность генома разных больных при разработке и назначении лекарств [20]. Однако наряду с фармакогеномикой появились фармакометаболомика, фармакопротеомика и т.д. Одной из целей всех этих наук является разделение пациентов (стратификация) согласно их омиксному профилю и применение тех лекарств, которые подходят конкретной группе (рис. 6). Так, в рамках соревнования sbv IMPROVER (см. врезку) одним из челленджей выступила классификация клинических образцов на основе транскриптомных данных для понимания, каким пациентам лучше всего подойдет та или иная терапия [21].
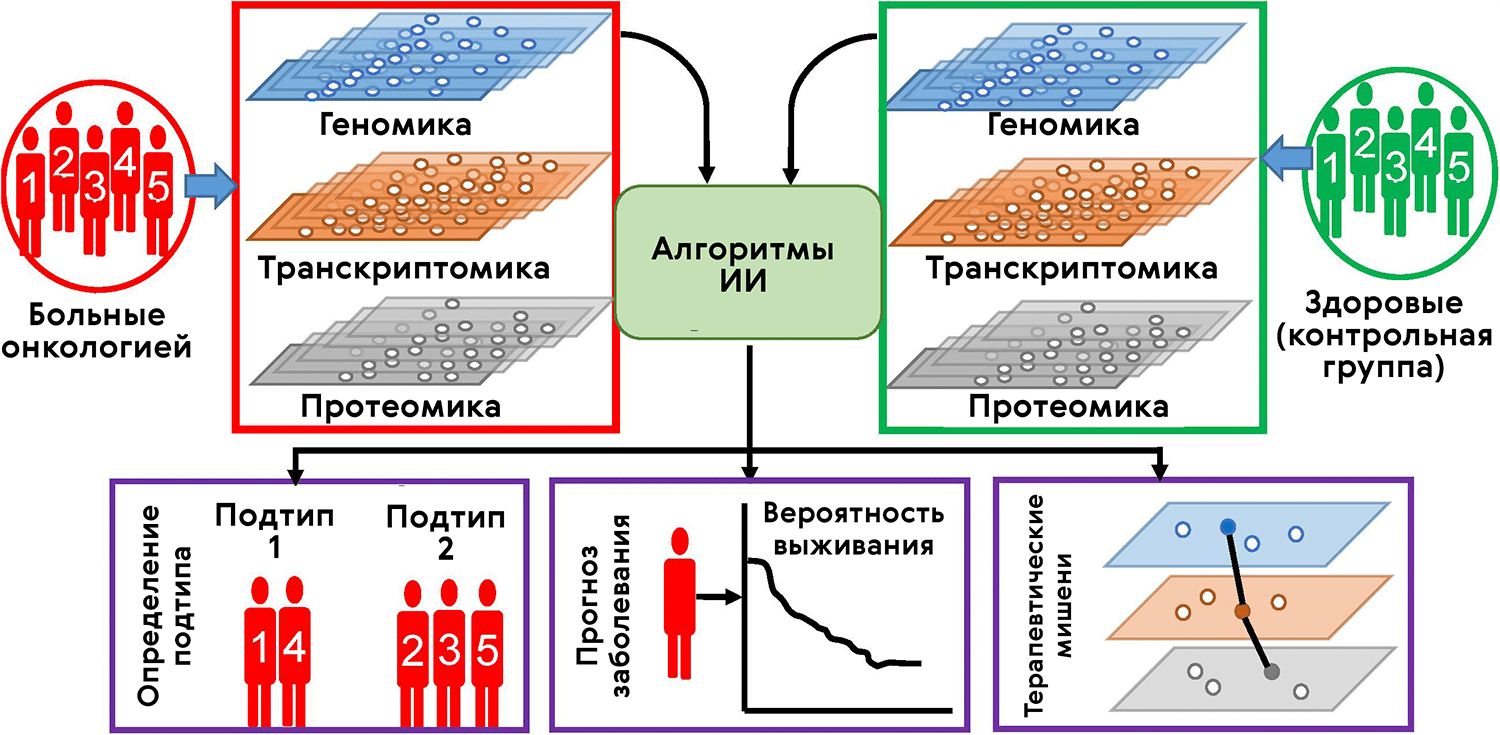
Рисунок 6. ИИ-анализ мультиомиксных данных для стратификации онкобольных и выявления мишеней
Ну разве не заманчивая картина: у пациента собирают омиксные данные, а на выходе говорят, какое лекарство подойдет лучше всего именно ему? Последующий мониторинг реакции больного на терапию сможет определить, необходимы ли изменения дозировки, времени приема, либо замена на другое лекарство (рис. 7).
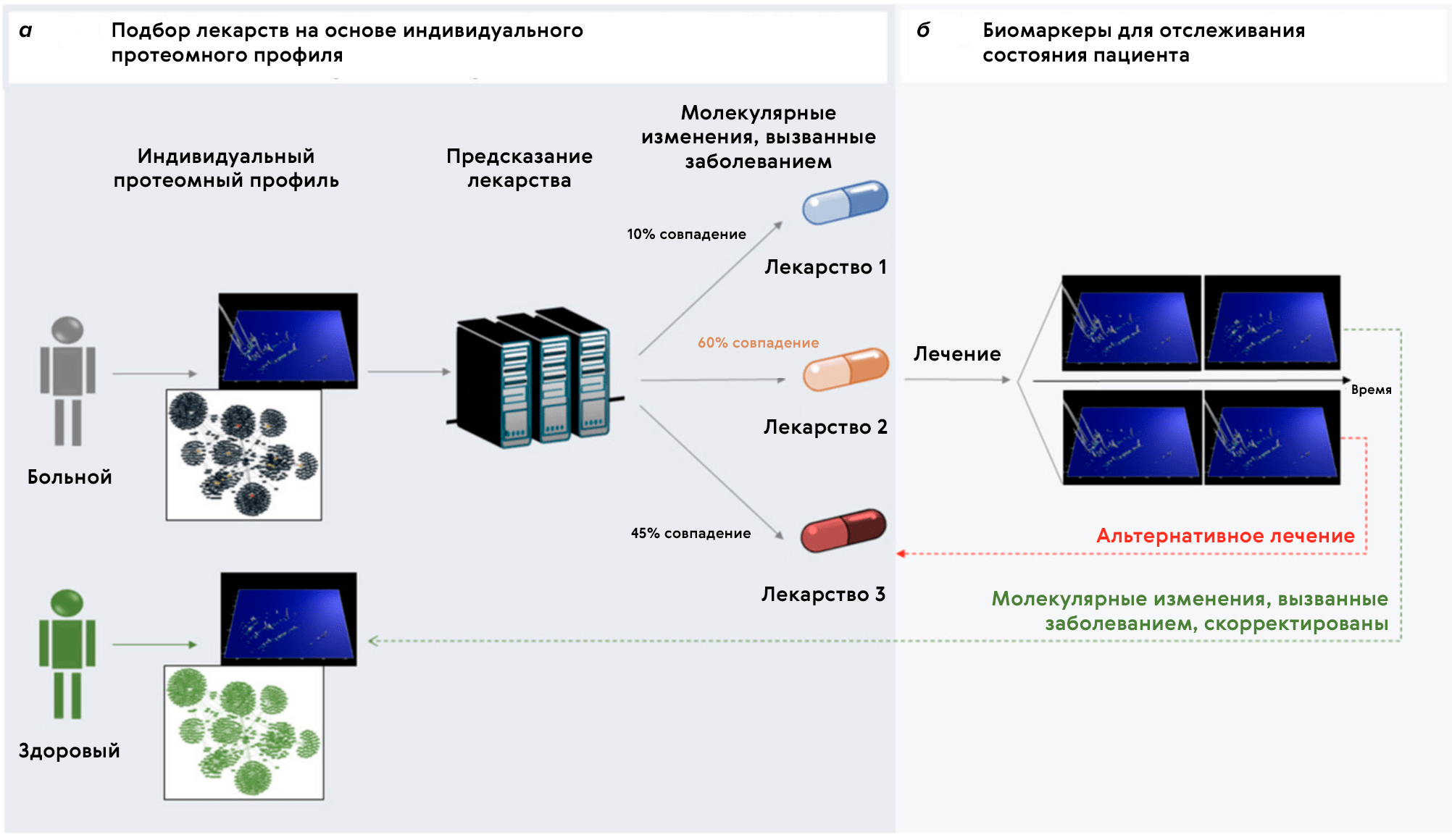
Рисунок 7. Предполагаемая картина выбора терапии на основе омиксного профиля пациента. Оценивается вероятность успеха лекарств и выбирается то, которое подходит лучше всех (в данном случае Лекарство 2). Спустя некоторое время мониторинга при условии провала подобранного лекарства оно заменяется на другое (Лекарство 3).
Вероятно, прекрасное будущее, когда наши мультиомиксные данные загружаются на одну платформу, подбирающую индивидуальное лекарственное лечение, — не за горами. Когда нет необходимости ждать проверки терапии на животных и бояться страшных побочек. А пока порадуемся тому, как омиксные данные и ИИ-алгоритмы помогают сократить время и средства, потраченные на разработку и тестирование лекарств.
Большие данные требуют больших усилий
Омиксные данные в корне меняют наше представление о разработке лекарств. Вместо того чтобы исходить из уже известных механизмов заболеваний, они предлагают начать с чистого листа: собрать все, что можно, проанализировать и лишь потом делать выводы. Более того, пошаговый анализ человеком сменяется автоматизированным анализом ИИ, который может выявлять паттерны и взаимосвязи мишень—кандидат, неподвластные человеческому разуму.
Многомерность — отличительная черта омиксных данных: сравнивая генетические профили тканей больных и здоровых людей, на выходе получаем сотни, а иногда тысячи дифференциально экспрессируемых генов. И чтобы понять, что же именно это значит, необходим очень мощный анализ сигнальных путей, который превратил бы сырые неструктурированные данные в несколько ключевых и легко интерпретируемых характеристик. Есть разные вариации таких методов [24–26], но суть одна — сосредоточиться на тех путях, молекулы-участники которых наиболее важны для понимания заболевания и определения мишеней.
Омиксные данные и их анализ едва ли облегчают учесть исследователей: теперь помимо знания командной строки Linux важно не только различать supervised и unsupervised learning, но и молекулярные пути не забывать. Ведь без биологически релевантной интерпретации таких данных — это просто набор молекул. Но, вероятно, именно так получится разрабатывать лекарства быстрее, дешевле и надежнее.
Литература
- От живого к неживому. Доклинические исследования сегодня и завтра;
- Лекарство в пробирке: доклинические исследования in vitro;
- Органы-на-чипе: когда технология сплетается с жизнью;
- «Омики» — эпоха большой биологии;
- Я б в биоинформатики пошёл, пусть меня научат!;
- Поиск лекарственных мишеней;
- Anna Cichońska, Balaguru Ravikumar, Robert J. Allaway, Fangping Wan, Sungjoon Park, et. al.. (2021). Crowdsourced mapping of unexplored target space of kinase inhibitors. Nat Commun. 12;
- Daniel Martinez Molina, Rozbeh Jafari, Marina Ignatushchenko, Takahiro Seki, E. Andreas Larsson, et. al.. (2013). Monitoring Drug Target Engagement in Cells and Tissues Using the Cellular Thermal Shift Assay. Science. 341, 84-87;
- 12 методов в картинках: протеомика;
- André Mateus, Nils Kurzawa, Isabelle Becher, Sindhuja Sridharan, Dominic Helm, et. al.. (2020). Thermal proteome profiling for interrogating protein interactions. Mol Syst Biol. 16;
- Mikhail M. Savitski, Friedrich B. M. Reinhard, Holger Franken, Thilo Werner, Maria Fälth Savitski, et. al.. (2014). Tracking cancer drugs in living cells by thermal profiling of the proteome. Science. 346;
- André Mateus, Jacob Bobonis, Nils Kurzawa, Frank Stein, Dominic Helm, et. al.. (2018). Thermal proteome profiling in bacteria: probing protein state in vivo. Mol Syst Biol. 14;
- Jessica Perrin, Thilo Werner, Nils Kurzawa, Anna Rutkowska, Dorothee D. Childs, et. al.. (2020). Identifying drug targets in tissues and whole blood with thermal-shift profiling. Nat Biotechnol. 38, 303-308;
- Виртуальные тропы реальных лекарств;
- Драг-дизайн: как в современном мире создаются новые лекарства;
- Bin Chen, Li Ma, Hyojung Paik, Marina Sirota, Wei Wei, et. al.. (2017). Reversal of cancer gene expression correlates with drug efficacy and reveals therapeutic targets. Nat Commun. 8;
- In-Wha Kim, Hayoung Jang, Jae Hyun Kim, Myeong Gyu Kim, Sangsoo Kim, Jung Mi Oh. (2019). Computational Drug Repositioning for Gastric Cancer using Reversal Gene Expression Profiles. Sci Rep. 9;
- Alexander Aliper, Sergey Plis, Artem Artemov, Alvaro Ulloa, Polina Mamoshina, Alex Zhavoronkov. (2016). Deep Learning Applications for Predicting Pharmacological Properties of Drugs and Drug Repurposing Using Transcriptomic Data. Mol. Pharmaceutics. 13, 2524-2530;
- Одна болезнь, сто генов, миллионы людей: как вылечить болезнь Шарко—Мари—Тута;
- Фармакогеномика: изучение генов на службе персонализированной медицины;
- Adi L. Tarca, Mario Lauria, Michael Unger, Erhan Bilal, Stephanie Boue, et. al.. (2013). Strengths and limitations of microarray-based phenotype prediction: lessons learned from the IMPROVER Diagnostic Signature Challenge. Bioinformatics. 29, 2892-2899;
- Kahn Rhrissorrakrai, Vincenzo Belcastro, Erhan Bilal, Raquel Norel, Carine Poussin, et. al.. (2015). Understanding the limits of animal models as predictors of human biology: lessons learned from the sbv IMPROVER Species Translation Challenge. Bioinformatics. 31, 471-483;
- Модельные организмы: грызуны;
- Ivan V. Ozerov, Ksenia V. Lezhnina, Evgeny Izumchenko, Artem V. Artemov, Sergey Medintsev, et. al.. (2016). In silico Pathway Activation Network Decomposition Analysis (iPANDA) as a method for biomarker development. Nat Commun. 7;
- Adi Laurentiu Tarca, Sorin Draghici, Purvesh Khatri, Sonia S. Hassan, Pooja Mittal, et. al.. (2009). A novel signaling pathway impact analysis. Bioinformatics. 25, 75-82;
- A. Subramanian, P. Tamayo, V. K. Mootha, S. Mukherjee, B. L. Ebert, et. al.. (2005). Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences. 102, 15545-15550;
- Nupur Biswas, Saikat Chakrabarti. (2020). Artificial Intelligence (AI)-Based Systems Biology Approaches in Multi-Omics Data Analysis of Cancer. Front. Oncol.. 10;
- Maria Frantzi, Agnieszka Latosinska, Harald Mischak. (2019). Proteomics in Drug Development: The Dawn of a New Era?. Prot. Clin. Appl.. 13, 1800087.