Самые нестандартные генетические коды
10 августа 2018
Самые нестандартные генетические коды
- 3186
- 0
- 3
Сегодня известно не менее трех десятков исключений из стандартного генетического кода
-
Автор
-
Редактор
После открытия правил генетического кода, по которым наследственная информация переписывается с языка нуклеотидов на язык аминокислот, они считались универсальными. Известно не менее 30 случаев, когда генетический код используется в несколько измененном виде. Изменения могут быть самыми разнообразными: изменится значение кодона, стоп-кодон начнет кодировать какую-то аминокислоту, обычный кодон начнет выполнять роль стартового. Мы предлагаем вам десять случаев наиболее любопытных отклонений от стандартного генетического кода.
Несмотря на общепринятую «стандартность» генетического кода, известно несколько десятков примеров, когда живые организмы используют несколько измененную его версию. Некоторые изменения присущи целым таксонам, а некоторые обнаруживаются всего у нескольких видов. Известны случаи, когда часть мРНК определенного гена транслируется по стандартным правилам, а другая — по измененным. Например, при трансляции мРНК малатдегидрогеназы человека, которая закодирована в ядре, в 4% случаев стандартный стоп-кодон кодирует триптофан и аргинин [1]. Очень часто отклонения от стандартного генетического кода наблюдаются только в некоторых органеллах. Так, впервые факт существования таких отклонений подтвердили еще в 1979 году, показав, что генетический код митохондрий человека отличается от ядерного [2]. Наша статья посвящена наиболее удивительным случаям отклонения генетического кода от стандарта .
«Биомолекула» не раз писала о генетическом коде. Статья «Такие разные синонимы» [3] посвящена явлению предпочтения кодонов. В статьях «У истоков генетического кода: родственные души» [4] и «Эволюция генетического кода» [5] рассказывается об эволюции генетического кода, а в публикациях «Расширенный геном» [6] и «Слово из четырёх букв» [7] можно почитать о перспективах его искусственного расширения.
Blastocrithidia
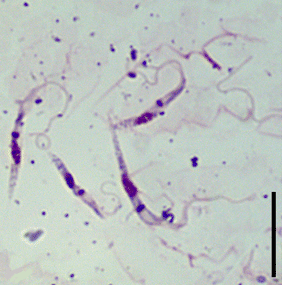
Рисунок 1. Клетки Blastocrithidia papi (окрашивание по Гимзе)
[19], рисунок с изменениями
У простейших рода Blastocrithidia, родственных трипаносомам (рис. 1), генетический код, используемый при трансляции ядерных генов, в прямом смысле «без тормозов»: все три стоп-кодона кодируют аминокислоты. Кодон UGA кодирует триптофан, а UAG и UAA — глутамат. При этом UAA и, реже, UAG все-таки могут выступать в роли терминаторных кодонов. Оказалось, что у одного из белков, необходимых для освобождения рибосомы от мРНК после трансляции, eRF1, чрезвычайно важный остаток серина заменен на другую аминокислоту, что понижает его сродство к UGA, благодаря чему этот стоп-кодон может функционировать как смысловой. Впрочем, окончательно неизвестно, благодаря чему UAG и UAA могут выступать и как смысловые, и как терминаторные кодоны [8].
Condylostoma magnum
У инфузории Condylostoma magnum каждый из стандартных стоп-кодонов способен выступать в роли смыслового: UAA и UAG могут кодировать глутамин, а UGA — триптофан. Однако механизм двойного кодирования у этого организма совершенно отличается от Blastocrithidia: значение каждого из стандартных стоп-кодонов зависит от их положения в мРНК. Стоп-кодоны, расположенные в средней части транскрипта, кодируют аминокислоты, а стоп-кодоны, находящиеся вблизи 3′-конца мРНК, работают «по специальности» и выполняют роль терминаторных. Вероятно, 3′-нетранслируемые области генов Condylostoma magnum очень короткие и консервативные и играют роль в распознавании стоп-кодонов [9].
Acetohalobium arabaticum
Бактерия Acetohalobium arabaticum (рис. 2) может переключаться со стандартного генетического кода на нестандартный в зависимости от внешних условий, а именно источника энергии. При выращивании на среде с пируватом эти бактерии пользуются обычным генетическим кодом, но в присутствии триметиламина стоп-кодон UAG начинает кодировать нестандартную аминокислоту пирролизин. Оказалось, что в отсутствие триметиламина у Acetohalobium arabaticum оперон, необходимый для синтеза пирролизина, подавлен. Остатки пирролизина у этой бактерии входят в состав таких белков, как две метиламинметилтрансферазы, необходимые для метаболизма триметиламина [10].
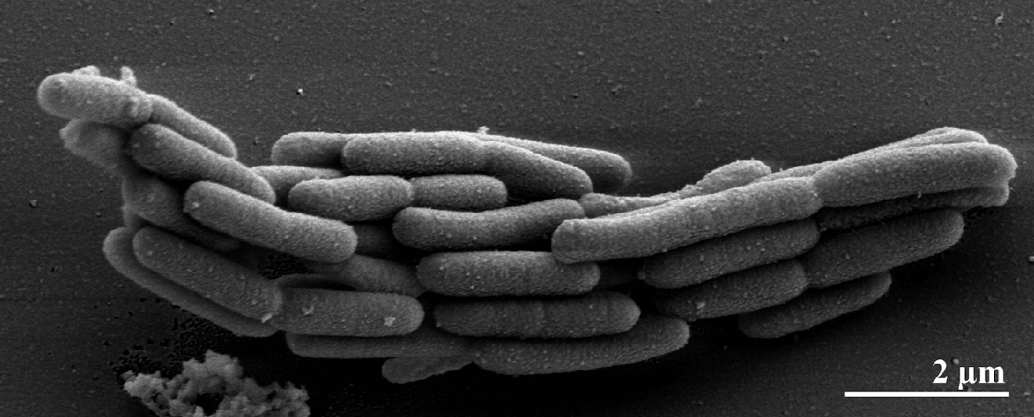
Рисунок 2. Acetohalobium arabaticum (сканирующая электронная микроскопия)
Rhabdopleura compacta
Rhabdopleura compacta — вторичноротое животное, входящее в состав типа полухордовые. В митохондриях этого животного стоп-кодон UGA кодирует триптофан, а кодоны AGA и AGG кодируют вместо аргинина серин и лизин соответственно. Любопытно, что AGG, кодирующий лизин, больше не встречается ни у кого из вторичноротых животных и обнаружен только у некоторых членистоногих. И у них, и у Rhabdopleura compacta в основе изменения значения кодона AGG лежит одна и та же мутация в антикодоновой петле тРНК, соответствующей лизину [11].
Scenedesmus obliquus
Генетический код митохондрий зеленой водоросли Scenedesmus obliquus (рис. 3) необычен тем, что кодон UCA, который обычно кодирует лейцин, функционирует как стоп-кодон. В митохондриальном геноме этой водоросли отсутствует ген, кодирующий тРНК, соответствующую кодону UCA. Вместо этого в митохондриях Scenedesmus obliquus лейцин кодирует стандартный стоп-кодон UAG [12].
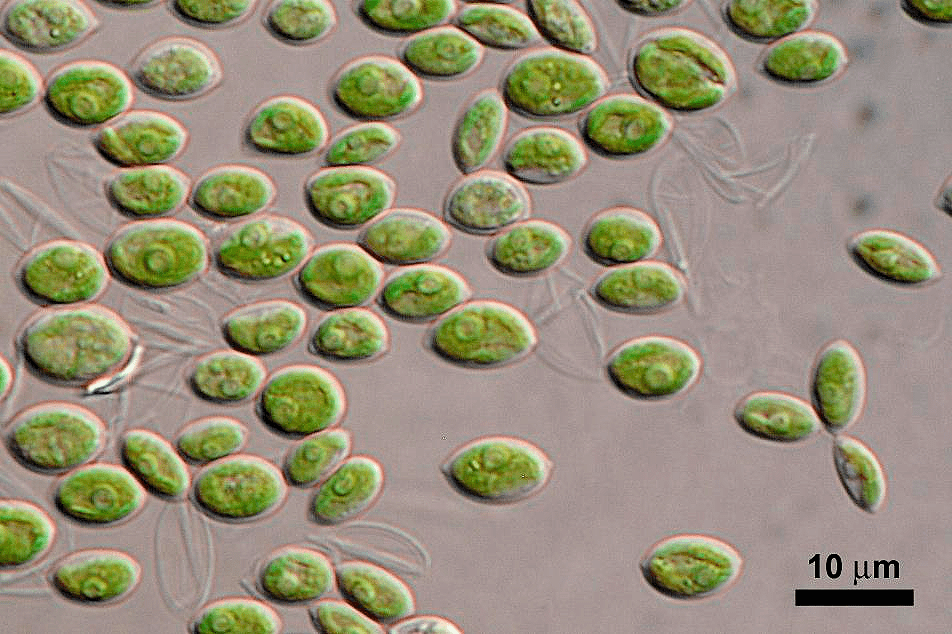
Рисунок 3. Клетки Scenedesmus obliquus
Плоские черви класса Rhabditophora
Класс плоских червей Rhabditophora может служить отличным примером того, как изменения стандартного генетического кода оказываются присущи всем представителям большого таксона. У всех плоских червей, входящих в этот класс, кодон AAA кодирует аспарагин вместо лизина, а AUA — изолейцин вместо метионина [13].
Radopholus similis
У паразитической нематоды Radopholus similis (рис. 4) в митохондриальном геноме стоп-кодон UAA кодирует тирозин — уникальный случай для животного мира. Кроме того, 7 из 12 белок-кодирующих генов в митохондриальном геноме не имеют стоп-кодона. У многих других организмов в митохондриальном геноме тоже есть гены, не имеющие стоп-кодонов, однако у них стоп-кодон UAA появляется в соответствующих транскриптах после полиаденилирования. У Radopholus similis, похоже, транскрипты без стоп-кодона не полиаденилируются, и механизм терминации трансляции в этом случае пока неизвестен [14].

Рисунок 4. Самка Radopholus similis. а — Головной отдел, б — Общий вид (v — вульва).
Инфузории-туфельки
Митохондриальный генетический код инфузорий-туфелек (род Paramecium) отличается от стандартного прежде всего числом стартовых кодонов. В роли старт-кодонов могут выступать целых пять или шесть: AUG, AUA, AUU, AUC, GUG и, возможно, GUA. Поскольку митохондриальный геном этих организмов содержит гены всего трех тРНК, бóльшая часть тРНК поступает из цитоплазмы. В связи с этим в митохондриях инфузорий-туфелек, как и в ядре многих инфузорий, стоп-кодоны UAG и UAA кодируют глутамин [15].
Ashbya gossypii
У дрожжей Ashbya gossypii в митохондриях кодон CUU, обычно кодирующий лейцин, кодирует аланин. Удивительно, что два других лейциновых кодона, CUC и CUG, в митохондриальном геноме полностью отсутствуют, поэтому у этих организмов лейцин кодируется только двумя кодонами — UUG и UUA — вместо стандартных пяти [16].
Mycobacterium smegmatis
У бактерии Mycobacterium smegmatis аспарагиновые кодоны приобретают дополнительное значение в стационарной фазе роста, а также в условиях низкого pH. Еще более любопытно, что, благодаря двусмысленности аспарагиновых кодонов, в β-субъединице РНК-полимеразы происходят замены, которые сохраняют ее функциональность, однако делают фермент устойчивым к антибиотику рифампицину, в норме блокирующему его работу [17], [18].
Разумеется, вариации стандартного генетического кода не ограничиваются приведенными примерами. Однако исключения только подтверждают правило, и это верно и для генетического кода. Несмотря на колоссальное разнообразие живых организмов, исключения из генетического кода настолько редки, что представляются не более чем любопытными курьезами. Однако эти исключения служат ценным материалом для реконструкции эволюции генетического кода и помогают глубже понять его фундаментальные свойства.
Литература
- Julia Hofhuis, Fabian Schueren, Christopher Nötzel, Thomas Lingner, Jutta Gärtner, et. al.. (2016). The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code. Open Biol.. 6, 160246;
- B. G. Barrell, A. T. Bankier, J. Drouin. (1979). A different genetic code in human mitochondria. Nature. 282, 189-194;
- Такие разные синонимы;
- У истоков генетического кода: родственные души;
- Эволюция генетического кода;
- Расширенный геном;
- Слово из четырёх букв;
- Kristína Záhonová, Alexei Y. Kostygov, Tereza Ševčíková, Vyacheslav Yurchenko, Marek Eliáš. (2016). An Unprecedented Non-canonical Nuclear Genetic Code with All Three Termination Codons Reassigned as Sense Codons. Current Biology. 26, 2364-2369;
- Stephen M. Heaphy, Marco Mariotti, Vadim N. Gladyshev, John F. Atkins, Pavel V. Baranov. (2016). Novel Ciliate Genetic Code Variants Including the Reassignment of All Three Stop Codons to Sense Codons inCondylostoma magnum. Mol Biol Evol. 33, 2885-2889;
- L. Prat, I. U. Heinemann, H. R. Aerni, J. Rinehart, P. O'Donoghue, D. Soll. (2012). Carbon source-dependent expansion of the genetic code in bacteria. Proceedings of the National Academy of Sciences. 109, 21070-21075;
- Marleen Perseke, Joerg Hetmank, Matthias Bernt, Peter F Stadler, Martin Schlegel, Detlef Bernhard. (2011). The enigmatic mitochondrial genome of Rhabdopleura compacta(Pterobranchia) reveals insights into selection of an efficient tRNA system and supports monophyly of Ambulacraria. BMC Evol Biol. 11;
- A. M. Nedelcu. (2000). The Complete Mitochondrial DNA Sequence of Scenedesmus obliquus Reflects an Intermediate Stage in the Evolution of the Green Algal Mitochondrial Genome. Genome Research. 10, 819-831;
- M. J. Telford, E. A. Herniou, R. B. Russell, D. T. J. Littlewood. (2000). Changes in mitochondrial genetic codes as phylogenetic characters: Two examples from the flatworms. Proceedings of the National Academy of Sciences. 97, 11359-11364;
- Joachim EM Jacob, Bartel Vanholme, Thomas Van Leeuwen, Godelieve Gheysen. (2009). A unique genetic code change in the mitochondrial genome of the parasitic nematode Radopholus similis. BMC Research Notes. 2, 192;
- Pritchard A.E., Seilhamer J.J., Mahalingam R., Sable C.L., Venuti S.E., Cummings D.J. (1990). Nucleotide sequence of the mitochondrial genome of Paramecium. Nucleic Acids Res. 18, 173–180;
- Jiqiang Ling, Rachid Daoud, Marc J. Lajoie, George M. Church, Dieter Söll, B. Franz Lang. (2014). Natural reassignment of CUU and CUA sense codons to alanine in Ashbya mitochondria. Nucleic Acids Research. 42, 499-508;
- Jiqiang Ling, Patrick O'Donoghue, Dieter Söll. (2015). Genetic code flexibility in microorganisms: novel mechanisms and impact on physiology. Nat Rev Micro. 13, 707-721;
- B. Javid, F. Sorrentino, M. Toosky, W. Zheng, J. T. Pinkham, et. al.. (2014). Mycobacterial mistranslation is necessary and sufficient for rifampicin phenotypic resistance. Proceedings of the National Academy of Sciences. 111, 1132-1137;
- Alexander O. Frolov, Marina N. Malysheva, Anna I. Ganyukova, Vyacheslav Yurchenko, Alexei Y. Kostygov. (2017). Life cycle of Blastocrithidia papi sp. n. (Kinetoplastea, Trypanosomatidae) in Pyrrhocoris apterus (Hemiptera, Pyrrhocoridae). European Journal of Protistology. 57, 85-98;
- Johannes Sikorski, Alla Lapidus, Olga Chertkov, Susan Lucas, Alex Copeland, et. al.. (2010). Complete genome sequence of Acetohalobium arabaticum type strain (Z-7288T). Stand. Genomic Sci.. 3, 57-65.