
Игра в «поймай промотор»
08 апреля 2024
Игра в «поймай промотор»
- 757
- 0
- 6
Поиск промоторов — это сложная игра для биоинформатиков © DALL-E 3.
Рисунок в полном размере.
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: ДНК — не такая уж скучная и «правильная» молекула, как принято считать. На ней уживаются вместе очень непохожие последовательности — и кодирующие, и регуляторные, и повторяющиеся, и мобильные элементы генома, и невесть еще какие. Те, что не служат матрицей для синтеза белка и/или РНК, могут, тем не менее, в нем активно участвовать в другом качестве — как в случае промоторов, на которых начинается транскрипция ДНК. Предсказание положения промоторов в пока неизученных геномах имеет огромное значение для аннотации геномов и биотехнологии. И это — один из самых старых, можно сказать, «проклятых вопросов» биоинформатики, который все еще далек от окончательного решения. Как так вышло и возможно ли в принципе точно «вылавливать» промоторные последовательности ДНК — в том числе из неведомых геномов, которые нам поставляет секвенирование нового поколения?
Конкурс «Био/Мол/Текст»-2023/2024
Эта работа опубликована в номинации «Своя работа» конкурса «Био/Мол/Текст»-2023/2024.

Генеральный партнер конкурса — международная инновационная биотехнологическая компания BIOCAD.
Партнер номинации — компания «Хеликон».

«Книжный» спонсор конкурса — «Альпина нон-фикшн».
Что в промоторе привлекает полимеразу?
ДНК часто определяют как молекулу, в которой хранится информация для синтеза белка. Это, безусловно, правда, однако не вся — можно сказать, что далеко не вся. В действительности у, скажем, человека доля кодирующих областей составляет всего 1–2% генома — именно они служат матрицей для синтеза РНК и, как правило, еще и белков. При этом почти половина генома Homo sapiens занята различными повторами ДНК и мобильными генетическими элементами.
Для ядерных организмов в целом характерна малая доля кодирующей ДНК, а вот у прокариот кодирующих участков в геноме куда больше. В среднем, ДНК бактерий состоит из них на 88%, хотя у отдельных представителей — до 97%. Это отражает глубокие различия в организации геномов эу- и прокариот, касающиеся и их регуляции [1].
От кодирующих областей с бесценной информацией о структуре белков не будет проку, если их не «переписать» и не «перевести». Именно так переводятся названия двух ключевых этапов экспрессии генов — транскрипции и трансляции. И если трансляция — это задача мРНК и рибосом (тоже в основе своей РНК с ферментативной активностью), то в транскрипции должна принимать участие сама ДНК. Для этого по ней разбросаны регуляторные элементы — промоторы, терминаторы, энхансеры, сайленсеры, инсуляторы и т.д. Это кусочки ровно той же молекулы-двойной спирали, — полимера из 4 нуклеотидов, — что и кодирующие области, но функционируют они принципиально иначе [2].
Все начинается с промотора — последовательности, который узнает и связывает ДНК-зависимая РНК-полимераза (далее будем называть ее просто «полимераза»). Это чрезвычайно важный фермент, а точнее, целое множество разных ферментов, которые синтезируют молекулы РНК, используя кодирующую ДНК в качестве матрицы [3].
Значимые различия между прокариотами и эукариотами касаются и пары «промотор—полимераза», которые в полном смысле созданы друг для друга эволюцией. У самых сложных и крупных клеток эукариот имеется несколько типов таких ферментов: из них самая ходовая — это РНК-полимераза II, тогда как РНК-полимеразы I и III — это «ферменты специального назначения». Вторая полимераза довольно громоздка: состоит из 12 субъединиц при молекулярной массе примерно 550 килодальтон. А еще для нормальной работы ей необходим длинный список транскрипционных факторов.
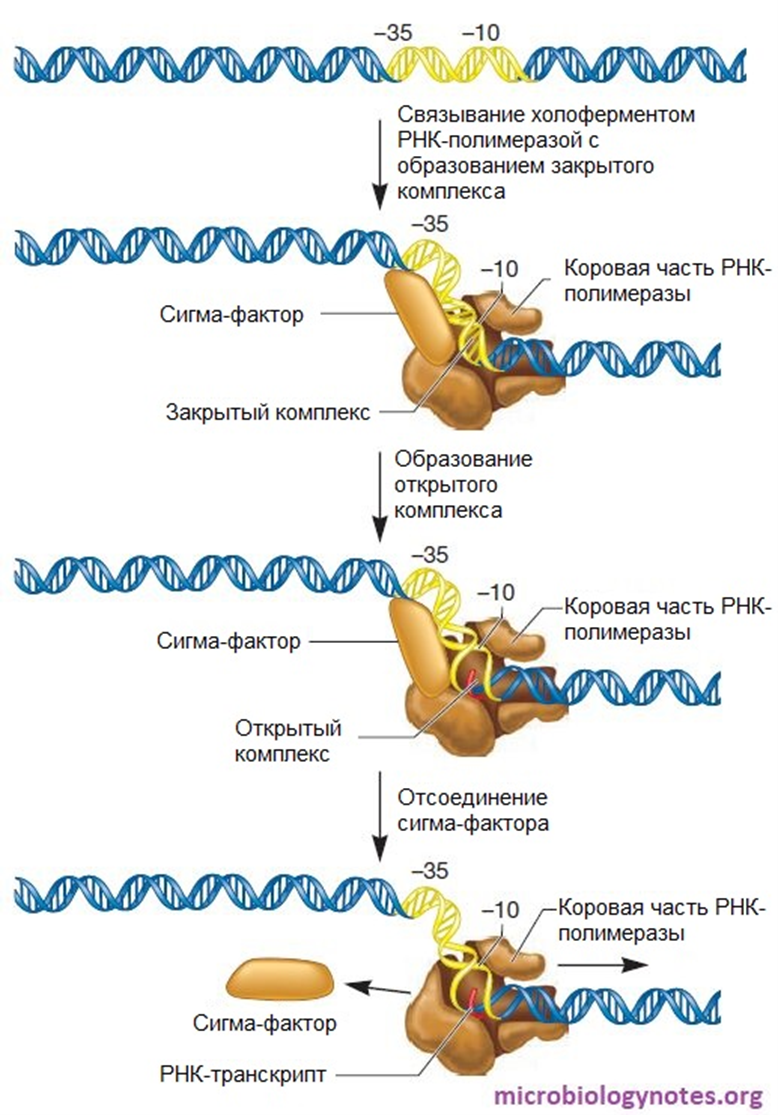
Рисунок 1. Этапы инициации транскрипции у прокариот и участие в них сигма-фактора.
С полимеразами бактерий — скажем, всеобщей любимицы Escherichia coli, — все попроще, поэтому основы промоторно-полимеразного узнавания проще изучать на них. У каждой бактерии имеется единственный фермент, который «весит» примерно столько же, сколько РНК-полимераза II эукариот — 480 килодальтон. Бактериальная РНК-полимераза состоит всего из пяти стандартных субъединиц и еще одной, скажем так, специальной. Работу единственной бактериальной полимеразы нужно как-то регулировать — за это как раз и отвечает шестая субъединица. Речь о сигма-факторах, которых у E. coli семь — они способны присоединяться и отсоединяться от ферментного комплекса (рис. 1). В фазе активного роста у E. coli работает сигма-70 — фактор «домашнего хозяйства», наиболее важная из сигма-субъединиц, названная так из-за молекулярного веса около 70 килодальтон. Прочие сигма-факторы присоединяются к полимеразе в особых случаях. К примеру, сигма-24 работает при стрессе, вызванном высокой температурой среды.
В соответствии с этим и промоторы кишечной палочки делятся на сигма-70, сигма-24 специфичные и так далее. Они избирательно связываются с бактериальной полимеразой, если та имеет нужный сигма-фактор, и помогают клетке оперативно реагировать на изменения — окружающей и внутренней среды. Получается, сигма-факторы работают как сменные насадки у отвертки — плоская, крестообразная, шестигранник и т.д. — и определяют специфичность РНК-полимеразы к промоторам на ДНК.
Для такой регулируемости и специфичности требуется молекулярное распознавание, т.е. нековалентные, организованные в пространстве взаимодействия молекул, которые происходят за счет специфических контактов. Конкретные участки сигма-субъединицы взаимодействуют с конкретными участками промоторной области и, если они друг другу подойдут, молекулы связывается и запускают инициацию — первый этап транскрипции.
Молекулярное распознавание промотора полимеразой — это деликатный многостадийный процесс. Вначале две молекулы сближаются, начинают взаимодействовать и образуют комплекс. Тот изменяет свою форму, сперва оставаясь закрытым, а затем ДНК в его составе плавится, т.е. «открывается» — происходит переход к открытому комплексу. Тот далее выпускает сделавший свое дело сигма-фактор, и на смену инициации приходит элонгация, а затем и терминация транскрипции. В результате мы имеем свежесинтезированную РНК [4], [5].
Плавление ДНК (открытие дуплекса) — это, разумеется, не то же самое, что плавление сосульки или слитка золота. В данном случае фазовый переход (плавление) означает, что цепочки двойной спирали расходятся и водородные связи между ними рвутся. Фрагмент ДНК считают расплавленным, если фазовый переход прошли половина его пар оснований [6], [7].
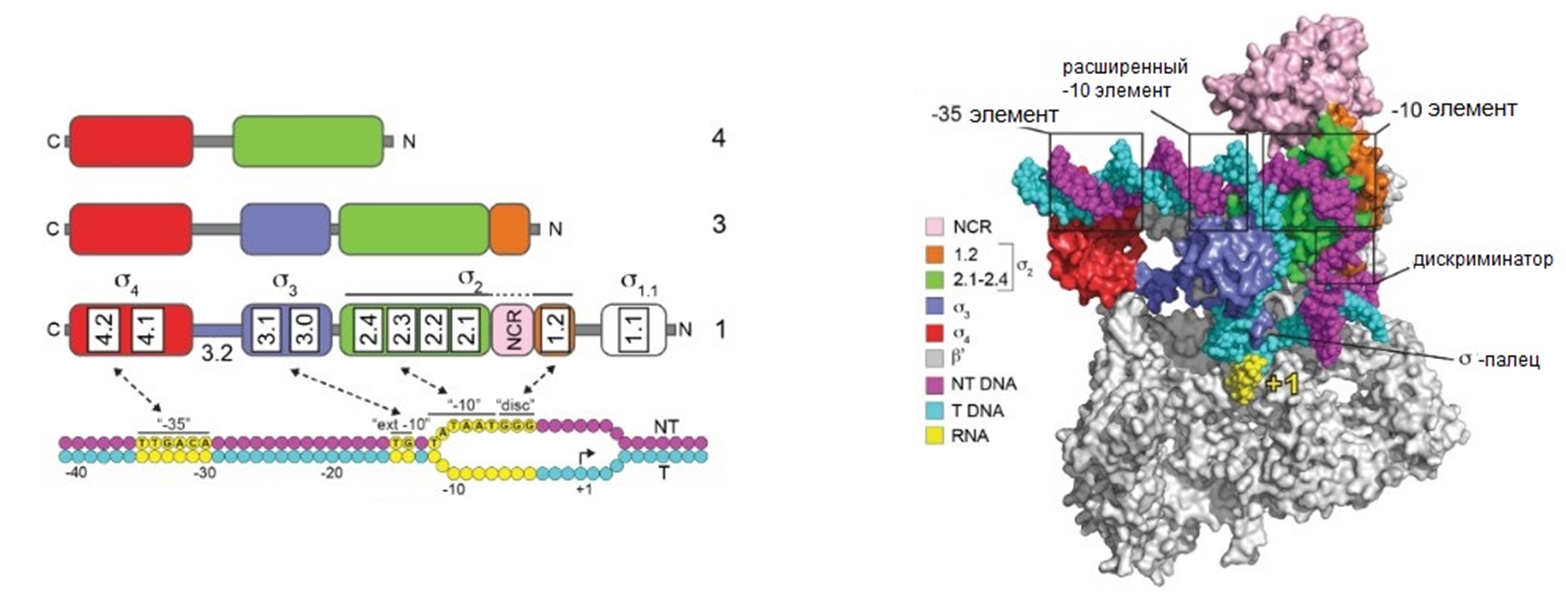
Рисунок 2. Функциональные области бактериального промотора и его связывание с РНК-полимеразой E. coli.
Какова роль промотора во всей этой хореографии? Чтобы процесс вообще начался, промоторная область должна иметь определенную структуру и вписываться в канавку РНК-полимеразы. Разумеется, речь прежде всего о первичной структуре ДНК, т.е. нуклеотидной последовательности. Действительно, все промоторы — будь то бактериальные, архейные или эукариотные — роднит наличие последовательности TATA. В случае бактерий это Прибнов-бокс («ящик Прибнова», по имени первооткрывателя Дэвида Прибнова) из шести нуклеотидов — TATAAT, отстоящий от точки старта транскрипции (ТСТ) на 10 «букв». Отсюда его другое название — −10 элемент (рис. 2). У архей и эукариот TATA-бокс имеет гексамерную последовательность TATAAA и расположен от ТСТ на 30 нуклеотидов левее, т.е. «выше по течению» ДНК. Область TATA в том или ином виде связывает РНК-полимеразу — элемент имеет центральное значение в узнавании друг другом двух молекул. Но только областью TATA дело не ограничивается [4], [7].
В среднем на 17 нуклеотидов левее −10 элемента находится −35 элемент, характерный мотив которого (TTGACA) тоже участвует в связывании полимеразы. Но промоторы не слишком требовательны к своей последовательности: для активности им совершенно необязательно, чтобы все буквы −10 и −35 элементов были по местам. Более того, «идеальные» синтетические промоторы с точной консенсусной последовательностью в эксперименте показали себя довольно плохо. Отклонения от консенсусных мотивов встречаются у многих активных промоторов, так что эти паттерны носят не обязательный, а скорее статистический характер [8–10].
Для описания таких нестрогих закономерностей в биомолекулах используют позиционные весовые матрицы (PWM). Их строят на основе выровненных последовательностей — расположенных друг под другом так, чтобы соответствующие области оказались напротив. PWM помогают выявить мотивы — паттерны последовательности (в данном случае нуклеотидной), которые эволюционно консервативны и связаны с какими-то биологическими функциями. Их изображают в виде логотипа — диаграммы, на которой относительные размеры букв в каждом положении указывают на частоту их встречаемости. Суммарная высота букв отражает информационное содержание нуклеотидного положения в битах (рис. 3) [11].
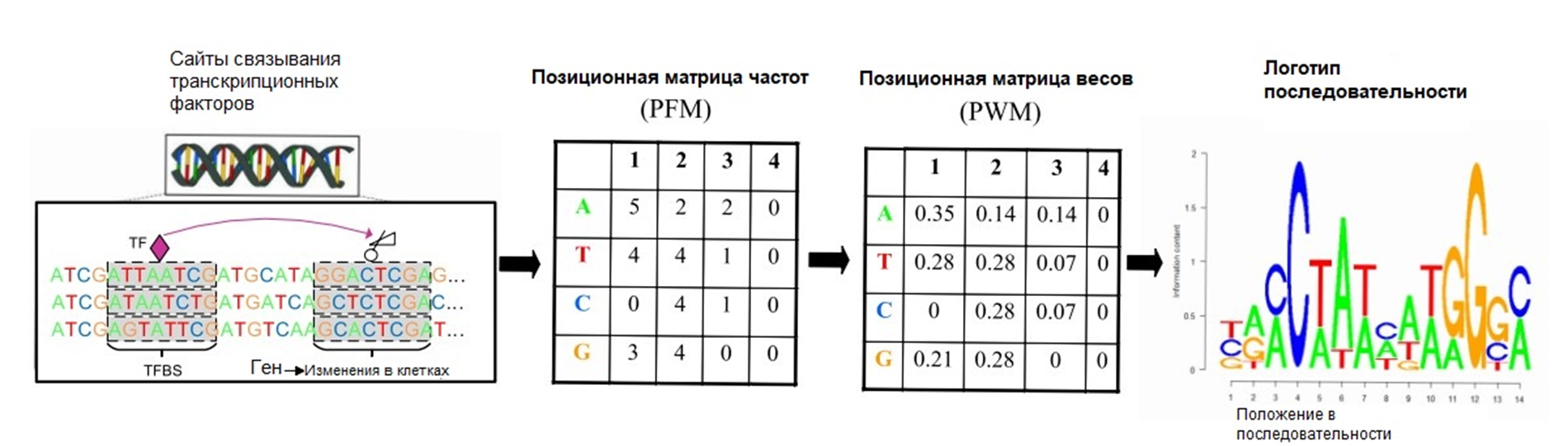
Рисунок 3. Построение позиционной весовой матрицы и логотипа последовательности.
Далее, более активные промоторы бактерий могут дополнительно иметь UP-элемент (AT-богатый upstream-элемент), расположенный еще левее −35-элемента. Он тоже описывается консенсусным мотивом из шести нуклеотидов и помогает связать РНК-полимеразу (рис. 2).
Не только строгие мотивы последовательности, но и фиксированное расстояние между регуляторными элементами и даже само их наличие не обязательны для активного промотора. Одни из них могут порой успешно заменяют другие — например, «расширенный» −10 элемент делает активным промотор, у которых −35 элемент отсутствует напрочь.
Алгоритмы-предсказатели
Так мы незаметно для себя вторглись в область биоинформатики — науки, использующей строгий математический аппарат и вычислительные возможности компьютера для анализа биологических данных. Последовательности биомолекул — нуклеиновых кислот и белков, своеобразные «тексты» или «строки» (strings) — это классический объект биоинформатических исследований.
Сейчас вычислительные биологи скрупулезно анализируют последовательности тысяч промоторов, связывающих факторы транскрипции участков ДНК (энхансеров) и т.д. Дело в том, что невиданный прогресс методов секвенирования (NGS, next generation sequencing) порождает поток последовательностей нуклеиновых кислот, в том числе совсем не изученных геномов. Отыскать промоторы и другие регуляторные элементы в них в ходе геномной аннотации — очень сложная, но и важная задача. Иначе не получится понять, как работают эти геномы, и использовать их в биомедицине и биотехнологии [12].
Как и в других подобных случаях, для «борьбы» с Big data используют машинное обучение (machine learning, ML). Это метод обработки данных, который использует не прямое решение, а обучение алгоритмов в ходе решения множества сходных задач. Известны три главных типа ML, которые отличаются тем, какие данные они используют (рис. 4).
Первый — обучение с учителем (supervised machine learning), которое исходно нуждается в размеченных данных. Это означает, что модели учатся на части общего датасета, для которых нам известны значения входных и выходных данных («правильные ответы»). Скажем, наши данные — это ряд свойств последовательности ДНК, а выходные — ее заданная (размеченная) принадлежность к промоторам, заведомым «непромоторам», промоторам конкретной группы и т.д. Такое ML попроще, и именно оно пригодится нам позже.
Второй тип машинного обучения использует не размеченные данные «как они есть» и не располагает даже небольшой шпаргалкой из правильных ответов. Это уже машинное обучение без учителя (unsupervised machine learning). Характерный пример таких методик — это кластерный анализ, который подробно описан в другой статье [12]. Наконец, выделяют также обучение с подкреплением (reinforcement learning), в ходе которого испытуемая система (агент) обучается, взаимодействуя с динамичной средой. Представляет собой частный случай обучения с учителем, однако учителем здесь является среда или ее модель. Такие методы тоже довольно популярны, так как имеют некоторые уникальные преимущества [13].
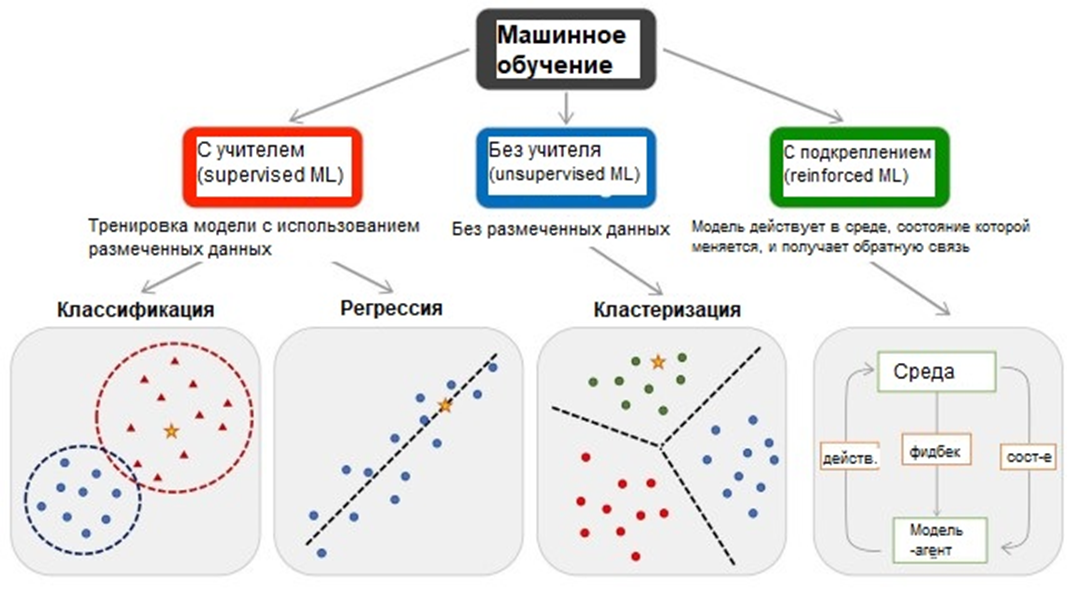
Рисунок 4. Типы машинного обучения с примерами.
Как работает машинное обучение, если говорить кратко? В случае обучения без учителя ML, по сути, просто «скармливают» исходные данные, возможно, после какой-то очистки или предобработки (например, нормировки). Кластерный анализ при этом выделяет в многомерных (т.е. охарактеризованных многими переменными) нетривиальные группы, которые могут, скажем, соответствовать определенным биологическим функциям. Помимо кластеризации и ей подобных, существуют методы понижения размерности вроде PCA (метода главных компонент) или t-SNE — набирающей популярность у ученых новинки со звучным переводом: «стохастическое внедрение соседей с t-распределением». Они как бы проецируют многомерное облако данных в пространство с меньшим числом измерений, более понятное для трехмерного человека [13].
Но сейчас нас интересует supervised ML — обучение с учителем, которое больше всего подходит для поиска промоторов. Мы помним, что ему требуются размеченные данные — так называемая обучающая выборка. В нашем случае это может быть набор из заведомо известных промоторов и каких-то «не-промоторов» — последовательностей, которые меньше всего на них похожи, или просто случайных кусков генома. С этим загвоздка, ведь инициировать транскрипцию РНК-полимераза, по сути, может с любого участка ДНК, только с разным успехом.
Вначале алгоритмы тренируют на размеченной «делянке данных», после чего им предлагают уже настоящую задачу — тестовую выборку. Исходя из своих соображений алгоритмы распределяют ее по заданным категориям (так называемая классификация) или приписывают им какие-либо численные значения (регрессия).
В случае наших промоторов классификаторы могут «отделить зерна от плевел» — т.е. предполагаемые промоторы от прочих последовательностей. Или, скажем, распределить промоторы на группы с разными функциями и свойствами. В самом простом случае используют бинарные классификаторы, которые умеют раскладывать одну «кучу» объектов на две. Алгоритмы регрессии способны предсказать силу промотора, имеющую численный вид. Сила промотора отнюдь не равна массе промотора, умноженной на его ускорение — она показывает, насколько активно в данных условиях на нем инициируется транскрипции. Ее можно определять по-разному — например, как скорость покидания промотора комплексами элонгации, т.е. его освобождения для нового «визита» РНК-полимеразы [14].
На входе алгоритмы получают данные — набор последовательностей, для которых известны различные характеристики, по которым мы хотим отличить промоторы от прочих участков ДНК. Для некоторых из них (обучающая выборка) также задана принадлежность к группе. Какого рода это характеристики? Чаще всего используют различные позиционные весовые матрицы (PWM) упомянутых регуляторных элементов (-10, −35, UP-element), расстояние между ними, частоту встречаемости в последовательности k-меров (слов длин 3, 4, 5, 6 нуклеотидов и более) и т.д. Самый очевидный признак, характеризующий нуклеотидный состав ДНК — содержание пар GC, которых в промоторах заведомо мало (а АТ — много), что нетрудно заметить при беглом взгляде на все эти консенсусные последовательности из шести нуклеотидов. Можно показать изобретательность и вычислить, скажем, GC-skew нашей ДНК (обогащение последовательности гуанином и цитозином) или даже получить загадочную Z-кривую для нее [15], [16].
На выходе мы обычно имеем набор предсказанных промоторов и сами модели, которым также есть о чем рассказать — ведь они содержат в себе информацию о вкладе отдельных переменных в свою работу. Но первым делом нам нужно оценить их качество (performance): таких метрик довольно много, самые простые — чувствительность (sensitivity), специфичность (specificity) и точность (accuracy) [16]. Все три лучше использовать вместе, чтобы избежать ошибочных или пристрастных интерпретаций — например, когда чувствительностью очень велика, специфичность неприлично низкая, но точность выходит солидной (рис. 5).

Рисунок 5. Метрики качества моделей машинного обучения.
рисунок автора, использованы фотографии с фотостоков
При работе классификатора возможно четыре исхода: верное попадание, т.е. промотор назван промотором (true positive); то же — в случае верно названного не-промотора (true negative); ошибка первого рода — ошибочно предсказан промотор (false positive); ошибка второго рода — промотор не назван промотором (false negative).
На основе этих четырех чисел, хранящихся в матрице два на два (матрица ошибок, или confusion matrix), и вычисляют метрики. Самая важная из них — это accuracy (точность), которая у отдельных промоторов может достигать чуть ли не 100 %. [23,17] Однако слабое место таких алгоритмов — это sensitivity (чувствительность): здесь получить 90% — очень хороший результат.
В чем же дело? Биоинформатики связывают это с рядом причин, из-за которых «поверить алгеброй» своенравные промоторы оказалось так сложно. В частности, они попросту слишком сильно различаются и работают на основе разных механизмов. Их активность сильно зависит от условий среды (в том числе в экспериментах in vitro) или состояния клетки. Более того, ТСТ зачастую любят «толпиться кучей», из-за чего накладываются друг на друга при предсказании. Далее, используемые биоинформатиками «мокрые» данные о промоторной активности сами далеки от идеала — и их сложно сделать идеальными и унифицированными. Датасеты биоинформатиков тоже слишком разношерстны и относятся к различным объектам исследования. Неплохо бы создать какой-то общепринятый эталон — стандартный набор данных (benchmark) о промоторах и других последовательностях ДНК [15–17]. Может, такой датасет стоит отправить в Парижскую палату мер и весов...
Чтобы добиться лучших предсказаний, можно, с одной стороны, совершенствовать «информатическую» сторону этого вопроса биоинформатики. Скажем, оптимизировать исходные данные, выбирая нужные переменные или даже создавая на их основе новые, использовать ансамблевые предсказания, которые разные алгоритмы делают вместе и т.д. — это тема для отдельного вдумчивого разговора. Однако стоит сосредоточиться на том, что не так с биологической стороной вопроса.
Промотор во плоти
Один из самых перспективных путей решить «проклятый промоторный вопрос» — это переключить внимание на новые предикторы, т.е. свойства, на основании которых предсказывают тип последовательности. Иными словами, отойти от проблемы на два шага назад и сформулировать ее заново. Есть ли вообще пресловутой ДНК-зависимой РНК-полимеразе дело до конкретных «букв» в нуклеотидной последовательности промотора, которые мы пытаемся анализировать? Разумеется, ферменту нужны специфические контакты, обеспечивающие молекулярное распознавание ДНК — вот только их вполне может обеспечить разный набор нуклеотидов. Или не обеспечить (очень плохо обеспечить) последовательность, которая очень похожа или даже идентична ДНК с сильной промоторной функцией. Отсюда берется и довольно пренебрежительное отношение полимеразы к мотивам промоторной ДНК [15], [18].
Безусловно, промотор хранит в себе информацию, необходимую для «привередливого» выбора молекулой партнера для связывания — иначе никакого специфического узнавания не выйдет. Но если матричная информация напрямую записана в «тексте» последовательности ДНК, то молекулярные взаимодействия, — в том числе сложный и многостадийный процесс инициации транскрипции — им не ограничивается. Такое кодирование информации последовательностью ДНК называют «непрямое» (indirect readout, непрямое узнавание). Оно использует различные физико-химические и структурные свойств ДНК, которые важны для ее взаимодействия с другими молекулами [19]. Можно сказать, что есть по меньшей мере два разных способа закодировать информацию в одной и той же ДНК — они напоминают стандартный способ (текст) и ASCII-графику, в основе которой — рисование буквами, другими знаками и словами.
На рисунке 6 в качестве примера приведено одновременно написанное на английском языке стихотворение «Ворон» Эдгара Аллана По и не требующий знания никакого языка для понимания силуэт птицы. Таким образом, изображение передает информацию на двух разных уровнях и с помощью разных принципов кодирования.

Рисунок 6. Стихотворение Эдгара Аллана По «Ворон» в ASCII-представлении.
Так вот, РНК-полимераза, как и другие ДНК-связывающие ферменты, «смотрит» промотору скорее не в текст, а на более «вещественные» характеристики. Можно также сказать, что он ДНК не «читает», а скорее «щупает» или «ощущает». Безусловно, физико-химические параметры тоже определяются последовательностью ДНК, но зависят от нее не напрямую и сложным образом — скажем, могут определяться широким контекстом и быть очень чувствительными к малым (даже точечным) изменениям первичной структуры. Более того, большая или меньшая роль отдельных физических свойств зависит от биологической роли промотора, его принадлежности к какой-то группе, а также варьирует на разных этапах инициации транскрипции [15], [18].
Так, на ранних стадиях взаимодействия промотора и РНК-полимеразы (рис. 1) особое значение имеет «дальнобойная» характеристика ДНК — электростатический потенциал [20], [21]. Напомним: из-за бесчисленных остатков фосфатов ДНК — это полианион и природная молекула с самой большой плотностью отрицательного заряда [22]. Оказалось, что промоторы даже более отрицательно заряжены, чем ДНК в среднем [23], а РНК-полимераза несет особые положительно заряженные участки для их связывания (рис. 7) [21]. На следующих этапах инициации преобладает уже, например, способность ДНК менять форму и изгибаться при связывании белков — она помогает комплексу изменить форму и удержать фермент на промоторе [24]. При переходе к открытому комплексу существенно, насколько легко промотор плавится и как успешно образовавшийся «пузырь» (открытое состояние ДНК, DNA bubble) может двигаться по двойной спирали (рис. 8). Естественно предположить, что эти свойства способствуют большей промоторной активности [25], [26].
Ранее при изучении прокариотических промоторов (в том числе в наших статьях) удалось описать ряд интересных закономерностей их физико-химических свойств. Например, характерные электростатические профили промоторов E. coli, различия в электростатике между группами промоторов паразита кишечной палочки — бактериофага T7, особые профили, характеризующие склонность промоторов к изгибанию, их отличия от прочей ДНК по значениям свободной энергии, стекинг-взаимодействиям, энтропии, температуре плавления, вызванной суперспиральностью дестабилизации дуплекса (SIDD) и т.д. [6], [16], [24], [27–30].
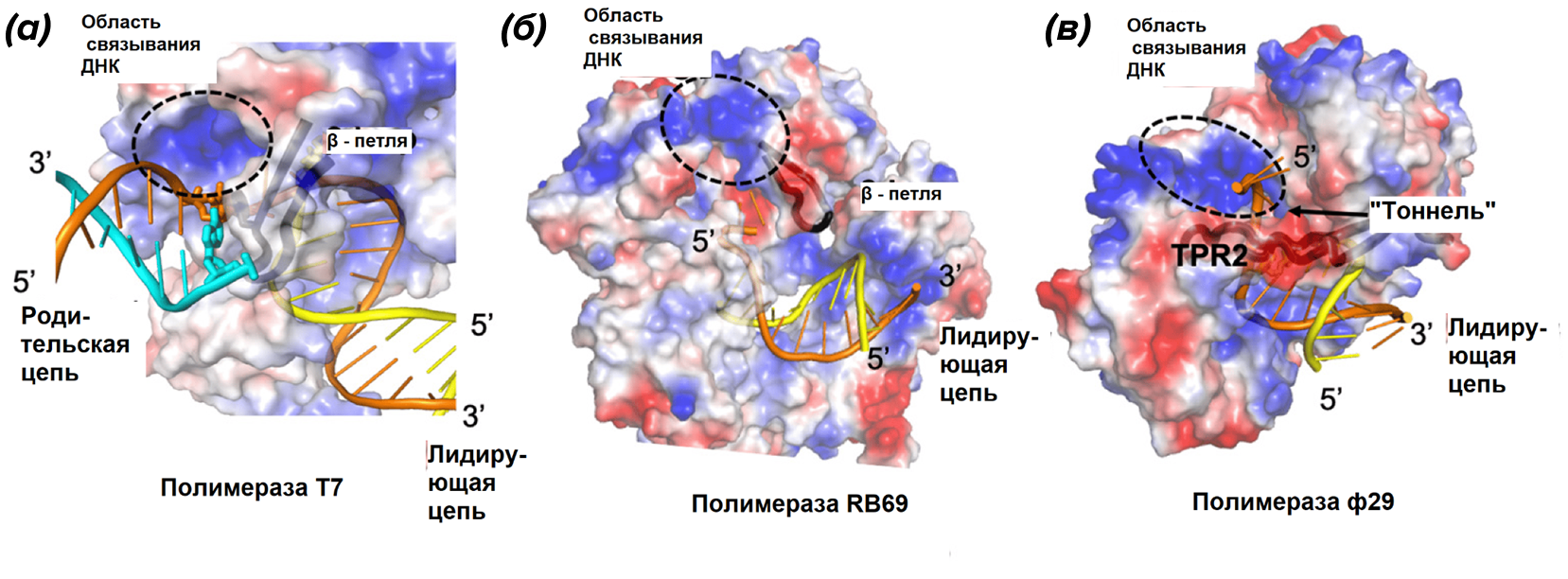
Рисунок 7. Полимеразы бактериофагов T7 (a), RB69 (б) и Φ29 (в) в комплексе с ДНК. Цветом обозначена величина электростатического заряда на поверхности фермента (красным — более отрицательный, синим — более положительный).
Разумеется, ученые сразу попытались построить модели, которые предсказывают промоторные последовательности на основании отдельных физико-химических свойств [23], [24] и сочетания таких характеристик друг с другом, а также с «текстовыми» свойствами [16]. При этом использование непохожих предикторов помогает «выловить» промоторы разных функциональных групп и те, которые используют для своей работы отличную регуляцию.
Именно этим принципом руководствовались мы в вышедшей некоторое время назад статье [15]. Мы использовали базу данных RegulonDB (версия 8.5), содержащую огромную информацию о регуляции транскрипции E. coli, и получили свойства 699 ее промоторных последовательностей. Это только те промоторы, что были подтверждены в «сыром» эксперименте, так что в их активности сомневаться не приходится. Также для генома E. coli получены последовательности других типов:
- 3427 кодирующих областей генов;
- 1880 «не-промоторов» — так мы обозначили случайные участки ДНК, отстоящие от известных ТСТ на 300 нуклеотидов и более, которые при этом не имеют какой-либо известной функции в геноме;
- 2000 lowscore-последовательностей — согласно анализу с помощью программы для предсказания промоторов PlatProm, по своей последовательности меньше всего похожи на промоторы;
- наконец, 2228 промоторных островков (promoter islands). Это открытые с помощью биоинформатики последовательности бактериальных геномов, которые содержат множество предсказанных ТСТ, хорошо связывают РНК-полимеразу, но не имеют промоторной активности [31].
Для этого разношерстного набора участков ДНК, которые очень похожи на промоторы, очень от них отличаются, являются чем-то средним или заведомо сами и есть промоторы, мы рассчитали различные характеристики. Это электростатический потенциал, ряд динамических характеристик «ДНК-пузырей» или открытых состояний (энергия активации, размер, скорость звука в них), а также GC-состав. Все профили получили на отрезке [-150;50] относительно ТСТ (или произвольной точки, если это не промоторов), что примерно соответствует границам бактериального промотора — вместе с контекстом, который также важно учитывать.
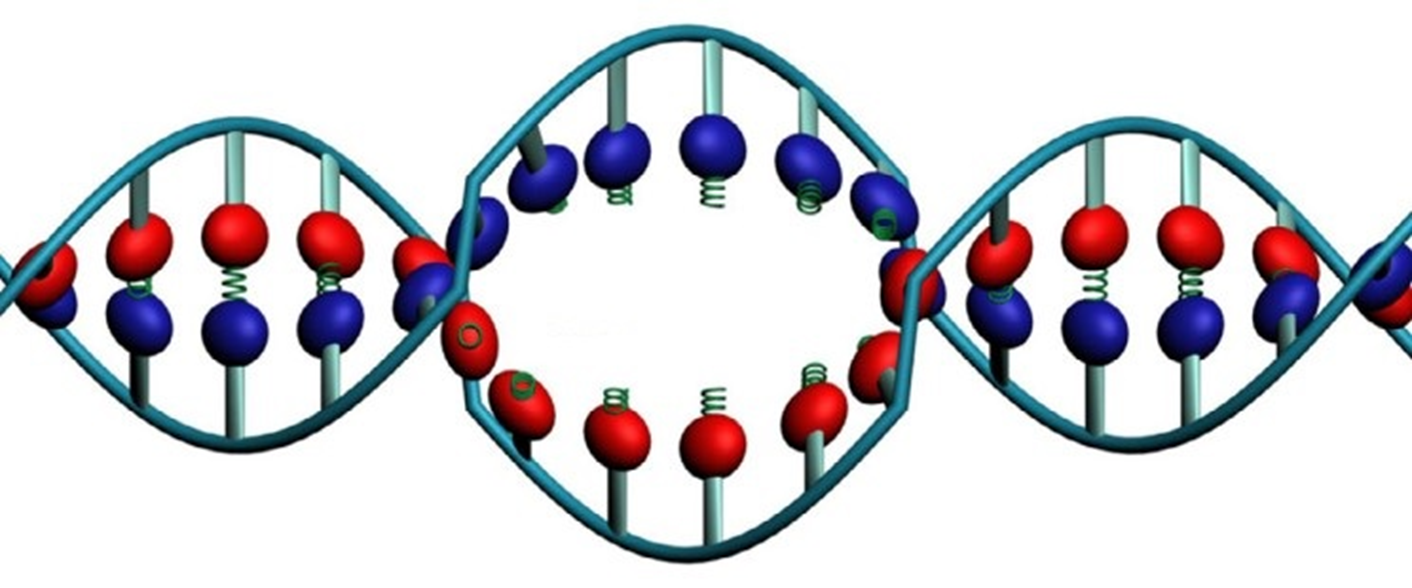
Рисунок 8. «ДНК-пузырь» (открытое состояние дуплекса).
Далее мы провели оптимизацию нашего датасета, выделив в нем избыточные предикторы. В этом нам помог иерархический кластерный анализ, который создал дендрограммы для разных характеристик — мы сравнили их между собой несколькими способами. Такой разведочный анализ данных показал, что используемые предикторы в действительности очень разные, не коррелируют между собой (что снизило бы качество моделей), так что далее мы использовали почти все переменные, удалив только одну характеристику открытых состояний ДНК.
Затем мы объединили векторы (наборы чисел, характеризующие определенные свойства последовательности) в матрицы — прямоугольные таблицы. К ним применили анализ главных компонент (PCA), который проецирует многомерное облако данных в новое пространство с другими, менее многочисленными и более информативными осями. В этом случае для сохранения большей части статистической дисперсии данных достаточно оставить небольшой их объем. Скажем, в нашем случае первые 50 главных компонент содержат в себе 97,5% дисперсии всего исходного датасета.
Поэтому в дальнейшем для тренировки алгоритмов мы использовали сами эти главные компоненты — 50, 100 или 150 штук. Так мы получили новые предикторы, которые намного меньше исходных переменных по размеру и к тому же «очищены» от значительной части статистического шума.
Полученный сильно «похудевший» датасет мы разделили на четыре части, которые описывают попарно по два типа последовательностей: промоторы—гены (G-P), промоторы-промоторные островки (I-P), промоторы—lowscore-последовательности (L-P) и промоторы—непромоторные последовательности (N-P). Такое деление позволяет лучше понять, что в каждом случае важнее для предсказания промоторов. На этом наборе данных тренировали бинарные классификаторы, использующие алгоритмы машинного обучения Naive Bayes и Random Forest, которые довольно сильно отличаются друг от друга по ряду признаков. В каждом случае обучающую выборку получали случайным образом — в нее попадало равное количество последовательностей двух типов и 70%, 80% или 90% общего набора данных, соответственно. Получение повторных выборок (ресемплинг) выполняли по 10 раз, перекрестную проверку (кросс-валидация) повторяли трижды — так мы получили множество разных моделей и снизили эффекты от ошибок отдельных.
Натренированные модели проверяли на тестовых выборках, содержащих 30, 20 или 10% от исходного набора данных. При этом обучающие и тестовые выборки не пересекались. Наконец, для каждого набора данных, алгоритма обучения, пропорции обучающей и тестовой выборки и количества главных компонент было обучено по 10 классификационных моделей — в итоге общее их количество составило 720.
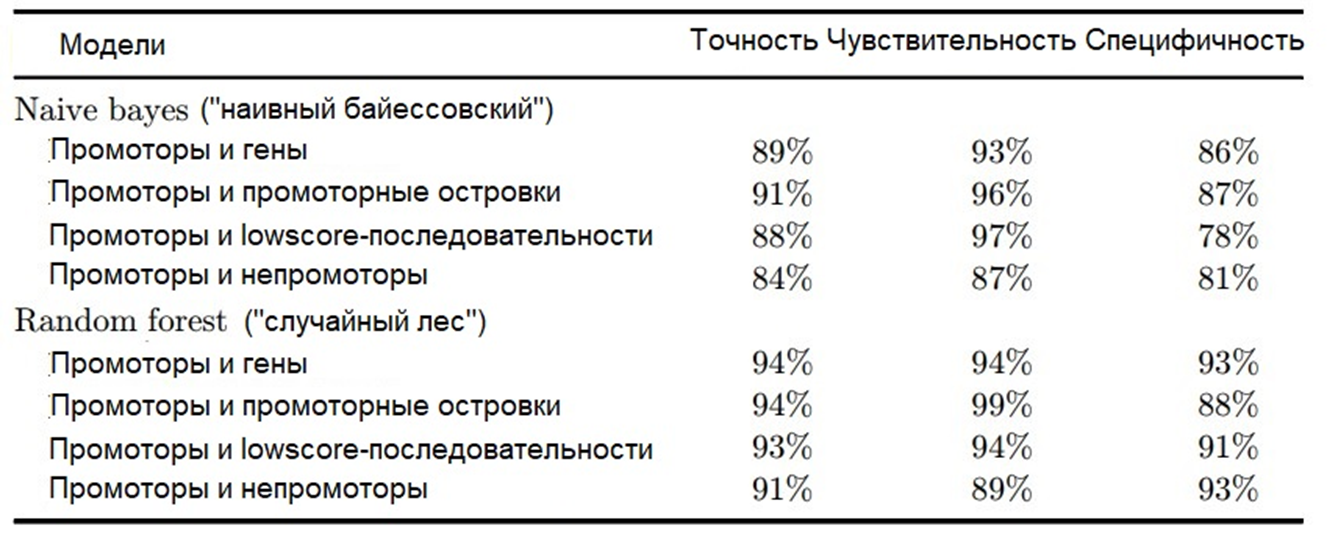
Рисунок 9. Средние показатели качества полученных классификаторов.
После этого мы приступили к оценке качества полученных моделей (рис. 9, 10). Довольно закономерно, что более сложные алгоритмы Random Forest превзошли алгоритмы Naive Bayes по стандартным показателям, например, их точность составила 91–94% и 84–91%, соответственно. Значения чувствительности были очень близки (89–99% и 87–97%), тогда как специфичность опять-таки выше у Random Forest, чем у Naive Bayes — 88–93% против 78–86%. В целом, количество правильно классифицированных промоторов у моделей первой группы оказалось больше.
Такие результаты можно назвать хорошими и сравнимыми с качеством современных моделей с лучшими показателями. Например, CNNProm, чувствительность и точность которого при поиске промоторов E. coli его авторы оценили как 90% и 96%, соответственно [17].
Стоит отметить, что в случае моделей Naive Bayes чувствительность нарастает с увеличением количества главных компонент, которые мы оставляем для машинного обучения. А вот их специфичность, наоборот, при этом снижается. Классификаторы Random Forest оказались в этом отношении более стабильны — они меньше зависят от различий во входных данных, если судить по их показателям качества.
Как ни странно, самые высокие значения точности были получены для классификаторов, отделяющих промоторы от промоторных островов. Т.е. последовательностей, которые по текстовым характеристикам по определению сильнее всего похожи на промоторы. Выходит, их физико-химические свойства (которые как раз определяют промоторную функцию) сильно различаются. В дальнейшем это может помочь отличать друг от друга промоторы и островки, которые обычные алгоритмы путают [15].
Подводя итог, стоит сказать, что мы учли основные тренды, характерные работы по предсказанию промоторов в последнее время. Так, мы отошли от анализа изолированных промоторных сигналов только в той области ДНК, которая напрямую связывает РНК-полимеразу. Этот подход обычно рассматривает лишь несколько десятков пар оснований и явно игнорирует влияние фланкирующих областей, как и сложность и разнообразие механизмов инициации транскрипции. Мы также не используем k-меры — популярную «валовую» характеристику последовательности ДНК, которая ищет «биологические строки» в ДНК вне зависимости от их положения относительно ТСТ. Это игнорирует структуру промотора, который состоит из неравнозначных областей [17].
Рассматривая широкий «генетический контекст» промоторов и используя сложные методы анализа (вроде глубокого обучения и нейронных сетей), биоинформатики могут лучше описать структурное и функциональное разнообразие промоторов. Более того, так мы можем в неявном виде учесть физические характеристики ДНК, которые косвенно закодированы в ее последовательности. Увы, на фоне хороших показателей качества моделей это затрудняет их интерпретацию — понимание того, чем руководствовались алгоритмы и что именно делает тот или иной промотор в реальной жизни промотирующим.
В случае нашей работы прозрачно, какие именно физические характеристики ДНК важнее для промоторов в целом либо какой-то их специализированной группы. При этом мы используем как можно более различные признаки ДНК, не ограничиваясь только коррелирующими структурными [17].
Разумеется, ускользающие промоторы мы по-прежнему вылавливаем недостаточно качественно. Возможно, в будущем стоит попробовать решать задачу предсказания промоторных ДНК поэтапно. Скажем, для начала отсеивать те из них, которые легко плавятся при посадке белков из-за суперспирального стресса — его характеризует так называемый SIDD (stress-induced duplex destabilization) [25]. Для значительной части промоторов этот признак не важен, тогда как другие явно используют высокую дестабилизацию [6]. Хорошо бы также создавать такие методики, которые применимы не только к отдельным геномам и учитывают самые универсальные признаки промоторных ДНК [17].
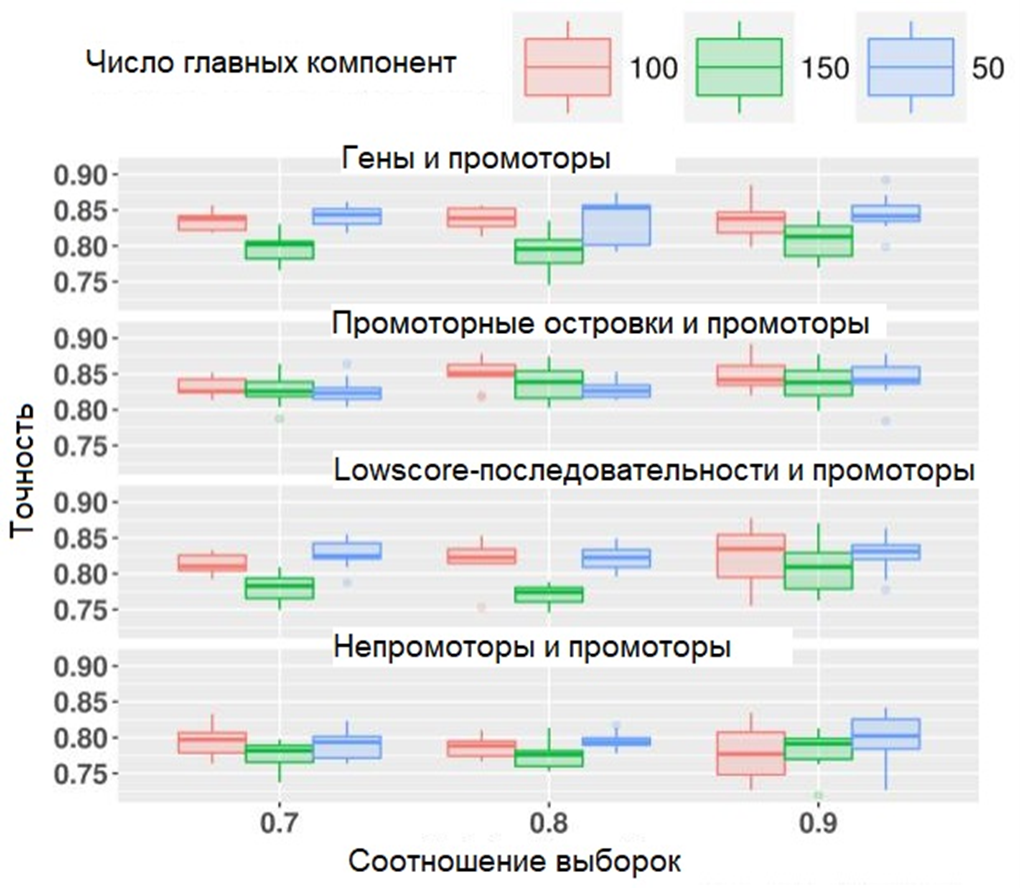
Рисунок 10. Значения точности (accuracy) полученных классификаторов.
Эпилог. О пользе моделей с ошибками
Подводя итог, хочется сказать что-то оптимистичное или хотя бы философское. Наш подход, безусловно, имеет перспективы — как и некоторые другие пути решения «проклятого вопроса биоинформатики». Однако едва ли вскоре (или вообще в будущем) появится идеальный алгоритм, который предскажет все промоторы любой бактерии со 100%-ными точностью, чувствительностью и специфичностью. Или даже 98%-ными...
Промоторы, как и многое в живом природе, слишком капризны и разнообразны для этого. Они всегда найдут, чем удивить биологов, показывая неисчерпаемое разнообразие живого на всех уровнях организации и все новые механизмы регуляции.
И все же наши и подобные алгоритмы способны помочь получить качественные предсказания промоторов определенных классов. А если их умело настроить и модифицировать — более широкую совокупность промоторных последовательностей. И это заставляет ценить наш результат, а также лишний раз вспомнить афоризм британского статистика Джорджа Фокса (который не стоит забывать тем, кто имеет дело с моделированием и биоинформатикой):
Все модели ошибочны, но некоторые, тем не менее, полезны
All models are wrong, but some are useful
Литература
- Paul C. Kirchberger, Marian L. Schmidt, Howard Ochman. (2020). The Ingenuity of Bacterial Genomes. Annu. Rev. Microbiol.. 74, 815-834;
- Второй язык ДНК;
- Али-Баба и 40 промоторов;
- Brian Bae, Andrey Feklistov, Agnieszka Lass-Napiorkowska, Robert Landick, Seth A Darst. (2015). Structure of a bacterial RNA polymerase holoenzyme open promoter complex. eLife. 4;
- William J Lane, Seth A Darst. (2006). The Structural Basis for Promoter −35 Element Recognition by the Group IV σ Factors. PLoS Biol. 4, e269;
- M. A. Orlov, A. A. Ryasik, A. A. Sorokin. (2018). Destabilization of the DNA Duplex of Actively Replicating Promoters of T7-Like Bacteriophages. Mol Biol. 52, 686-692;
- Mark Paget. (2015). Bacterial Sigma Factors and Anti-Sigma Factors: Structure, Function and Distribution. Biomolecules. 5, 1245-1265;
- Shawn T. Estrem, Tamas Gaal, Wilma Ross, Richard L. Gourse. (1998). Identification of an UP element consensus sequence for bacterial promoters. Proc. Natl. Acad. Sci. U.S.A.. 95, 9761-9766;
- Emily A Warman, Shivani S Singh, Alicia G Gubieda, David C Grainger. (2020). A non-canonical promoter element drives spurious transcription of horizontally acquired bacterial genes. Nucleic Acids Research. 48, 4891-4901;
- Yang Yang, Chang Liu, Wei Zhou, Wei Shi, Ming Chen, et. al.. (2021). Structural visualization of transcription activated by a multidrug-sensing MerR family regulator. Nat Commun. 12;
- Yuanqi Zeng, Meiqin Gong, Meng Lin, Dongrui Gao, Yongqing Zhang. (2020). A Review About Transcription Factor Binding Sites Prediction Based on Deep Learning. IEEE Access. 8, 219256-219274;
- Кластерный анализ и дилемма биологического пользователя;
- Junjie Peng, Elizabeth C. Jury, Pierre Dönnes, Coziana Ciurtin. (2021). Machine Learning Techniques for Personalised Medicine Approaches in Immune-Mediated Chronic Inflammatory Diseases: Applications and Challenges. Front. Pharmacol.. 12;
- 10.18745/th.15592;
- Artem Ryasik, Mikhail Orlov, Evgenia Zykova, Timofei Ermak, Anatoly Sorokin. (2018). Bacterial promoter prediction: Selection of dynamic and static physical properties of DNA for reliable sequence classification. J. Bioinform. Comput. Biol.. 16, 1840003;
- Ilham Ayub Shahmuradov, Rozaimi Mohamad Razali, Salim Bougouffa, Aleksandar Radovanovic, Vladimir B. Bajic. (2016). bTSSfinder: a novel tool for the prediction of promoters in cyanobacteria andEscherichia coli. Bioinformatics. btw629;
- Akhilesh Mishra, Sahil Dhanda, Priyanka Siwach, Shruti Aggarwal, B Jayaram. (2020). A novel methodSEPromfor prokaryotic promoter prediction based on DNA structure and energetics. Bioinformatics. 36, 2375-2384;
- Mikhail A. Orlov, Anatoly A. Sorokin. (2020). DNA sequence, physics, and promoter function: Analysis of high-throughput data On T7 promoter variants activity. J. Bioinform. Comput. Biol.. 18, 2040001;
- Орлов М.А. (2021). Второй язык ДНК. Природа. 9, 3–12;
- Сорокин А.А., Джелядин Т.Р., Орлов М.А., Зыкова Е.А., Камзолова С.Г. (2016). Пространственная организация электростатических взаимодействий Т7 РНК-полимеразы с поздними промоторами Т7 ДНК. Вестник биотехнологии и физико-химической биологии им. Ю.А. Овчинникова. 12, 64–71;
- Kamzolova S.G., Sorokin A.A., Dzhelyadin T.D., Beskaravainy P.M., Osypov A.A. (2005). Electrostatic potentials of E.coli genome DNA. J Biomol Struct Dyn. 23, 341–345;
- Manoj Kumar, Subhasis Haldar, Mridula Gupta, Radhey S. Gupta. (2016). DS Schottky barrier cylindrical GAA MOSFET: nanosensor for biochips. Nanomaterials and Energy. 5, 10-19;
- Темлякова Е.А. Роль электростатического потенциала ДНК в формировании промоторной функции в геноме E.coli: дис. канд. ф.-м. наук. — Пущино, 2016. — 19 с.;
- O. N. Ozoline, A. A. Deev, E. N. Trifonov. (1999). DNA Bendability—;A Novel Feature inE. coliPromoter Recognition. Journal of Biomolecular Structure and Dynamics. 16, 825-831;
- Huiquan Wang, Craig J Benham. (2006). Promoter prediction and annotation of microbial genomes based on DNA sequence and structural responses to superhelical stress. BMC Bioinformatics. 7;
- A.A. Grinevich, A.A. Ryasik, L.V. Yakushevich. (2015). Trajectories of DNA bubbles. Chaos, Solitons & Fractals. 75, 62-75;
- Орлов М.А., Камзолова С.Г., Рясик А.А., Зыкова Е.А., Сорокин А.А. (2018). Профили вызванной суперспирализацией дестабилизации дуплекса ДНК (SIDD) для промоторов бактериофага T7. Компьютерные исследования и моделирование. 10, 867–878;
- Mikhail Orlov, Irina Garanina, Gleb Y. Fisunov, Anatoly Sorokin. (2018). Comparative Analysis of Mycoplasma gallisepticum vlhA Promoters. Front. Genet.. 9;
- М.А. Орлов. (2020). Паразитизм в особо мелком размере: микоплазма и ее 40 промоторов, "Природа". Priroda. 3-10;
- Chen-Yu Lo, Yang Gao. (2021). DNA Helicase–Polymerase Coupling in Bacteriophage DNA Replication. Viruses. 13, 1739;
- Valeriy V. Panyukov, Olga N. Ozoline. (2013). Promoters of Escherichia coli versus Promoter Islands: Function and Structure Comparison. PLoS ONE. 8, e62601.







