Второй язык ДНК
22 февраля 2021
Второй язык ДНК
- 2729
- 0
- 4
ДНК не то, чем кажется. И то, чем кажется, тоже. Но не всегда...
иллюстрация автора статьи
-
Автор
-
Редакторы
Статья на конкурс «Био/Мол/Текст»: ДНК — основа жизни. В «сердце» клеточного ядра, в самом главном «узле» замысловатых путей метаболизма, в основании потока генетической информации покоится эта молекула — хранилище данных. Но только ли покоится? И ограничивается ли такое крупное и очень раннее изобретение эволюции «инертной» функцией хранения информации? Чтобы разобраться с этими вопросами, мы рассмотрим различные аспекты организации этой молекулы. Каждый из них может «выйти на первый план» при смене ролей ДНК — она может быть и матрицей для копирования, и местом хирургически точной посадки белков или сложных и динамичных взаимодействий с ними... В центре нашего внимания — свойства промоторов, участков, на которых начинается процесс транскрипции — «переписывания» ДНК в форму РНК. Место действия — крошечный геном бактериофага Т7 и его минималистические, очень похожие и такие различные промоторы.
Конкурс «Био/Мол/Текст»-2020/2021
 Эта статья участвовала в конкурсе «Био/Мол/Текст»-2020/2021 в номинации «Своя работа» и будет опубликована в журнале «Природа».
Эта статья участвовала в конкурсе «Био/Мол/Текст»-2020/2021 в номинации «Своя работа» и будет опубликована в журнале «Природа».

Партнер номинации — Российский научный фонд.

Генеральный партнер конкурса — ежегодная биотехнологическая конференция BiotechClub, организованная международной инновационной биотехнологической компанией BIOCAD.

Спонсор конкурса — компания SkyGen: передовой дистрибьютор продукции для life science на российском рынке.

Спонсор конкурса — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.

«Книжный» спонсор конкурса — «Альпина нон-фикшн»
Место ДНК в клетке и обществе
Представления о ДНК сейчас есть у любого. Рэперы упоминают ее в своих текстах, sci-fi-фильмы и сериалы показывают нам ДНК разноцветной и вращающейся, а ее имя и образ мелькают в дизайне и рекламе — виртуальных и уличных. Не столько с сожалением, сколько с иронией отметим: образ, порой напрочь потерявший асимметрию желобков и правильное число оснований на виток, не говоря уже о правозакрученности. А ведь есть еще и ДНК стиля или бренда... Действительно, двухспиральный, копирующийся, таящий информацию о самом сокровенном образ плотно «лег в руку» творческих людей. Но самое, пожалуй, неопровержимое доказательство врастания ДНК в культуру и язык — ее собственная пиктограмма. Мессенджеры даже предлагают данный значок для обозначения слова «наука» наряду с учеными в белых халатах, пробирками и телескопами (рис. 1).

Рисунок 1. Пиктограммы ДНК в различных мессенджерах и социальных сетях сделают разговоры о генетике короткими и образными. Важно только не забывать — ДНК не всегда длиной в несколько витков и не сугубо «слева направо вверх».
Всё это радует и служит популяризации науки, однако не стоит забывать что в жизни — в биологической жизни — ДНК — это, чаще всего, правозакрученная двойная спираль, шаг которой охватывает примерно 10 нуклеотидов.
В тени молекулярной догмы
Итак, дезоксирибонуклеиновая кислота чрезвычайно важна и хранит информацию — а именно генетическую информацию. Чтобы ее ценное содержимое не оставалось «вещью в себе», ДНК необходимо записывать, копировать и считывать. Такие «информационные связи» ДНК были емко сформулированы Фрэнсисом Криком в его центральной догме молекулярной биологии (обозначим кратко — ЦДМБ) вскоре после установления знаменитой двухспиральной структуры молекулы в 1953 г. Согласно этой «научной догме» информация, сохраненная в последовательности нуклеотидов ДНК, может:
- реплицироваться в новую копию ДНК;
- транскрибироваться в РНК.
- транслироваться в белок;
- «откатиться» обратно в ДНК в ходе обратной транскрипции (рис. 2).
И уже РНК далее способна (хотя и не обязательно — может просто функционировать в клетке сама по себе):
или в особом, описанном позже, случае
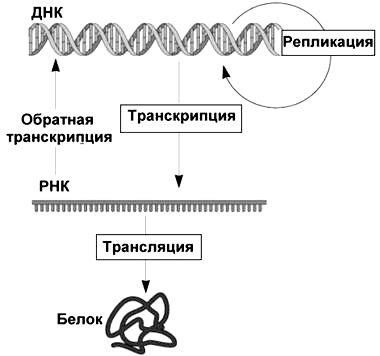
Рисунок 2. Центральная догма молекулярной биологии
Простая и легко запоминающаяся схема ЦДМБ имеет всего 3 блока (соответствуют трем типам биополимеров) и 4 стрелки (соответствуют процессам, связанным с переносом информации). Она действительно стала столпом в центре молекулярной биологии, придала генетике новый смысл и оказалась «точкой роста» бесчисленных метаболических путей биохимии. И в центре центральной догмы — молекула ДНК! Однако рассматривая в ней блок ДНК, стоит помнить: «ДНК» здесь — это не вся и не всякая ДНК. Речь идет только о кодирующей ДНК, нуклеотидной последовательности которой предстоит быть транскрибируемой, а далее еще и транслируемой (причем не всегда). Репликация, впрочем, копирует всю молекулу, не разбирая областей разного типа. Зато точки ее начала — óриджины (от англ. origin — «начало») — имеют вполне определенное положение.
Что же осталось в тени процессов, объединенных информационными потоками центральной догмы? Что бы это ни было, ему есть где развернутся. На некодирующие области приходится большая часть эукариотических геномов (в случае человека — увесистые 98%). У прокариот их поменьше — в среднем, 20%. По началу такие области, не содержащие послание для передачи в РНК, называли мусорной ДНК (junk DNA) [1] и относились к ним с пренебрежением. Прошли годы, методики совершенствовались, знания неуклонно копились. И теперь мы знаем: в этом сумраке таится много полезного. Что же именно и в какой пропорции?
В некодирующей области ДНК есть, скажем, мобильные генетические элементы (прежде всего, способные перемещаться по геному транспозоны [2]), тандемные повторы разного сорта (от сателлитов до микросателлитов) [3] и прочее. Наконец, огромное значение имеют области ДНК, нужные для регуляции. Действительно, помимо блоков, ЦДМБ имеет еще и стрелки — они обозначают процессы передачи информации, вполне конкретные биохимические процессы.
Их делают возможными ферменты, по своей природе — белки. Целый «ансамбль» белков требуется для молекулярной хореографии при репликации, РНК-полимераза и ее свита необходимы при транскрипции и т.д. Важно помнить: ДНК — также активный и деятельный участник этих процессов [4].
Что регулирует эти сложные процессы, что управляет ими? Специфичное, хорошо оркестрованное взаимодействие ДНК и ДНК-связывающих белков. Дело в том, что в живой клетке (ткани, природе...) все должно быть динамично и управляемо. Это жизненно необходимо и для разумного реагирования на удары судьбы (стрессы), и для разного рода взаимодействий клеток, и для индивидуального развития (онтогенеза).
И ДНК в этих межмолекулярных взаимодействиях подойдет не всякая: вряд ли транскрипция случайного участка генома станет разумной стратегией. В этом месте кажущаяся монотонной бесконечной ниткой с бусинами-нуклеотидами ДНК должна заиграть в нашем воображении новыми красками. В фокусе нашего внимания оказываются участники регуляции «со стороны ДНК» — ориджины репликации, промоторы, сайты формирования нуклеосом и т.п. И здесь ДНК не отделается снисходительным «позволением себя считать». Ее специализированные области регуляции чувствительны и деятельны. Они «ощущают» происходящее вокруг них — могут изменять свое состояние при изменении условий среды или при приближении «нацеленного» на них белка [5].
Притча о слепых белках и ДНК
Безусловно, ДНК далеко до структурного разнообразия белков с их неисчислимыми фолдами, укладками и т.п. Однако и у нее можно выделить иерархию уровней организации: от первичной (последовательность нуклеотидов) через небольшое разнообразие вторичных и третичных структурных блоков до четвертичной. Последняя представляет собой надмолекулярные объединения — как между разными молекулами ДНК, так и между ДНК и ДНК-связывающими белками.
Кодирующая функция той части ДНК, которая служит «матрицей» для синтеза других биополимеров, связана с особой организацией. А именно — информативные части генов руководствуются особыми «правилами грамматики» генетического кода:
- триплетность;
- неперекрываемость;
- вырожденность;
- универсальность;
- наличие кодонов — знаков препинания;
и некоторыми другими.
Все эти законы не действуют на некодирующей части генома. А действуют совсем другие и для каждого типа последовательности они, в общем, свои.
Итак, работа ДНК как матрицы реализуется на уровне ее нуклеотидной последовательности. Она сводится к прочтению последовательности нуклеотидов в качестве своеобразного сигнала — такая информация может рассматриваться в отрыве от «физической сущности», самой молекулы ДНК. Об этом очень емко сказал Леонард Адлеман: «ДНК по своей сути что-то цифровое» (DNA is essentially digital). Разумеется, такому хранилищу байтов безразличны более сложные уровни организации — здесь ДНК лучше вытянуться в нитку и быть одномерной.
Таковы принципы кодирования и хранения генетической информации, изображенные в виде блоков ЦДМБ. А как насчет способов ее воплощения, то есть перевода информации одного биополимера в другой, а также всех последующих этапов экспрессии генов? Что стоит за стрелками на схеме догмы, соединяющей квадраты? Большая биохимическая работа и сложные структурные основы. Эти процессы протекают во вполне физической реальности — на них согласованно работают многие ферменты, факторы... Вовлечены в эту 3D-хореографию и специализированные области ДНК. Что же делает их такими специализированными?
В первом приближении — им следует связывать белковые молекулы. Для этого служат опять-таки разные уровни организации регуляторных ДНК: особая последовательность, структура и физико-химические свойства.
С первичной структурой (другой способ обозначить ее — сказать «нуклеотидная последовательность») все вроде проще. Если ДНК-связывающий белок находит целевой сигнал (особую последовательность нуклеотидов), то для начала связывается в этой локации. Такой сигнал может быть строгим и однозначным, как, например, в случае рестриктаз с вполне определенным сигналом из небольшого числа нуклеотидов и четким же положением, по которому происходит расщепление ДНК относительно него (в чем, собственно, и состоит задача этих эндонуклеаз) (рис. 3).

Рисунок 3. Сайт специфичного и точного связывания важной рестриктазы EcoRI и место разреза — расщепления ДНК
рисунок автора статьи
С белками вроде факторов транскрипции (TF) все посложнее. Уровень аффинности — то, насколько конкретные регулирующие уровень транскрипции белки активно связывают участок ДНК, — значительно разнится. Области ДНК, в принципе способные вызвать интерес фактора транскрипции, должны соответствовать некоторому нестрогому «фотороботу» последовательности. В результате сайтами их связывания может стать целый спектр различных первичных структур. В ряде положений им следует иметь вполне конкретный нуклеотид, в других — можно позволить себе некоторую свободу замены, в третьих — нет особой разницы, что за мономер находится в данном положении. Такие местами-строгие-местами-не-очень требования к участку ДНК описывают позиционными весовыми матрицами и изображают в виде логотипов последовательности. В них высота соответствующей буквы отражает вероятность нахождения здесь данного нуклеотида либо долю сайтов с этим мономером для рассматриваемого набора (рис. 4).

Рисунок 4. Сайт связывания фактора транскрипции CEBPB
Таким образом,
- скользящая вдоль кодирующей области гена РНК-полимераза;
- вносящая строго определенный разрез вблизи строго определенной последовательности распознавания рестриктаза;
- фактор транскрипции, сидящий на «примерно таком вот участке, где А почти всегда в седьмом положении, но не когда G — в третьем»
видят ДНК совершенно по-разному.
Картина напоминает притчу о слоне и трех слепых мудрецах. Кому-то достался бок, кому-то хвост или хобот. Однако ДНК-связывающих «мудрецов» в клетке существует огромное разнообразие, и спорить кто прав им ни к чему — это сугубо человеческое занятие.
Пo мере того, как ученые вникали в интимные стороны взаимодействий ДНК и ДНК-связывающих белков, они стали различать прямое (direct) и непрямое прочтения (indirect readout) нуклеотидной последовательности. В случае прямого прочтения распознавание и связывание двух биомолекул определяется теми парами оснований, которые, собственно, контактируют с белком. Здесь все понятно. Но уже довольно давно биологи стали отмечать: не вовлеченные в прямые взаимодействия нуклеотиды определяют стабильность и специфичность связывания. В этом и состоит непрямое прочтение.
В числе первых этот аспект распознавания описан на примере такого важного фактора транскррипции, как TATA-связывающий белок (TATA-binding protein, TBP). После этого последовало множество других примеров непрямого прочтения [6].
Но и этого мало: выделили также прочтение формы (shape readout) — оно определяется трехмерной формой дуплекса ДНК. В основе этого нового видения той же ДНК — вандерваальсовы и электростатические взаимодействия. Напомним, что прямое прочтение ДНК белком зависит от водородных связей, образованных конкретными нуклеотидами и аминокислотными остатками [7].
Едем дальше: поскольку для специфического связывания ДНК и белков им нужно найти друг друга в плотном и подвижном клеточном супе, начинать надо издалека. Для этого служат различные физико-химические параметры дуплекса, причем с довольно широким контекстом — влияние физики простирается довольно далеко. В определенных случаях — на тысячи пар оснований! Список важных для регуляторных ДНК качеств следует дополнить более крупномасштабной электростатикой, исходной изогнутостью и склонностью «прогибаться» под белком, термостабильностью и многими-многими (не преувеличение) другими.
Следующий пункт. Важность великого множества одних только физико-химических и структурных свойств (не забывая и первичную структуру!) связано с тем, что, скажем, транскрипция — процесс многостадийный, и на разных этапах одни параметры ДНК могут быть важнее других. Даже инициация транскрипции насчитывает несколько этапов: посадка белков, образование закрытого комплекса, плавление дуплекса (то есть расхождение цепей) с образованием открытого комплекса и т.д. Кстати, именно инициация транскрипции — в особенности на примере прокариотических промоторов и простых РНК-полимераз, — служит элементарной системой для изучения всей сложной кухни молекулярного узнавания [8], [9].
Игра «найди промотор»
Помимо развития фундаментальной науки — включая структурную биологию и молекулярную генетику — интерес к структурным и физическим основам ДНК-белковых взаимодействий растет и из насущной практической задачи. Речь об автоматизированной аннотации геномов: применении методов биоинформатики для предсказания положения участков ДНК различного типа. Данные на входе такого анализа сейчас как из рога изобилия сыплются из секвенаторов нового поколения (NGS) [10], и их нередко замедляют затруднения «сухой» части аннотирующего конвейера. Безусловно, неоценимую помощь ученым оказывают машинное обучение и анализ данных [11]. Однако проблема не только в их несовершенстве. Дело в том, что прямой учет последовательности («текста» и «букв») четко позволяет узнать кодирующие последовательности, организованные в виде триплетов. А вот с регуляторными участками все сложнее — структура и физико-химия ДНК определяются последовательностью в широком контексте, подчас неявно и противоречиво. Рассчитав профили разных свойств ДНК, настроив надлежащий размер скользящего окна и других параметров расчета вроде условий в клетке, мы можем лучше предсказывать «размытые» и плохо предсказываемые сайты для ДНК-связывающих белков. Из таких задач самые насущные — предсказание промоторов (давняя, но едва ли решенная проблема биоинформатики) и факторов транскрипции [8], [9], [12], [13].
«Бактериофаг системы Т7»
Прогресс в науках о живом знает немало научных революций и периодов прорывного развития. Такие вдохновляющие эпохи не всегда определялись новой концепцией или гениальным пророком. Иногда он был связан с находкой новой модельной системы, которая очень хорошо подходит для исследования определенного круга проблем. «Биомолекула» посвятила настенный календарь 2020 года таким «супермоделям» — эпохальным для науки исследуемым ею живым системам. Так, Neurospora grassa — плесневый гриб, имеющий гаплоидный набор хромосом и оттого немедленно фенотипически выдающий любые мутации; маленькая аквариумная рыбка Danio rerio — удобный объект для изучения развития позвоночных и т.д. Хорошей живую модель делает простота организации, удобство в содержании и «прозрачность» — легкость, с которой мы можем наблюдать интересующий нас целевой процесс или явление. В идеале в этом модельном объекте хотелось бы еще «что-то подкрутить и поломать», чтобы узнать, для чего же оно там было исходно.
На каком примере стоит изучать ДНК-белковые взаимодействия и дополнительные к кодирующим свойства ДНК? Хороший вариант — транскрипция, первая «стрелка» на пути от ДНК «вниз по центральной догме». В ней в ходе сложных и согласованных взаимодействий сразу многих молекул происходит инициация на вполне определенных и находящихся в положении «ВКЛ» в данный момент промоторах. А факторы транскрипции участвуют в этом на правах регуляторов процесса, садясь на некоторые сайты посадки либо покидая их. Эта вакханалия подставляет великое множество примеров молекулярного узнавания и иерархической регуляции.
Пожалуй, хотелось бы вариант попроще... Для этого стоит взяться за транскрипцию прокариот. Дело в том, что ситуация в мудреном случае ядерных пугает. У них (и, стало быть, у нас) есть как минимум три РНК-полимеразы, в растениях и вовсе пять, и для их инициации и последующей элонгации нужно немало вспомогательных белков-факторов. Бактерии же обнадеживают: РНК-полимераза у них единственная, всего лишь «о пяти субъединицах» (самая ходовая эукариотическая Pol II имеет 12) и снабжена комплектом сигма-субъединиц. Эти «пристегивающиеся» модули определяют специфичность прокариотической РНК-полимеразы — то, какие промоторы могут ее заинтересовать прямо сейчас. Дежурный из этих факторов, используемый чаще прочих на фазе роста культуры, — сигма-70.
Но и это не предел. Меньше бактерий только вирусы, в том числе, бактериофаги. Эти «пожиратели» прокариотических жертв имеют соответствующий генетический аппарат. И он может на фоне исключительной изобретательности таких наноразмерных паразитов поражать небывалой простой. Обратимся в качестве удобного и ну совсем элементарного примера полимер-полимерного узнавания и роли физико-химии ДНК в инициации ДНК к бактериофагу Т7. Это последний в ряду исходно описанных фагов, поражающих Escherichia coli (рис. 5).

Рисунок 5. Бактериофаг Т7 — срез через вирион (слева) и общий вид
Сложно представить себе что-то более просто устроенное, чем Т7. Вирион бактериофага или, скажем, комплекс поры ядра клетки — примеры того, как наноразмерные биологические структуры могут превзойти любого фантаста. Тут и икосаэдрическая вирусная частица, и хвостовые нити, и система «вбрасывания» чужеродной ДНК в клетку хозяина... Настоящий роботизированный зонд-подрывник!
Его жертва — кишечная палочка E. coli — самый популярный прокариотический объект для исследований. И Т7 — инфекционный агент — внедряется внутрь ее клетки. Точнее будет сказать «в клетку внедряется его геном» — он вбрасывается через канал в основании вирусной частицы, в то время как пустой «скафандр» остается на поверхности. Его задачей была доставка генома и специфическое связывание с E. coli. После этого начинается самое захватывающее — жизненный цикл бактериофага Т7.
Биография Т7 от начала до конца... До 3′-конца
Что же попало внутрь ничего не подозревающей и гигантской по сравнению с Т7 кишечной палочки? Молекула ДНК длиной около 40 тыс. пар оснований (килобаз), имеющая обе комплементарные цепочки. У вирусов бывает и совсем по-другому — на чем и основана система Балтимора. Не замкнутая в кольцо, а линейная ДНК, — в этом отношении тоже «всякое бывает». В 40 килобазах — и сам бактериофаг Т7 (в этот момент ничего больше у него не имеется), и история его жизни. У генома фага есть интересная особенность: его «левые» области (расположены ближе к 5′-концу) работают раньше, чем расположенные в середине и, тем более, прилегающие к 3′-концу. Получается, что Т7-ДНК «прочитывается» в правильном порядке слева направо. Поэтому ее разделяют на три последовательно «включающиеся» области. Пробежимся кратко по всем (рис. 6).
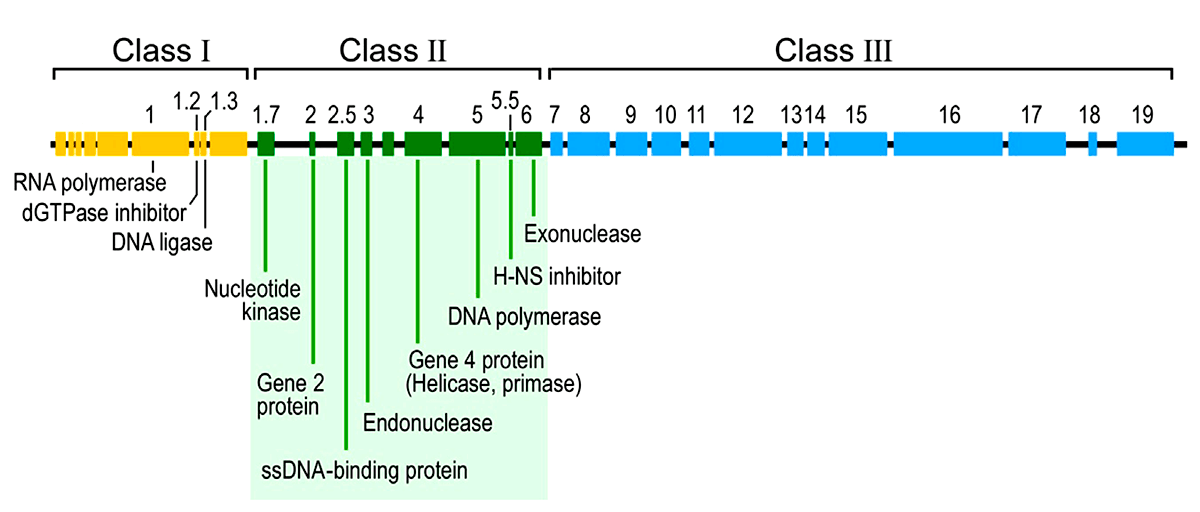
Рисунок 6. Геномная карта бактериофага Т7 — это и история его жизни «слева направо»
Ранняя область ближе всего в 5′-концу и включается первой. Она населена соответствующими ранними генами. Для их транскрипции не захвативший собственную РНК-полимеразу фаг «угоняет» таковую у E. coli. Для этого у Т7 имеются подходящие «посадочные площадки» — стандартные бактериальные промоторы. Пожалуй, разве что сильнее привлекающие прокариотическую РНК-полимеразу, чем ее собственные. Прием успешного паразита, отметим мы, — будь то бактериофаг или кукушка. К тому же многие из них «столпились» вместе у самого 5′-конца. В результате фазы I коварного плана бактериофаг изменяет состояние бактерии «под себя» и появляется его собственная Т7-РНК-полимераза, которая и берет дальнейшую транскрипцию (генов II и III класса) на себя.
Далее включается область генов II класса и соответствующих промоторов. Их задача — активная наработка ДНК бактериофага (ее репликация), а также синтез лизоцима — антибактериального фермента, с помощью которого потомкам бактериофага предстоит выбраться из гибнущей клетки. Стандартный сценарий для такого литического фага, как Т7.
Наконец, область генов и промоторов III класса. Их задача — обеспечить созревание фаговой ДНК, сборку новых вирусных частиц и затем упаковать одно в другое.
На этом жизненный цикл Т7 заканчивается литической и трагической концовкой. Весь экшн занимает обычно 17 мин, по минутам же расписано переключение между его фазами [13], [14]. Но остается довольно животрепещущий вопрос: что переключает активность областей этого элементарного генома?
Промоторы — промотируют, полимераза — полимеразит
Мы уже упоминали, что раннюю область Т7-ДНК транскрибирует «хозяйская» РНК-полимераза бактерии, а далее эту работу берет на себя фагоспецифичная Т7-РНК-полимераза. Стало быть, на одной небольшой ДНК соседствуют сильно различающиеся промоторы для этих двух ферментов. «Собственные» промоторы генома Т7 меньше: они имеют длину всего 19 нуклеотидов. Ферменты и сами не одного калибра: фагоспецифичная полимераза «весит» около 100 килодальтонов (кДа), бактериальная втрое увесистее. При этом транскрипционный дуэт «РНК-полимераза Т7 + промотор Т7» связывается очень точно и специфично, что пригодится в условиях, когда можно влезть в транскрипцию соседнего похожего фага.
Вопрос возникает в связи с тем, что промоторы Т7 из разных классов или даже внутри одного класса очень похожи по своей последовательности. Многие промоторы класса III и вовсе идентичны. Напомним: речь, идет о последовательностях рекордно малой длины — всего 19 нуклеотидов. Очень напоминают они по своей последовательности и таких фагов, как SP6 или, особенно, Т3. Каким же образом Т7-РНК-полимера различает «свои» промоторы и среди своих — те, которые пришла пора связать? Различать вроде как особенно нечего... (рис. 7).

Рисунок 7. Едва различимые последовательности очень непохожих по своим свойствам нативных промоторов бактериофага T7
Здесь на сцену выходят физико-химические свойства дуплекса промоторных областей. Они могут объяснить, как РНК-полимераза Т7 распознает их и специфично, и переключаемо. Для этого процесса молекулярного узнавания важнее именно уровень физико-химических свойств ДНК — что особенно явственно в случае предельно простой транскрипционной системы Т7 [14].
Какими же параметрами ДНК могут направляться взаимодействия биомолекул?
Задолго до прямого контакта молекулярному узнаванию промотора и полимеразы может способствовать электростатический потенциал. Действительно, если мы вспомним, что каждый ее мономер-нуклеотид имеет заряженную отрицательно фосфатную группу, станет понятно — на ДНК есть к чему притянутся положительному заряду. В полном соответствии со школьным законом Кулона. Действительно, ДНК-связывающие белки с этой целью снабжены положительно заряженными участками. В случае же промоторов Т7 присутствуют и особые мотивы распределения заряда [15]. Если мы рассчитаем значение заряда вдоль оси ДНК (на основе известной последовательности нуклеотидов), то получим характерные профили (рис. 8). Заметим, что свой профиль есть и у промоторов II и III класса — по ним, в частности, фаговая РНК-полимераза их и различает.
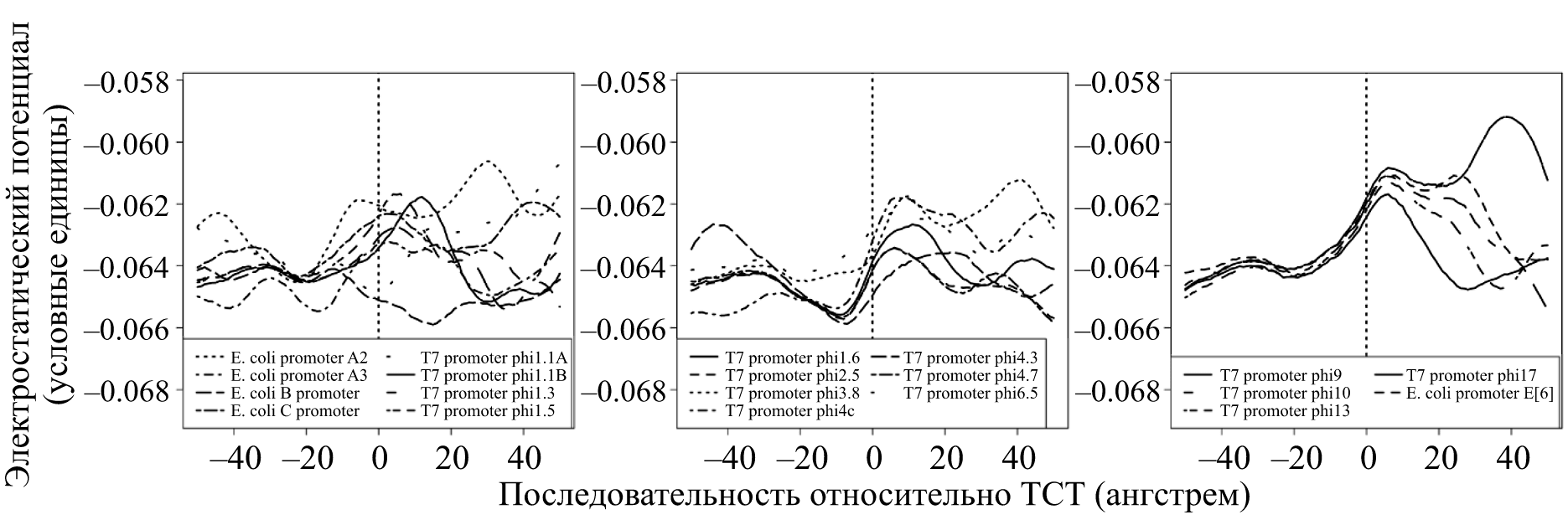
Рисунок 8. Профили электростатического потенциала для промоторов Т7: ранних, II класса и III класса
Следующее важное свойство ДНК — дестабилизация при скручивании. Сложно подобрать простое название этому параметру, так что приведем какое есть: вызванная суперспирализацией дестабилизации дуплекса ДНК (Stress-Induced Duplex Destabilization). Если коротко, модель SIDD — это способ описать, что будет с ДНК, если на нее как следует надавить, а точнее — скрутить. Дело в том, что одни последовательности в такой напряженной ситуации могут легко расплавиться (так называют расхождение цепей ДНК), другие — выстоят, третьи — плавятся совсем не там, где от них ждешь... Это свойство важно для взаимодействия ДНК с белками и, в частности, хорошо зарекомендовало себя для предсказания промоторов [9]. Как же обстоят дела с SIDD у промоторов Т7?

Рисунок 9. Профиль SIDD для генома Т7
рисунок автора статьи
Посмотрим на профиль SIDD для всего генома бактериофага Т7. Его действительно лучше рассматривать в крупном масштабе. Дело в том, что SIDD может быть очень чувствителен к изменениям последовательности, причем довольно непредсказуемым образом. Создатели этой модели продемонстрировали: небольшое изменение последовательности может резко изменить способность плавиться при суперспирализации участка ДНК, отдаленного от «мутации» на многие сотни пар оснований. По-видимому, такие эффекты использует и природа.
Из набора промоторов Т7 SIDD показывает следующий тренд. Если промотор представляет класс III, то он гораздо более легкоплавкий — цепи дуплекса такого участка ДНК расходятся при меньшей температуре и более низкой суперспирализации. Это неплохо соотносится с экспериментальными данными о том, что промоторы разных классов активны при разных условиях окружающей среды — включая как раз таки показатель суперспиральности [4], [9].
Другая закономерность затрагивает репликацию, для которой Т7-ДНК также является хорошей моделью. Этот геном имеет специализированный ориджин (точку начала репликации), отдаленный от 5′-конца на 17% длины генома. Однако его копирование может начинаться также в ряде других точек — и это как раз таки некоторые промоторы. Удивительно, но именно они относятся к числу дестабилизированных. Удивительно и осмысленно, поскольку репликация также может требовать особых физико-химических свойств дуплекса. В частности, легкость плавление дуплекса ДНК здесь точно не помешает [5].
Безусловно, на электростатике и SIDD важные физико-химические свойства ДНК не кончаются. Изучают и термостабильность, и изогнутость, энтропию, энергию стекинга (складывания нуклеотидов «тарелками» друг на друга) и бесчисленные другие [8].
Подобные закономерности физико-химических различий промоторов ранее оказались важны для бактерии — внутриклеточного паразита Mycoplasma gallisepticum. Ее геном содержит гены семейства vlhA, собственно, делающие этого патогена патогенным — способным «обходить» иммунную защиту хозяина за счет быстрой смены репертуара поверхностных антигенов. Выяснилось: гены vlhA снабжены промоторами [16], разительно отличающимися по физико-химии дуплекса от остальных промоторов генома [17].
Исчерпывающие мутанты
Сложный характер зависимости промоторной активности от физико-химических свойств ДНК и от нуклеотидной последовательности требует полных или по меньшей мере больших данных. Вот если бы получить все или почти все возможные варианты промотора Т7 и узнать, насколько они активны... А ведь в постгеномную эпоху и век дешевых NGS это как раз таки реализуемо! И уже проделано в отношении промотора Т7. В работе [17] использована высокопроизводительная методология: в небольшие фрагменты ДНК встроили почти 8 тысяч случайным образом измененных последовательностей промотора фага Т7, а также последовательности-метки (баркоды) — чтобы потом разобрать, кто есть кто. Далее напрямую замерили их промоторную активность. Имея в распоряжении полные последовательности (промотор + небольшой контекст), нетрудно и рассчитать те физико-химические параметры дуплекса, которые не требуют значительных участков ДНК на входе (SIDD здесь, к сожалению, отпадает — ведь для расчета этого параметра дуплекса нужны очень крупные фрагменты ДНК).
Что показала данная работа и последующий анализ данных о последовательности, физико-химии и величине промоторной активности? Прежде всего, она подтвердила многие выводы о важности отдельных нуклеотидов в конкретных положениях и согласованности таких замен. Нетривиальный результат: ранее для этого ученые не одно десятилетие скрупулезно изучали геном T7 с помощью доступных тогда методов. И теперь мы знаем, что высокопроизводительный, основанный на NGS подход — хороший способ разобраться в устройстве других промоторов и вообще вовлеченных в регуляцию областей ДНК. В том числе в новых, не исследованных геномах, которые нам поставляют бесчисленные секвенаторы.
В отношении теоретической стороны вопроса промоторно-полимеразного узнавания эти работы также дали интересный результат. Оказалось, что положение –2 промотора (если считать за единицу точку старта транскрипции), которому уделялось сравнительно мало внимания при описании структурных основ инициации, очень важно для физико-химических свойств ДНК. Действительно, замена данного нуклеотида способна резко изменить профиль электростатического потенциала или склонности к изгибам. В этом положении промоторы бактериофагов (не только Т7, но и нескольких других сходной «конструкции») почти всегда находятся нуклеотиды A либо T, в единичных промоторах из десятков — G или C (рис. 7). В пределах же промоторов Т7-ДНК нахождение А или Т определяется как раз принадлежностью к «противопоставленным» друг другу группам — классам II и III. Означает ли это особую миссию –2 положения промотора Т7 или нет? Мы надеемся, что эксперименты вскоре помогут это установить [18], [19].
Литература
- Сколько сора в нашей ДНК;
- Мода на ретро. Где встречается обратная транскрипция, и как она эволюционировала;
- Повтор, еще повтор!;
- М. А. Орлов, А. А. Рясик, А. А. Сорокин. (2018). Дестабилизация дуплекса ДНК активно реплицирующихся промоторов бактериофагов группы Т7. Молекул. биол.. 52, 793-800;
- Mikhail Anatolievich Orlov, Svetlana Grigor'evnu Kamzolova, Artem Andreevich Ryasik, Evgenia Andreevna Zykova, A. A. Sorokin. (2018). Stress-induced duplex destabilization (SIDD) profiles for T7 bacteriophage promoters. CRM. 10, 867-878;
- Gerald B. Koudelka, Steven A. Mauro, Mihai Ciubotaru. (2006). Indirect Readout of DNA Sequence by Proteins: The Roles of DNA Sequence‐Dependent Intrinsic and Extrinsic Forces. Progress in Nucleic Acid Research and Molecular Biology. 143-177;
- Max Schnepf, Marc von Reutern, Claudia Ludwig, Christophe Jung, Ulrike Gaul. (2020). Transcription Factor Binding Affinities and DNA Shape Readout. iScience. 23, 101694;
- Ilham Ayub Shahmuradov, Rozaimi Mohamad Razali, Salim Bougouffa, Aleksandar Radovanovic, Vladimir B. Bajic. (2016). bTSSfinder: a novel tool for the prediction of promoters in cyanobacteria andEscherichia coli. Bioinformatics. btw629;
- Huiquan Wang, Craig J Benham. (2006). Promoter prediction and annotation of microbial genomes based on DNA sequence and structural responses to superhelical stress. BMC Bioinformatics. 7;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Кластерный анализ и дилемма биологического пользователя;
- Artem Ryasik, Mikhail Orlov, Evgenia Zykova, Timofei Ermak, Anatoly Sorokin. (2018). Bacterial promoter prediction: Selection of dynamic and static physical properties of DNA for reliable sequence classification. J. Bioinform. Comput. Biol.. 16, 1840003;
- A.W. Kulczyk, C.C. Richardson. (2016). The Replication System of Bacteriophage T7. DNA Replication Across Taxa. 89-136;
- Орлов М.А. (2020). Текст и подтекст: физические свойства ДНК. «Потенциал. Химия. Биология. Медицина». 2, 13–23;
- Сорокин А.А., Джелядин Т.Р., Орлов М.А., Зыкова Е.А., Камзолова С.Г. (2016). Пространственная организация электростатических взаимодействий Т7 РНК-полимеразы с поздними промоторами Т7 ДНК. «Вестник биотехнологии и физико-химической биологии имени Ю.А. Овчинникова». 4, 64–71;
- Али-Баба и 40 промоторов;
- Mikhail Orlov, Irina Garanina, Gleb Y. Fisunov, Anatoly Sorokin. (2018). Comparative Analysis of Mycoplasma gallisepticum vlhA Promoters. Front. Genet.. 9;
- Ryo Komura, Wataru Aoki, Keisuke Motone, Atsushi Satomura, Mitsuyoshi Ueda. (2018). High-throughput evaluation of T7 promoter variants using biased randomization and DNA barcoding. PLoS ONE. 13, e0196905;
- Mikhail A. Orlov, Anatoly A. Sorokin. (2020). DNA sequence, physics, and promoter function: Analysis of high-throughput data On T7 promoter variants activity. J. Bioinform. Comput. Biol.. 18, 2040001;
- Z. Chen. (2005). Information theory based T7-like promoter models: classification of bacteriophages and differential evolution of promoters and their polymerases. Nucleic Acids Research. 33, 6172-6187.