Автоматизация омиксных технологий
05 февраля 2021
Автоматизация омиксных технологий
- 2297
- 0
- 7
Омиксные технологии помогают решать комплексные задачи биологии человека. Рисунок в полном размере.
рисунок Анастасии Волчок
-
Авторы
-
Редакторы
-
Иллюстратор
Омиксные технологии — в первую очередь, геномика и протеомика, — работают с огромными объемами данных, поэтому без автоматизации процессов не обойтись. В этой статье спецпроекта «Автоматизация в биологии» мы поговорим о том, что можно и что нельзя автоматизировать в этих областях, и как это реализовано в современных приборах.
Автоматизация в биологии

Работа в современной биологической лаборатории кардинально отличается от той, что велась в ней каких-то 100–150 лет назад. Многие процедуры давно превратились в рутину, которую зачастую выполняет не сам экспериментатор, а автоматизированные приборы. Это позволяет не только упростить процесс, но и высвободить время для новых творческих задач. В спецпроекте «Автоматизация в биологии» мы проследим, какой путь прошли современные приборы, повсеместно использующиеся в экспериментах по геномике и протеомике, цитометрии, генной инженерии и драг-дизайну.
Куратором спецпроекта стал Денис Баев — глава центра коллективного пользования по проточной цитометрии и сортировке клеток Института канцерогенеза Кандиоло, Италия.
Партнер спецпроекта — компания Beckman Coulter Life Sciences, мировой лидер в разработке решений для автоматизации сложных биомедицинских лабораторных процессов.
Геномика: автоматизировали, автоматизируем и будем автоматизировать
«Нельзя так много учиться», — подумал 25-летний студент медицинской школы Лукас Вартман, когда начал сильно уставать. Но дело было не в учебе: вскоре у него диагностировали острый лимфобластный лейкоз. Юноша прошел курсы химиотерапии, выучился на онколога и стал врачом и исследователем в медицинской школе университета Дж. Вашингтона. Однако в 2011 году, через восемь лет после диагноза, случился рецидив. Состояние Лукаса было тяжелым, никто не знал, выживет ли он. В то время мужчина работал в лаборатории, которая изучала различия в геноме раковых и нормальных клеток. Эти знания уже применялись на практике: было известно, что некоторые лекарства действуют на клетки с мутациями в конкретных генах. В лаборатории решили отсеквенировать геном и транскриптом Лукаса и попробовать найти такие мутации. В результате была обнаружена избыточная экспрессия гена FLT3, ингибитором которого является низкомолекулярное соединение сунитиниб, одобренное FDA для лечения других онкологических заболеваний. Через несколько недель приема лекарства Лукас вошел в ремиссию, которая продолжается до сих пор. Так геномика спасла его жизнь, когда казалось, что уже ничто не поможет.
Геномика — что за зверь?
Исследование геномов раковых клеток для поиска мишеней для лечения онкологических заболеваний — лишь одно из направлений геномики. Геномика — это изучение полного генома организмов, возможность расшифровать геномы разных видов или особей внутри одного, сравнить их и найти какие-то закономерности: точнее определить, на что влияет тот или иной ген и к чему приводят его мутации или вариации. Такие знания нужны и фундаментальной науке, и для вполне утилитарных целей.
Например, геномика помогает больше узнать и об истории человечества. Ученые находят ответы на вопросы, как наши предки расселялись по миру [1], [2], какие у них были отношения с неандертальцами [3] или за что отвечают «неандертальские» гены, которые есть у некоторых людей. Судя по тому, что наличие таких генов коррелирует с курением, чувством одиночества и режимом «совы», многие из нас неандертальцы. Хотя бы в душе. Подробнее про генетическую генеалогию можно прочитать в спецпроекте «Генетическая генеалогия».
Геномика также служит и правосудию: помогает находить виноватых и освобождать невиновных. Так, в 2009 году благодаря тесту ДНК вышел на свободу Джеймс Бейн, который провел в тюрьме 35 лет по ошибке следствия. И таких невинно осужденных много: с 1989 года в США освободили 367 человек, и в среднем каждый отсидел по 14 лет (!). Подробнее о применении биологии в криминалистике читайте в другом нашем спецпроекте — о криминалистике.
Постепенно знания о геноме ложатся в основу утилитарных и доступных каждому генетических тестов. Сегодня любой желающий может обратиться в компанию, сдать слюну и получить «генетический паспорт» с информацией не только о вероятности заболеть той или иной болезнью и эффективности некоторых лекарств, но и о происхождении своих предков.
Данные о геноме нужны везде: от распутывания истории человечества и преступлений до диагностики и лечения разных заболеваний — преимущественно онкологических. Как же происходит расшифровка генома и как сделать процесс быстрее и дешевле? Три слона, на которых держится путь от биологического образца до знания о геноме, — выделение и очистка нуклеиновых кислот [4], полимеразная цепная реакция (ПЦР) [5] и секвенирование [6]. Все они подразумевают многоэтапную работу с маленькими объемами жидкостей. Чтобы сделать всё вручную и при этом не перепутать и не загрязнить образцы, исследователю понадобится много времени, внимания и аккуратности. Не все могут похвастаться такими качествами, а вот машина — может. Автоматизация — отличный способ сделать процесс быстрее и точнее и освободить исследователя от рутины. Посмотрим, как можно автоматизировать каждый этап.
Начинаем движение: готовим исходный образец
Современная геномика возникла в 70-х годах XX века, когда появились первые методы секвенирования. Однако в истории развития знаний о геноме можно выделить более ранние и не менее знаковые моменты. Например, в 1869 году Фридрих Мишер впервые заметил и выделил из клеток нуклеиновые кислоты. В 1953 году Фрэнсис Крик и Джеймс Уотсон (конечно, не без помощи Розалинды Франклин) открыли структуру ДНК. Постепенно значение нуклеиновых кислот как основы жизни на Земле становилось всё более очевидным, а методы работы с ними совершенствовались.
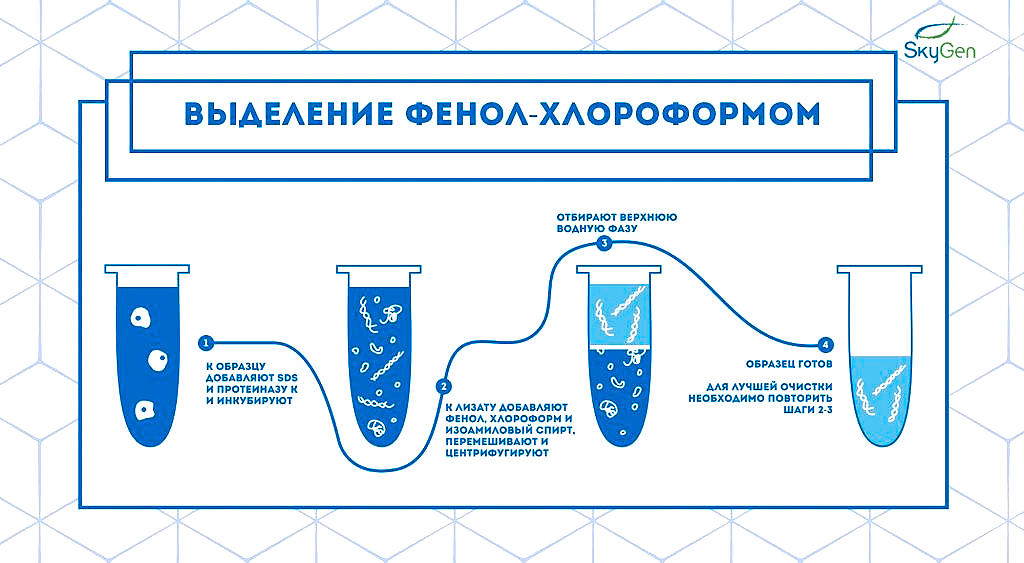
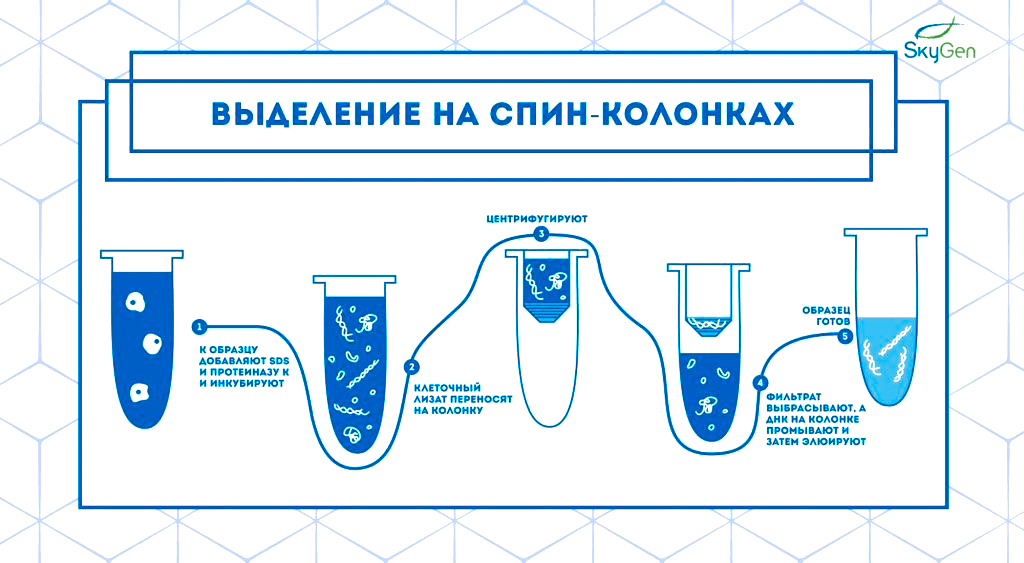
Сейчас методы выделения нуклеиновых кислот для дальнейшего анализа можно разделить на две группы: жидкофазные и твердофазные — в зависимости от того, в какой среде оказывается итоговый продукт [4], [7]. К первой относится, например, экстракция системой фенол-хлороформ: после центрифугирования лизированных клеток белки и липиды остаются в нижней органической фазе, а нуклеиновые кислоты — в верхней водной (рис. 1а). Пример твердофазного метода выделения — использование микроколонок (рис. 1б). Нуклеиновые кислоты связываются с силикатами, расположенными на мембране колонки, после чего промываются растворами и элюируются. Экстракция на микроколонках удобнее: не надо использовать токсичные вещества и предельно аккуратно отбирать нужную фазу, и выделенные нуклеиновые кислоты получаются чище. К тому же такое выделение проще автоматизировать: есть наборы множества производителей (например, Qiagen, Analytik Jena, NEB, ThermoFisher и др.), а также полу- и полностью автоматизированные платформы, например, от ADS BIOTEC. При работе с платформами QuickGene Auto 12S и Auto 24S пользователь только раскладывает необходимый пластик и готовые наборы реагентов, а также раскапывает EDB (раствор протеазы) и образцы. Всё остальное машина делает сама.
Но есть и недостатки: высокая вероятность контаминации из-за большого количества манипуляций и значительно более высокая стоимость одного выделения. Кроме того, в процессе множественных центрифугирований нуклеиновые кислоты могут деградировать, если оставить их на долгое время на воздухе без растворителя.
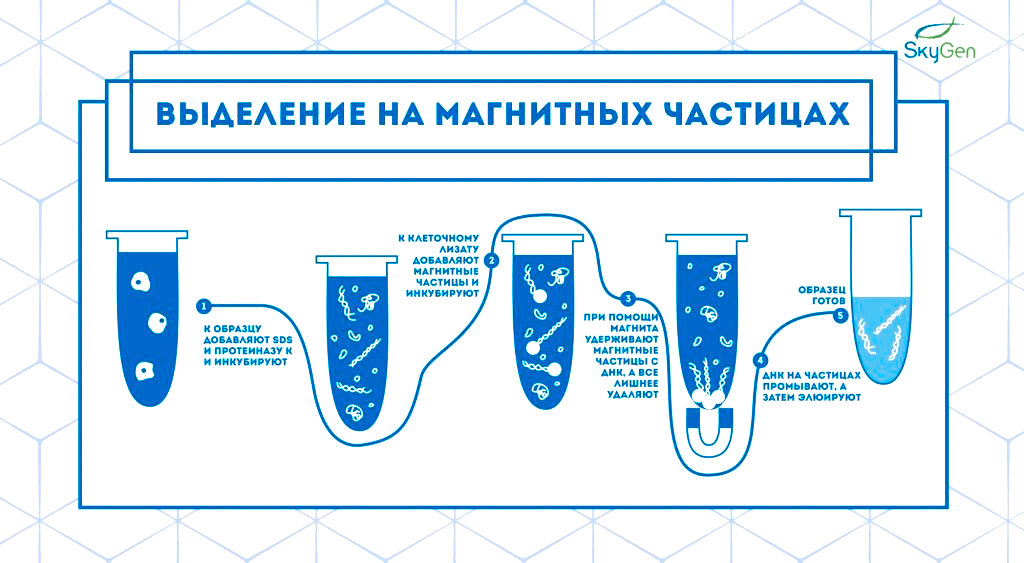
Решение без этого недостатка — выделение при помощи магнитных частиц (рис. 1в). Нуклеиновые кислоты связываются с веществом на поверхности частиц (это может быть целлюлоза, сефадекс, сефакрил, dT-олигонуклеотиды, специфичные олигонуклеотиды и др.), и их легко промыть, после чего отделить от раствора магнитом и отсоединить очищенный продукт [8]. У этого метода, конечно, тоже есть недостатки: образец по-прежнему легко загрязнить, да и стоимость выделения получается высокой — даже выше, чем при использовании колонок. Тем не менее многие автоматические станции выделения (например, платформы от PerkinElmer, Roche, ThermoFisher Scientific, Beckman Coulter, Biochain и Promega) используют именно эту технологию.
Существуют и «экзотические» решения для выделения нуклеиновых кислот, например, основанные на применении изотахофореза (это реализовано на платформе от Purigen Biosystems): выделение происходит на чипах, работающих по принципам микрофлюидики. Метод не требует этапов связывания или промывки, это снижает вероятность потери, фрагментации или контаминации продукта (видео 1).
Видео 1. Работа платформы IonicTM Purification System
Следующая остановка — ПЦР
ПЦР (полимеразная цепная реакция) — важный метод молекулярной биологии, который позволяет на несколько порядков увеличить содержание конкретного фрагмента ДНК в образце. Если кратко, это работает так: к образцу ДНК добавляется смесь реагентов, состоящая из ДНК-полимеразы, праймеров (коротких последовательностей нуклеотидов, комплементарных определенным участкам ДНК) и свободных нуклеотидов. Готовая смесь проходит циклы нагревания и охлаждения. Сначала при нагревании две цепочки исходной ДНК расходятся (денатурируют), потом при специально подобранной температуре к нужным участкам образца присоединяются («отжигаются») праймеры. Затем выставляется температура, оптимальная для работы ДНК-полимеразы, и та достраивает цепочки ДНК, начиная с праймеров и используя свободные нуклеотиды из смеси. Выбирая праймеры, мы определяем, какие участки образца амплифицировать. При каждом последующем цикле число нужных фрагментов ДНК будет увеличиваться в два раза: если сначала у нас был один фрагмент, в конце первого цикла их будет два, затем — четыре, восемь и так далее. За подробностями читатель благоволит обратиться к статье «12 методов в картинках: полимеразная цепная реакция» [5].
Сейчас проведение ПЦР стало рутинной молекулярно-биологической процедурой: услуги ПЦР-анализа доступны в любом медицинском центре и используются при диагностике практически всех мыслимых болезней. К тому же ПЦР — это часть подготовки к секвенированию на большинстве современных платформ: этот этап увеличивает концентрацию нужных фрагментов для последующего секвенирования. Также ПЦР — неотъемлемый шаг при редактировании генома.
Автоматизация метода ПЦР началась сразу после одобрения патента в 1985 году. Этому помогли, во-первых, технологические новшества, во-вторых, уже существовавшие открытия из смежных областей биологии. Так, в том же 1985 году появился первый прототип термоциклера — Mr. Cycle (рис. 2). Доработанный прибор PCR-1000 Thermal Cycler поступил в продажу через два года, в 1987-м. Это позволило автоматизировать простую, но монотонную работу: на протяжении многих часов эксперимента множество раз точно по секундомеру менять температуру пробирок — например, перемещая их между водяными банями с разной температурой. А в 1990 году была запатентована ПЦР с термостабильной Taq-полимеразой. Нетермостабильные аналоги быстро денатурировали и теряли активность под действием высоких температур, и перед каждым новым циклом приходилось добавлять в реакционную смесь новую порцию фермента. Оба нововведения позволили повысить контролируемость, точность и эффективность процесса.

Рисунок 2. Первый прототип термоциклера — Mr. Cycle, 1985 г.
Сейчас существует множество версий протокола постановки ПЦР. Есть и принципиально новые решения. Например, ПЦР в реальном времени — незаменимый в наше время метод, в котором совмещены амплификация и детекция результата. В оригинальном варианте для детекции используется электрофорез в агарозном или полиакриламидном геле. В ПЦР в реальном времени накопление продуктов реакции с присоединенными флуоресцентными метками или с интеркалирующими красителями фиксируют оптические датчики. Это позволяет получить количественный результат: если в обычной ПЦР мы только можем узнать, был ли в нашем образце конкретный фрагмент или нет, то в ПЦР в реальном времени при наличии контролей мы можем измерить количество копий, а, значит, точно рассчитать, сколько было исходной матрицы.
Еще более новое решение — капельная цифровая ПЦР. Коммерческое использование этой технологии началось в 2011 году. Из 20 мкл образца получают эмульсию, которую делят на 20 000 капель. В каждой капле проводится ПЦР, и наличие продуктов также проверяется по уровню флуоресценции меток. Целевая ДНК распределяется по каплям совершенно случайно, и по данным из каждой отдельной капли программное обеспечение прибора рассчитывает концентрацию ДНК-мишени. Подробнее о методе ПЦР, его вариациях и истории можно узнать в обзоре, посвященном ПЦР [5].
Итак, чтобы поставить ПЦР, надо раскапать множество компонентов и нигде не ошибиться. Поэтому и тут многофункциональные станции для дозирования жидкостей приходятся как нельзя кстати. А если к станции присоединить термоциклер, то прибор поставит, проведет ПЦР и очистит продукты реакции. И всё сам.
Существуют мини-лаборатории, в основе работы которых лежат принципы микрофлюидики. Это, например, система GeneXpert MTB/RIF для проведения быстрого теста на туберкулез. Оператор растворяет образец мокроты в реагенте, содержащем гидроксид натрия и изопропанол, переносит раствор на специальный картридж и вставляет его в прибор. В картридже есть несколько отделений и готовые сухие реагенты. Система отфильтровывает бактерии от остальной жидкости и разрушает их ультразвуком, а также смешивает готовый гомогенат с реагентами для ПЦР. После этого ПЦР в реальном времени детектирует наличие бактериальной ДНК (видео 2). Другие продукты из линейки GeneXpert используются в разных сферах молекулярной диагностики: с их помощью выявляют SARS-CoV-2, золотистого стафилококка, вирусы Эбола и гриппа и многие другие, а также определяют генетическую склонность к тромбозу и уровень онкогенной трансформации клеток при хроническом миелоидном лейкозе.
Видео 2. Строение и принцип работы картриджа GeneXpert MTB/RIF
Более универсальное решение — полностью автоматизированные многофункциональные станции для работы с жидкостями. Например, платформы Biomek от компании Beckman Coulter (видео 3). Платформы представлены в разных вариантах, совместимы с наборами от разных компаний, а в программном обеспечении уже записаны готовые программы для самых популярных процедур. При желании пользователь может адаптировать их под свои нужды или составить новые. Помимо выделения генетического материала, станция может, например, поставить ПЦР, приготовить библиотеку для секвенирования и многое другое. Станция передвигает и раскапывает образцы и реагенты по заданному протоколу. Платформы совместимы с более чем 300 приборами от других производителей. Для выделения нуклеиновых кислот уже есть готовые киты от Beckman Coulter: Beckman Formapure, RNAdvance, GenFind и пр.
Видео 3. Очистка ДНК технологией AMPure XP на платформе Biomek
Последняя разработка Beckman Coulter — станция акустического дозирования Echo 525, как раз заточенная под задачи геномики, протеомики и синтетической биологии. Жидкости переносятся каплями по 25 нанолитров, что позволяет дозировать реагенты с высокой точностью и скоростью — раскапывание планшета на 384 лунки занимает всего несколько секунд. Это в 50–100 раз быстрее, чем на обычных платформах для работы с жидкостями. Станцию можно использовать для подготовки ПЦР, секвенирования по Сэнгеру и NGS и других задач лаборатории.
Полный вперед: секвенирование
Выделение нуклеиновых кислот и ПЦР — это только присказка. Основной метод геномики — конечно же, секвенирование.
Немного истории: первая последовательность нуклеотидов была расшифрована лишь в 1964 году. Это была тРНК дрожжей, которая переносит аланин и состоит из 77 нуклеотидов. Исследователи семь (!) лет выделяли ее из дрожжей, расщепляли РНКазами и идентифицировали нуклеотиды методами хроматографии и спектрофотометрии. Первый геном целого организма — гемофильной палочки Haemophilus influenzae — был расшифрован через 21 год, в 1995 году.
В 2001 году был опубликован первый черновик генома человека. Его подготовка — международный проект «Геном человека» — заняла больше десяти лет, из них 15 месяцев самого секвенирования [9]. Чтобы расшифровать еще тысячу геномов, понадобилось всего три года [10]. Сейчас секвенирование одного генома занимает лишь пару дней. Стоимость анализа тоже снизилась в сотни раз: первый черновик человеческого генома обошелся в примерно $300 млн, сейчас одно секвенирование стоит меньше $1000 (рис. 3). Методы секвенирования очень хорошо разобраны в статьях «Биомолекулы» [6], [11], [12].
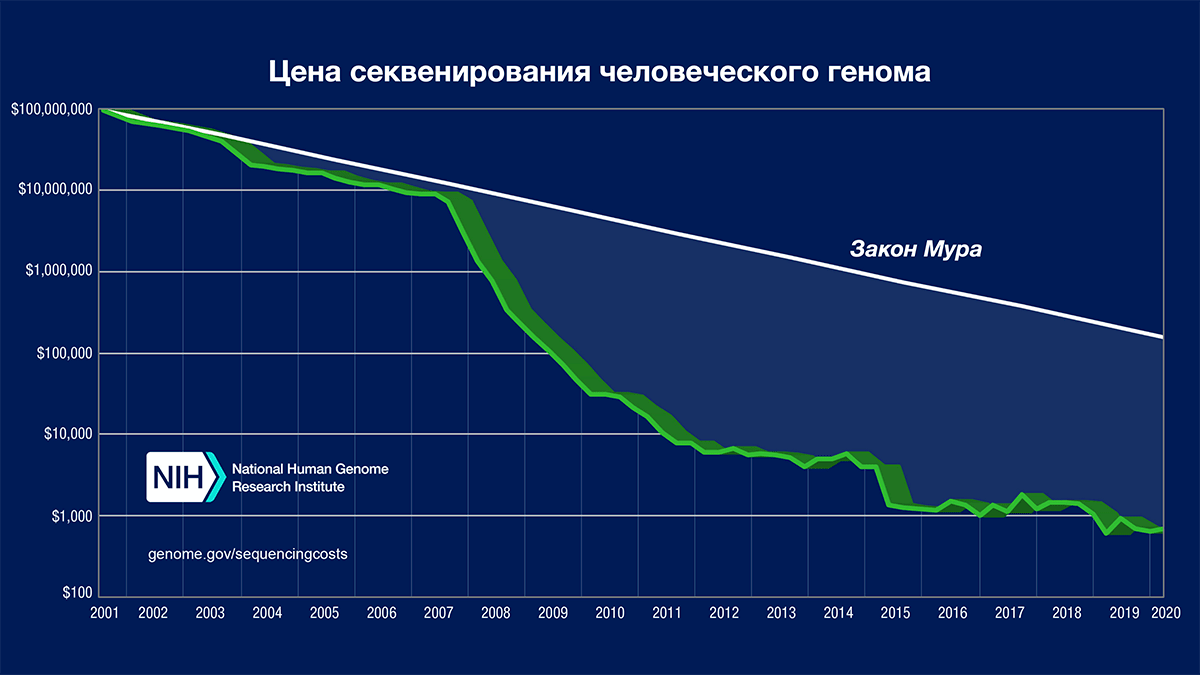
Рисунок 3. График снижения цены секвенирования человеческого генома
Всё это — благодаря новым технологиям секвенирования. Методов секвенирования много, их часто делят на три поколения. Первое — это методы из 60–70-х годов прошлого века. Тогда из множества вариаций широко распространенными стали две. Секвенирование по Максаму—Гилберту было первым распространенным методом. Суть метода — в химической деградации: радиоактивно меченную ДНК обрабатывают реагентами, которые разрывают цепочку по определенным основаниям. После по результатам электрофореза в полиакриламидном геле можно определить длину полученных фрагментов (а значит, и положение определенных нуклеотидов) и определить исходную последовательность. Секвенирование по Сэнгеру оказалось проще, точнее и надежнее, и в итоге стало самым популярным методом на последующие 30 лет. В этой технологии используется принцип обрыва цепи: в реакционную смесь добавляют радиоактивно меченные дидезоксинуклеотиды, которые при включении в синтезируемую цепь останавливают ее синтез. Проведя четыре параллельные реакции с использованием разных дидезоксинуклеотидов и сопоставив результаты в полиакриламидном геле, можно определить исходную последовательность. Метод развивался: на замену радиоактивным меткам пришли флуоресцентные, начал использоваться капиллярный электрофорез, появились автоматические секвенаторы. Именно секвенированием по Сэнгеру был сделан проект «Геном человека». Метод быстрый, менее затратный и более точный, чем NGS, поэтому повсеместно используется и сейчас для секвенирования небольшого количества фрагментов.
Секвенирование второго (нового) поколения (next generation sequencing, NGS) берет начало в 90-х годах XX века и гораздо более производительно по сравнению с секвенированием по Сэнгеру. С его помощью можно считывать миллионы фрагментов одновременно и определять последовательности десятков геномов за один запуск прибора (конечно, в зависимости от их размера). Так, прибор NextSeq 2000 на платформе Illumina — самой популярной на сегодняшний день — способен прочитать 30 образцов небольших геномов по 130 миллионов пар оснований за 29 часов. А прибор от Thermo Fisher Scientific, секвенирующий по Сэнгеру, считывает всего 400–900 пар оснований в образце, зато в зависимости от программы может обработать до 2,1 млн пар оснований (3840 образцов) в день — на один образец уходит всего 36 минут.
Посмотрим, как выглядит подготовка к секвенированию с использованием одного из наборов реагентов — Illumina TruSight HLA v2. Он готовит образцы ДНК к секвенированию и типированию HLA — процедуре, которая необходима, например, для правильного подбора доноров при трансплантации органов. Согласно протоколу, не менее трех с половиной часов уходит на монотонную работу с пипеткой в руках. Процесс состоит из восьми шагов, а это значит, что существует большая вероятность ошибиться. К тому же между этапами работы надо ждать от 12 минут до десяти с половиной часов. Человеку сложно в точности соблюсти все интервалы и уложиться в минимальное время пробоподготовки.
Многофункциональные станции для пробоподготовки к секвенированию сделают всё быстро и точно. На сайте Illumina перечислены решения от разных компаний, совместимые с протоколами Illumina. Среди них, например, станции Biomek от Beckman Coulter. Компания Illumina проверила и может гарантировать точность использования большинства наборов на этих станциях. У станций i-Series (Beckman Coulter) открытая платформа, в них можно интегрировать внешние устройства — например, термоциклер. Так процесс можно сделать полностью автономным и не прерываться на перенос образцов в отдельно стоящий амплификатор и обратно.
Существует и более новое секвенирование так называемого третьего поколения — к нему относятся секвенаторы компании Oxford Nanopore [12]. Суть методов второго поколения — в прочтении и последующем сопоставлении множества коротких фрагментов и их копий, полученных в ходе ПЦР. Секвенирование третьего поколения работает по другому принципу, считывая длинные фрагменты ДНК в реальном времени: десятки и сотни тысяч пар оснований против нескольких сотен при использовании Illumina. При наличии достаточного количества ДНК хорошего качества подготовка к секвенированию происходит без ПЦР: это позволяет детектировать не только последовательность нуклеотидов, но и модификации оснований, а также избежать искажения распределения продуктов ПЦР из-за неравной амплификации. Подготовку к такому секвенированию тоже можно автоматизировать: тот же Oxford Nanopore выпускает компактный прибор VolTRAX, который полностью подготавливает образец к секвенированию.
Подробнее о принципах секвенирования и используемых платформах можно прочитать в обзоре, посвященном секвенированию [6].
Автоматизация нужна на всех этапах работы с генетическим материалом: от подготовки исходного образца до проведения ПЦР и секвенирования. Всё это помогает нам быстрее и подробнее познавать миры и возможности, которые открывает изучение геномики: диагностировать и лечить редкие заболевания, раскрывать преступления и многое-многое другое.
Протеомика: первые шаги автоматизации
Протеомика — высокопроизводительный анализ белков — по сравнению с состоянием методов исследования нуклеиновых кислот находится, без преувеличения, в пубертатном периоде . Виной тому объективные обстоятельства. Полимеры, состоящие из двадцати с лишним элементов — аминокислот, каждая из которых способна к разнообразным природным модификациям, — идентифицировать значительно сложнее, чем нуклеиновые молекулы из четырех нуклеотидов. Аналитические проблемы в протеомике усугубляются низкой чувствительностью, ведь белковые молекулы нельзя амплифицировать подобно полинуклеотидам.
Подробнее о развитии и состоянии протеомики можно прочитать в одноименной статье из цикла «12 методов в картинках» [13].
Для автоматизации всей процедуры анализа белкового состава или какого-то ее этапа нужны веские основания. Вряд ли вы станете покупать роботизированные станции за десятки или сотни тысяч долларов при объемах работы лаборатории, занятой фундаментальными проектами. Пока протеомика только ищет себя в масштабных, воспроизводимых анализах, используемых при разработке лекарств или способов диагностики. Тем не менее постепенно автоматизация проникает и в эту область. Что же там можно автоматизировать?
Конвейер протеомных методов, в общем, состоит из пробоподготовки (выделения из природного образца белков) и их приготовления (например, разделения в пространстве и во времени) для идентификации и количественного анализа. Последние этапы чаще всего осуществляются физическим методом масс-спектрометрии, но иногда — при помощи реагентов со сродством к определенному белку, самые известные из которых — антитела.
Современный вариант аналитического устройства для протеомики — это высокоэффективный хроматограф, сопряженный с масс-спектрометром высокого разрешения (рис. 4). Система в типовом варианте уже роботизирована — там имеется устройство автоматической подачи проб в хроматограф. Но этого явно недостаточно для настоящей автоматизации процесса, ведь предшествующая работа с биологическими образцами остается во многом ручной и штучной. Давайте посмотрим на подходы к автоматизации процесса, которые сопровождали развитие протеомики.
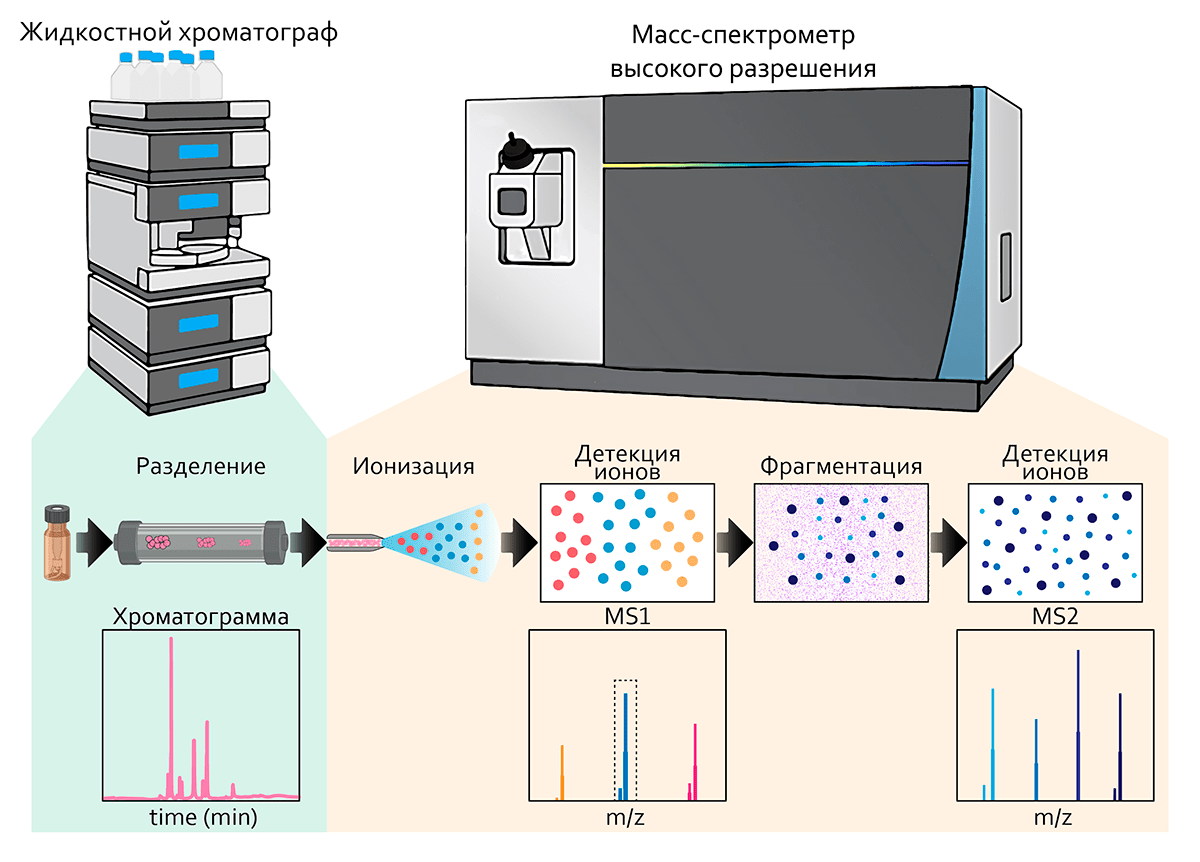
Рисунок 4. Главный метод панорамной (shotgun) протеомики — хроматомасс-спектрометрия высокого разрешения. Белки расщепляют на пептиды, и полученную смесь вводят на колонку для высокоэффективной жидкостной хроматографии. В качестве детектора используют современный масс-спектрометр, в котором исходные пептиды разделяются на фрагменты. По совокупности измеренных значений m/z (отношение молекулярной массы к заряду) исходных пептидов и их фрагментов идентифицируют белки (подробнее в статье спецпроекта «12 методов в картинках» [13]).
рисунок Антона Гончарова
Экскурс в историю: автоматизация двумерного гель-электрофореза
Двумерный гель-электрофорез был главным методом разделения белков на заре развития протеомики [14]. Эта техника позволяет разделить и визуализировать белки сложных образцов с использованием двух независимых свойств этих молекул. Первое измерение разделяет белки в соответствии с их изоэлектрической точкой или, грубо говоря, зарядом молекулы. Этот процесс называется изоэлектрическим фокусированием. Во втором измерении белки разделяются в соответствии с их молекулярной массой широко известным методом денатурирующего электрофореза [15]. Применение двумерного электрофореза позволяет проводить количественный анализ белков и, в ряде случаев, выявлять посттрансляционные модификации.
Сеглдня 2D-электрофорез белков используют редко из-за существенных недостатков метода. Для создания, запуска и анализа одного электрофоретического геля может потребоваться несколько дней и много ручной работы [16]. Тем не менее в начале 2000-х годов, когда этот метод использовали в том числе и для анализа клинических образцов, встал вопрос о создании автоматических высокопроизводительных систем для проведения 2D-электрофореза.
Разработки в области автоматизированного проведения 2D-электрофореза предложили коммерческие компании, такие как NextGen Sciences и Large Scale Biology. Технология a2DEoptimizer, представленная NextGen Sciences в 2004 году, позволяла полностью автоматизировать процесс заливки геля. Нововведение оказалось востребованным, поскольку большинство исследователей тогда перешли на широкоформатные гели (например, 26×20 см) для увеличения разрешения. Заливка таких гелей вручную — процесс весьма трудоемкий, который, к тому же, может приводить к значительной изменчивости получаемых результатов, а предварительно изготовленные коммерческие гели дороги и имеют небольшой срок хранения. a2DEoptimizer проводит процедуру заливки с точностью робота, повышая воспроизводимость результатов.
Панорамная, или скорострельная протеомика, которая использует для разделения фрагментов белков высокоэффективную жидкостную хроматографию в сочетании с масс-спектрометрией, вытеснила 2D-электрофорез из широкого применения. Подходы к автоматизации этого процесса, в целом, ушли в прошлое.
Подробнее о разделении биомолекул читайте в специальной статье из цикла цикла «12 методов в картинках» [4].
Автоматизация пробоподготовки для масс-спектрометрии
Масс-спектрометрия стала для протеомики мощным подходом для качественного и количественного анализа белков, их посттрансляционных модификаций, взаимодействий и многих других происходящих с ними явлений [17]. Стремление протеомики стать инструментом доказательной медицины повышает требования не только к производительности, но и к воспроизводимости. Например, представлены эффективные алгоритмы по протеомному анализу плазмы крови человека в норме и при заболеваниях [18]. Совершенствуются направленные на производительность способы жидкостной хроматографии с масс-спектрометрией (LC-MS), которые, как показано, могут надежно анализировать на одном устройстве более 50 образцов в день [19]. Помимо разведочного анализа — когда требуется оценить содержание белков в образце широким взглядом, — масс-спектрометрию научились применять направленно, измеряя в образце концентрации нескольких конкретных белков [20]. В формате направленного мониторинга выбранных реакций (от англ. selected reaction monitoring, SRM) масс-спектрометрическая протеомика конкурирует с иммунными методами количественного анализа белков. Как и при этих методах, направленный подход анализирует заведомо известные белки и подходит для масштабирования. Известны первые тесты на основе SRM белков, которые доводятся до клинического применения.
Если хроматомасс-спектрометрия, которую используют в протеомных методах, во многом автоматизирована, то прогресс в роботизации пробоподготовки образцов — критически важном этапе протеомного анализа — отстает. Подготовка образца для наиболее распространенного сейчас вида протеомного анализа представляет собой многоступенчатый процесс (рис. 5).
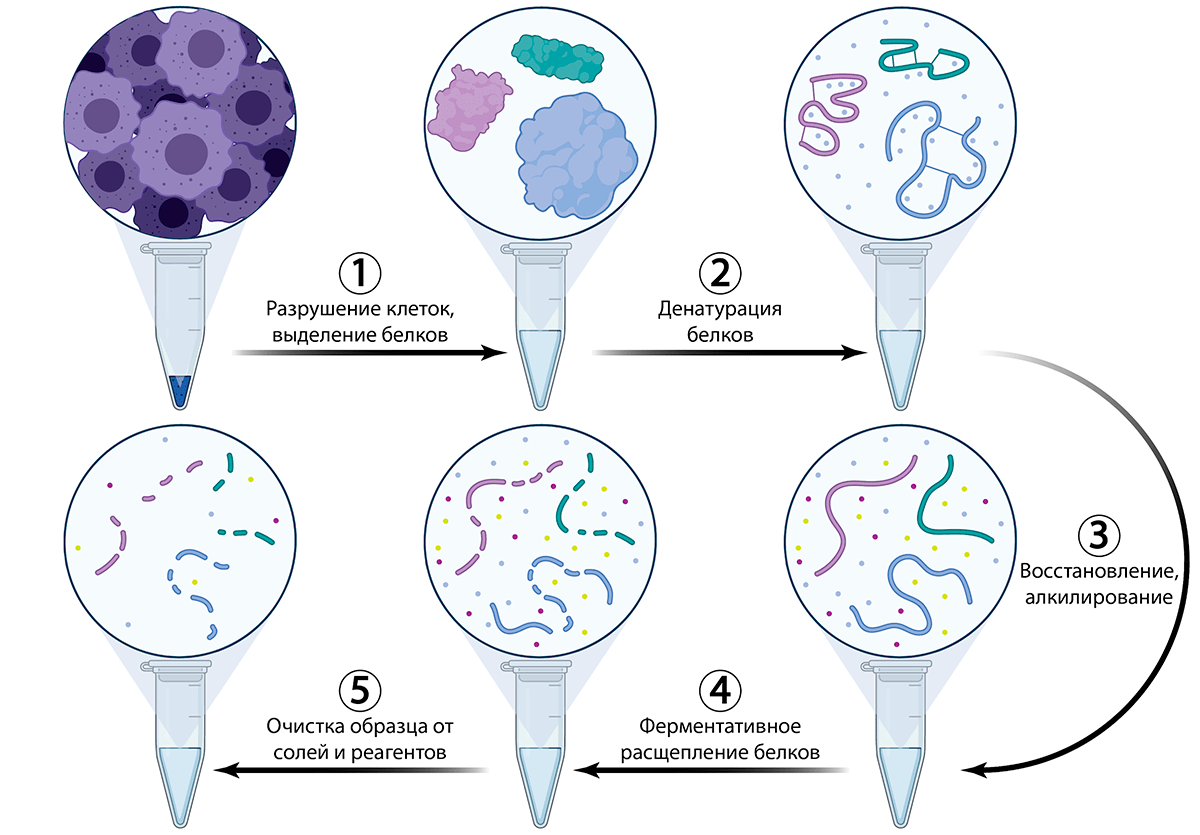
Рисунок 5. Подготовка образца к хроматомасс-спектрометрии. Разрушение (лизис) клеток — первый шаг практически всех биохимических экспериментов (1). На данный момент существует множество методов для различных организмов, типов образцов (клеток или тканей), субклеточных структур или конкретных белков [4]. Выделенные белки затем денатурируют сильным детергентами, такими как додецилсульфат натрия (SDS), или хаотропными агентами, такими как мочевина или тиомочевина (2). Стадия денатурации сопровождается также восстановлением дисульфидных связей, образованных остатками цистеина. Это происходит в присутствии восстанавливающего агента, такого как трис-(2-карбоксиэтил)-фосфин (TCEP) или дитиотреитол (DTT). Свободные сульфгидрильные группы на остатках цистеина затем алкилируются реагентами, такими как йодацетамид или йодауксусная кислота, чтобы необратимо препятствовать превращению свободных сульфгидрилов в дисульфидные связи (3). Затем денатурированные, восстановленные и алкилированные белки расщепляются на пептиды специфическими эндопротеазами (например, трипсином, химотрипсином, Arg-C и Lys-C), которые гидролитически разрывают пептидные связи (4). Зачастую, для успешного анализа полученную смесь необходимо очистить от коктейля использованных реагентов, которые могут помешать работе хроматографа и/или масс-спектрометра.
Сейчас большинство исследователей проводит этапы пробоподготовки в ручном режиме. С некоторыми видами образцов (например, с плотными тканями) реализовать эти этапы сложно. Так, с трудом поддается автоматизации использование центрифуг, а особенно гомогенизаторов. Естественно, при наличии большого количества многоэтапных процедур и частого переноса жидкостей ручными дозаторами, возникает изменчивость между образцами, из-за чего страдает воспроизводимость.
Тем не менее использование в протеомике роботизированных рабочих станций позволило автоматизировать процесс пробоподготовки. При должной настройке они способны автоматически проводить денатурацию, восстановление, алкилирование и расщепление белков. Помимо этого, если в качестве материала для исследования используются однородные образцы, например плазма крови или спинномозговая жидкость, процесс пробоподготовки можно проводить с минимальным вмешательством оператора.
Геномика и протеомика единичных клеток — без автоматизации не обойтись
Прорыв в биологии последних лет — полногеномный молекулярный анализ единичных клеток [21]. В первую очередь, двигателем этого направления стало секвенирование нового поколения для анализа транскриптомов [6]. Давно известны способы дезинтеграции живых тканей с сохранением целостности клеток, да и разделение их в пространстве — уже освоенная технология .
О развитии сортировки клеток подробнее можно узнать в статье о проточной цитометрии из цикла цикла «12 методов в картинках» [22].
Получить большое количество ДНК или РНК из единичной клетки тоже возможно, поскольку в анализе нуклеиновых кислот используется сверхчувствительные методы амплификации [5]. Когда секвенаторы стали настолько эффективными, что в анализе одного образца стало возможным получить сотни и даже тысячи индивидуальных геномов или транскриптомов, появилась, с позволения сказать, «транскриптомная гистология». В этих исследованиях клеточные типы выделяли не с помощью микроскопии, как это делали несколько веков до этого, а по различию в полногеномном профиле экспрессии, то есть транскриптоме. Так в последние годы были заново «препарированы» многие клеточные процессы (например, различные стадии онтогенеза многих модельных организмов и их отдельных органов) и обнаружены новые, функционально значимые клеточные типы.
Геномика единичных клеток
Секвенирование ДНК или РНК единичных клеток от «обычного» отличается шагами, которые необходимо сделать на стадии пробоподготовки. Клетки необходимо не просто выделить из органа или ткани, но и отделить друг от друга, а уже затем амплифицировать весь геном или интересующий регион отдельно для каждой клетки, создать библиотеки для секвенирования и только потом считать их.
Секвенирование ДНК единичных клеток (scDNAseq) позволяет охарактеризовать состав микробиома, не культивируя отдельные микроорганизмы [23], а также активно используется в онкологии [24]: можно точнее оценить прогрессирование заболевания или ответ на терапию, смотря на геномные особенности отдельных клеток, а не их множества. В случае с гетерогенными опухолями врача-исследователя может интересовать минорная популяция опухолевых клеток: при обычном анализе без предварительного отбора их будет невозможно идентифицировать в совокупности получаемых в результате секвенирования данных!
Возрастающей популярностью сортинг и исследования единичных клеток «обязаны» секвенированию нового поколения для анализа транскриптомов [6]: количество публикаций об исследованиях единичных клеток, в том числе секвенировании РНК, только за последние два года выросло в шесть раз, по информации одного из лидеров этого рынка — компании 10х Genomics. Секвенирование РНК единичных клеток (scRNA-seq) стало золотым стандартом характеризации клеточных фенотипов [25] (рис. 6) и даже приводит к обнаружению новых, ранее неизвестных типов клеток. Так, в 2018 году был открыт особый подвид клеток респираторного эпителия — ионоциты, — которые, предположительно, отвечают за вязкость слизи [26].
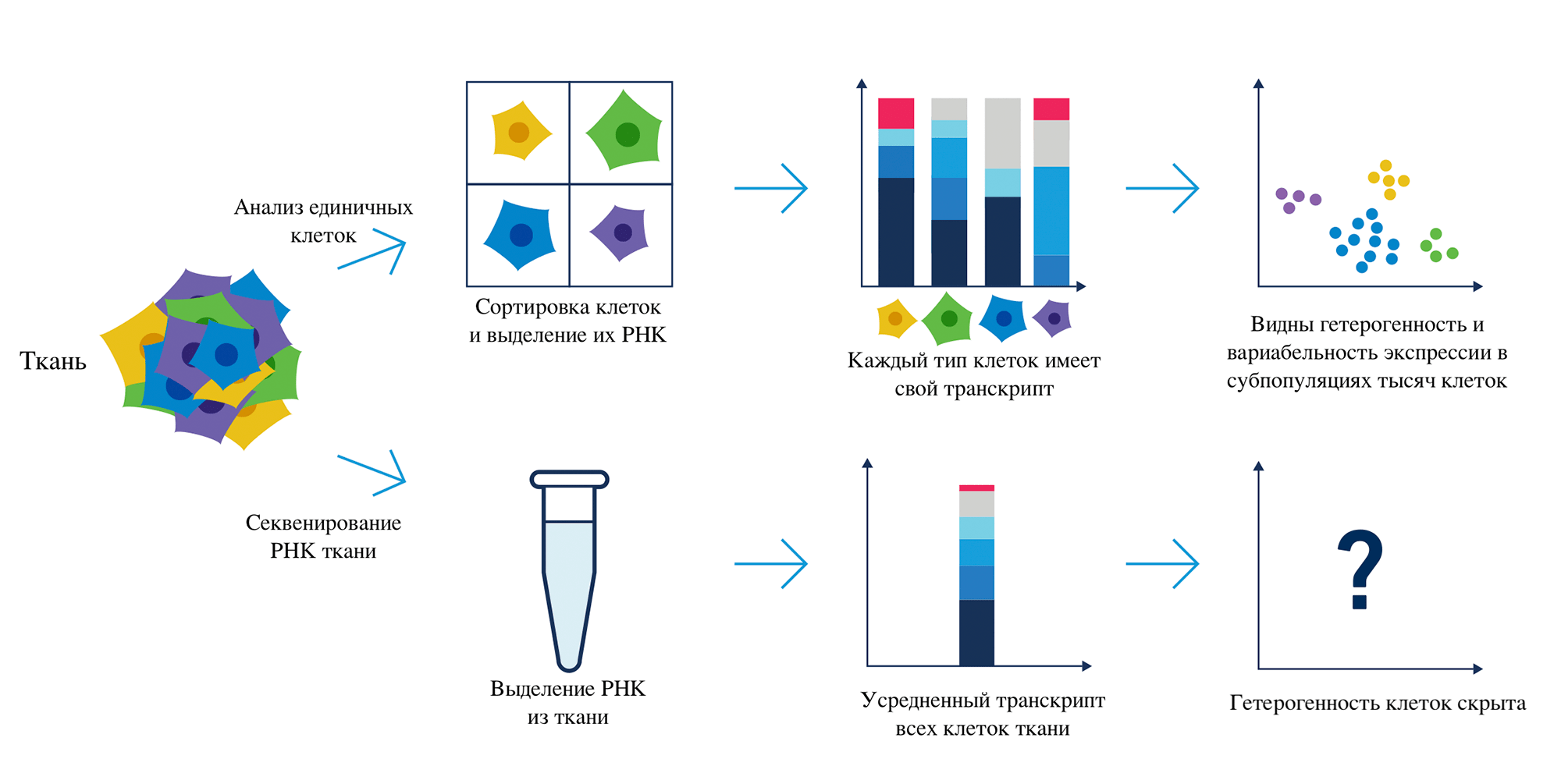
Рисунок 6. Работа с РНК единичной клетки отличается от обычного выделения РНК из ткани. Клетки сначала сортируют, чтобы затем получить не «усредненный» транскрипт из всех клеток в образце (сверху), а по отдельному транскрипту для каждой из проанализированных клеток (снизу). При последующем анализе данных можно выделить субпопуляции и изучить различия в экспрессии между разными типами клеток. Работа с РНК единичных клеток позволяет не только идентифицировать и охарактеризовать все типы клеток в гетерогенных клеточных популяциях (и даже их происхождение!), но и обнаружить новые маркеры или сигнальные пути: часть этой информации уже используется в разработке новых скринингов для оценки здоровья конкретного органа или ткани организма.
Итак, давайте рассмотрим основу работы с единичными клетками, то есть методы их сортировки и выделения, от общих принципов к самым последним технологиям, сочетающим в себе несколько подходов.
Если цель выделения — желание работать с клетками с определенными молекулярными характеристиками (например, наличие какого-то рецептора или одна и та же стадия развития), то отобрать их можно, основываясь на размере, морфологии и синтезе нужных белков. Для этого активно используются флуоресцентные красители (например, ДНК-связывающие красители для выделения пролиферирующих клеток) или флуоресцентно меченные антитела, специфичные к белку, маркирующему нужную популяцию клеток. После этого клетки, соответствующие заданным параметрам, например, имеющие интересующие рецепторы, могут быть отделены от тех, которые их не имеют. Но как именно это сделать?
Методы выделения единичных клеток можно разделить на низко- и высокопроизводительные (рис. 7). К первым относятся микропипетирование, аспирация цитоплазмы и лазерная захватывающая микродиссекция (laser capture microdissection, LCM). Все три метода подразумевают предварительное исследование образца под микроскопом: это могут быть как бактерии, продуцирующие флуоресцентный белок на чашке Петри, или культуры эукариотических клеток, так и цитологические образцы. Использование специальных мембран и лазеров в случае LCM позволило разработать несколько техник выделения, позволяющих не касаться клеток наконечником пипетки: такая пробоподготовка не влияет на последующие результаты анализа нуклеиновых кислот или белков. Существенным минусом этой группы методов является низкая скорость и малое количество образцов — не более нескольких сотен отобранных клеток.
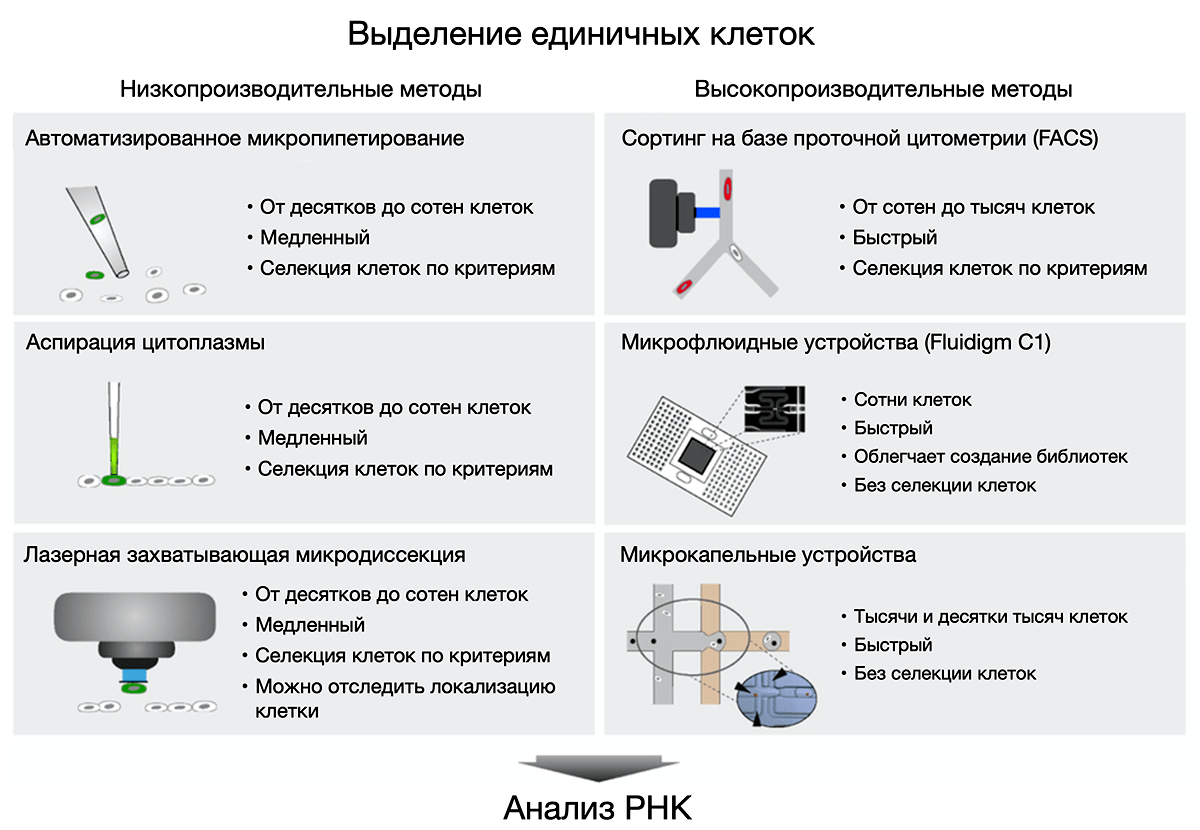
Рисунок 7. Методы выделения единичных клеток. Низкопроизводительные методы (слева) позволяют выделить не более тысячи клеток, но, с другой стороны, позволяют выбирать клетки, соответствующие необходимым критериям: продуцирующие флуоресцентный белок, имеющие нужные рецепторы или размер. Высокопроизводительные методы (справа) дают возможность выделять тысячи клеток, но только проточная цитофлуориметрия (fluorescence activated cell sorting, FACS) позволяет еще и сортировать их по заданным параметрам. В случае микрофлюидных устройств сортинг нужно проводить заранее.
Высокопроизводительные методы, позволяющие получить сотни, тысячи и даже десятки тысяч клеток, можно разделить еще на две группы: те, в которых используются флуоресцентные метки для выбора клеток по определенным маркерам, и те, где клетки разделяются благодаря малым объемам дозирования. К первым относится сортинг на базе проточной цитометрии , а ко вторым — микрофлюидные устройства или устройства, работающие по принципу создания микрокапель [27]. Разделение происходит на микрофлюидных чипах: под каждую задачу можно создавать свой, наиболее подходящий чип, при этом стоимость производства уже достаточно невысокая.
О принципах проточной цитометрии и сортировке клеток можно узнать в статье «12 методов в картинках: проточная цитофлуориметрия» [22].
На микрофлюидных технологиях стоит остановиться подробнее: они сейчас стремительно развиваются и очень популярны. Причина проста: работа с малыми объемами позволяет не только сократить количество расходуемых реагентов и создавать компактные устройства, но и существенно экономить время, затрачиваемое на проведение реакций. Первые устройства помещали каждую клетку в отдельную ячейку плашки, но сейчас большее распространение получили микрофлюидные технологии, когда каждая клетка находится в отдельной капле. В случае с секвенированием единичных клеток это значит, что в этой же капле затем происходит обратная транскрипция, позволяющая синтезировать с каждой отдельной РНК соответствующую ей кДНК. Но так как каждая капля изначально содержит не только реагенты для обратной транскрипции, но и индивидуальный штрих-код (англ. barcode) в виде нуклеотидной последовательности [28], то получающаяся в результате кДНК имеет однозначную метку. Это дает возможность смешать все кДНК из разных клеток для проведения секвенирования, но при этом сохранить возможность затем идентифицировать конкретный транскрипт при помощи заранее добавленного штрих-кода.
Современные реалии нередко подразумевают объединение технологий: один прибор может сначала отсортировать клетки на основе FACS, затем разделить выбранные на микрокапли, провести в каждой из них необходимые реакции и осуществить секвенирование. За счет использования 96-луночных планшетов или чипов удается увеличить скорость анализа, уменьшив при этом размер устройств и расход реагентов. Основным минусом, как правило, остается цена — всё новое и передовое стоит недешево.
Перейдем от теории к практике и рассмотрим подробнее систему компании 10х Genomics для анализа транскриптомов единичных клеток.
Система 10x Genomics для геномики и транскриптомики единичных клеток
В общем виде протоколы для scRNA-seq включают выделение единичных клеток и их РНК, а затем следуют всё те же шаги, что и при работе с РНК, выделенной из всех клеток образца: обратная транскрипция, амплификация, создание библиотек и непосредственно секвенирование.
В платформе от компании 10x Genomics для шагов, предшествующих секвенированию, используется упомянутый выше принцип микрокапель. Микрофлюидное устройство выстраивает в потоке жидкости по одному специальные гелевые шарики, несущие олигонуклеотидные «штрих-коды» и последовательности, распознающие РНК, — чаще всего это общие последовательности poly(T), распознающие poly(A) хвосты на 3′-концах первичной мРНК (рис. 8). К каждой из сфер добавляется по одной клетке из образца и необходимые для обратной транскрипции ферменты, после чего эта смесь попадает в специальную масляную среду. Так образуется GEM-частица (англ. Gel Bead-in-emulsion), в которой происходит лизис и обратная транскрипция РНК единичной клетки. Эти шаги осуществляются внутри микрофлюидного чипа, помещаемого в платформу Chromium Controller. Полученная кДНК может быть объединена для секвенирования нового поколения, например, на секвенаторах Illumina, а итоговые данные проанализированы специальным программным обеспечением, которое идентифицирует каждый транскрипт с помощью олигонуклеотидного штрихода.
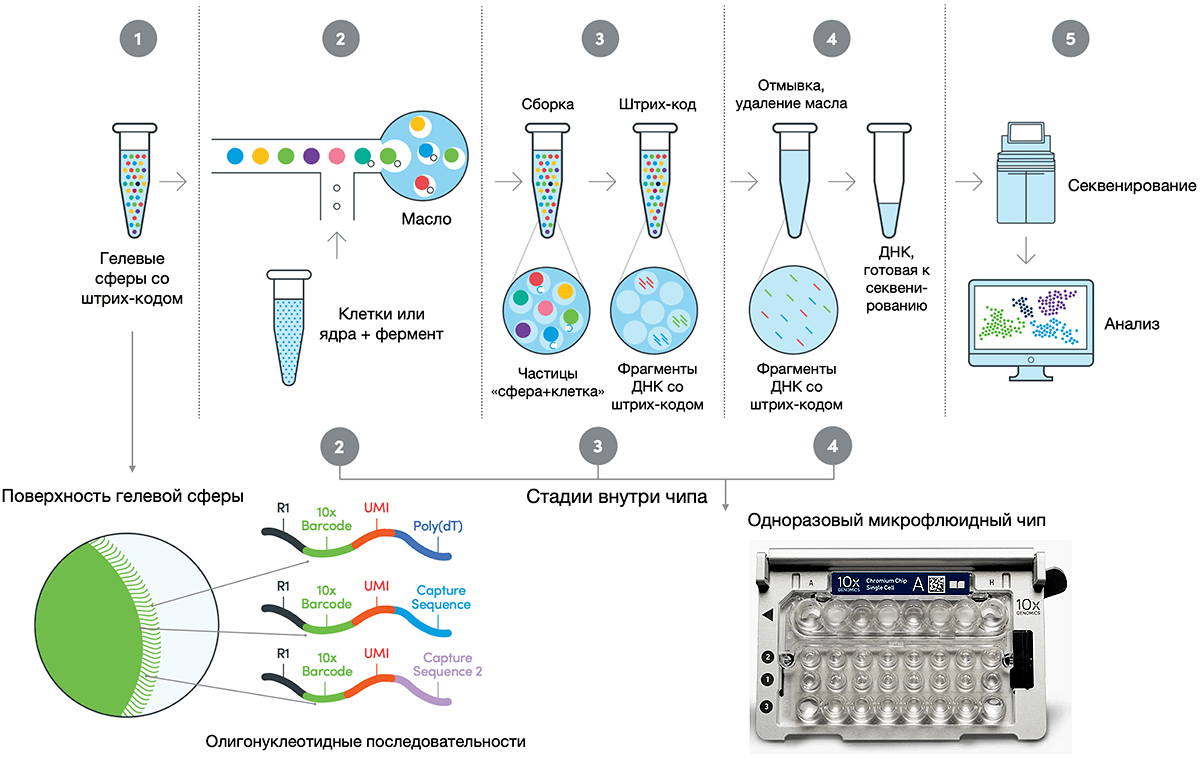
Рисунок 8. Схема анализа транскриптома единичных клеток в системе 10х Genomics. Гелевая сфера, к поверхности которой прикреплены олигонуклеотидные последовательности с идентификационными фрагментами (штрих-коды, англ. barcodes), образует частицу с клетками и реагентами для обратной транскрипции, стабильную в масляном растворе. После прохождения обратной транскрипции каждая из частиц содержит кДНК с уникальным штрих-кодом — все кДНК могут быть объединены для сборки библиотек и секвенирования, а затем разделены и идентифицированы при помощи программного обеспечения для анализа.
Трудности в секвенировании единичных клеток
Описанные решения для сортинга единичных клеток и их последующего анализа весьма непросты, и у них существует ряд сложностей. Одна из них очевидна: чрезвычайно малое количество материала, которое можно получить из одной клетки, поэтому не всегда легко сказать, являются ли обнаруженные генетические различия между клетками случайностью или закономерностью, особенно когда речь идет об однонуклеотидных полиморфизмах. С целью увеличения количества исходного материала может быть проведена дополнительная неспецифическая амплификация, но одни регионы генома могут амплифицироваться сильнее, чем другие, что исказит итоговые результаты. Кроме того, сложно работать и с последовательностями, богатыми GC-повторами, что особенно актуально при анализе бактерий. Эти трудности решаемы, но предполагают изменение стандартных протоколов — например, использование термостабильных полимераз, чтобы уменьшить искажения результатов от GC-повторов в геноме [29].
scRNAseq имеет свои специфические ограничения, связанные с необходимостью проведения обратной транскрипции — от выбора праймеров и эффективности обратной транскриптазы зависит, какая кДНК будет синтезирована для дальнейшего анализа. Неправильный выбор условий на этой стадии может привести к созданию библиотек, смещенных в сторону 3′- или 5′-конца относительно реального транскриптома. Кроме того, в большинстве случаев для анализа используется мРНК — ее удобно выделить для дальнейшей работы благодаря полиаденилированию, как в рассмотренном выше примере системы 10х Genomics. Однако в этом случае некодирующие РНК и микроРНК не попадают в проводимый транскриптомный анализ. По этой же причине возникают сложности с анализом единичных бактерий: у них полиаденилированной мРНК просто недостаточно. Тем не менее наука не стоит на месте — вероятно, вскоре мы увидим решение и этих проблем.
Протеомика единичных клеток
Протеомика, как уже не раз указывалось, это бедная родственница геномики, поскольку она вынуждена довольствоваться для анализа тем количеством молекул, которые находятся в образце — белки, как известно, амплифицировать нельзя. Однако современная масс-спектрометрия обладает очень высокой чувствительностью — в коммерчески доступном детекторе может быть зарегистрировано от нескольких тысяч пептидных молекул. Проблема в том, что их нужно туда доставить. Все этапы доведения биомолекул до вакуумной ячейки масс-спектрометра сопровождаются большими потерями.
Тем не менее и протеомика пытается усилиями нескольких научных групп энергично войти в тематику анализа единичных клеток . Конечно, для этого приходится идти на существенные методические ухищрения.
Подробнее о протеомике единичных клеток можно узнать, прочитав мини-обзор, недавно вышедший на русском языке в журнале «Биохимия» [30].
Разрыв между продемонстрированной приборами аналитической чувствительностью и реальным анализом обусловлен ограничениями в пробоподготовке. Потери случаются на каждом этапе — при выделении белков, их расщеплении протеазами на короткие пептиды (это необходимо для большей части аналитических подходов), очистке и доставке пептидов в масс-спектрометр.
По мере уменьшения размера образца до одной клетки возникает много сложностей с его подготовкой, ведь теперь исследуемых компонентов в нем совсем немного. Проблемой становится неспецифическая адсорбция белков и пептидов на поверхности реакционных сосудов, падает эффективность протеолитического расщепления. Минимальный объем реакционной смеси зачастую ограничен интенсивностью испарения и операционными объемами использующихся дозаторов (пипеток или автоматических раскапывающих станций). Усилия по совершенствованию процедур пробоподготовки включают использование пробирок с низкой адсорбционной способностью [31], и использование протоколов расщепления «в одной пробирке» (one-pot) для ограничения общего взаимодействия образца с поверхностью [32], [33]. Кроме того, для анализа минимальных количеств образца использовали экстракцию и денатурацию белков на основе трифторэтанола [32], пробоподготовку на фильтрах [34], совместимые с масс-спектрометрией поверхностно-активные вещества [35], [36], высокотемпературный трипсиновый гидролиз [33], адаптивно сфокусированную экстракцию белков с акустической поддержкой [37] и иммобилизованные протоколы расщепления [34], что обеспечило достижение определенных успехов в обработке небольших количеств образца.
Использование этих подходов при анализе образцов, содержащих около 100 клеток, позволило идентифицировать и количественно оценить несколько сотен — до тысячи — белков [33], [38]. Предтечей современных результатов в протеомике единичных клеток стал анализ относительно крупных клеток — отдельных бластомеров, выделенных из эмбрионов шпорцевой лягушки Xenopus laevis [39], [40]. Отчасти, это оказалось возможным из-за того, что каждый такой бластомер содержит микрограммы белка, по сравнению с ≈0,1 нг белка, обнаруживаемого в типичных клетках млекопитающих.
Значительные успехи в пробоподготовке наноразмерных биологических образцов, содержащих нанограммовые количества белка, достигнуты благодаря использованию микрофлюидных чипов и роботизации процесса. Одна из таких платформ, названная nanoPOTS (Nanodroplet Processing in One pot for Trace Samples), уменьшает общий объем реакционной смеси с привычных сотен микролитров до 200 нл в одной капле. Недавно было показано, что в сочетании с хроматомасс-спектрометрией высокого разрешения, платформа nanoPOTS позволяет идентифицировать от 1500 до 3000 белков в образцах от 10 до 140 человеческих клеток обычного размера [41]. Такой уровень покрытия протеома ранее достигался только для нескольких тысяч клеток в одном образце. На данный момент, nanoPOTS позволяет идентифицировать более 1000 белковых групп при анализе одной клетки линии HeLa с использованием специально адаптированных методов масс-спектрометрии [42].
Система nanoPOTS для протеомики единичных клеток
nanoPOTS — это уникальная научная разработка, которая создана одним коллективом. Чипы nanoPOTS изготавливаются из обычного предметного стекла. С помощью фотолитографии на его поверхности формируется основа чипа — окруженные гидрофобной областью гидрофильные пьедесталы, которые служат реакционными сосудами для наноразмерных капель. В этих каплях происходит многоступенчатый процесс пробоподготовки образцов. Сверху чип герметизируется с помощью стекла с мембранным покрытием для минимизации испарения содержимого нанопокрытия во время различных этапов инкубации. Стеклянная подложка обеспечивает возможность микроскопической визуализации образцов и сводит к минимуму адсорбцию белков и пептидов по сравнению со многими другими материалами благодаря своей гидрофильности и уменьшенному поверхностному заряду при низком pH [43]. Пьедесталы nanoPOTS дополнительно уменьшают контакт с поверхностью по сравнению с использованием вогнутых лунок.
Для распределения клеток и жидких реагентов в nanoPOTS используется роботизированная платформа DropLab, обладающая субмикронной (<10−6 м) точностью позиционирования и способная работать с пиколитровыми (10−12 л) объемами жидкостей. DropLab состоит из четырех частей, включая трехмерную передвижную платформу для автоматизированного управления положением капилляра, самодельный высокоточный шприцевой насос для дозирования жидкости, микроскопическую камеру для контроля процесса дозирования и конусообразный капиллярный зонд для дозирования жидкости. Капиллярный зонд изготавливается нагреванием и вытягиванием кварцевого капилляра с внутренним диаметром 200 мкм для получения конического наконечника с внутренним диаметром 30 мкм. Программное обеспечение для всей роботизированной системы было создано в научном коллективе, которому принадлежит разработка. Чтобы свести к минимуму испарение на всех этапах пробоподготовки, система заключается в изготовленную из лексана камеру, внутри которой поддерживается относительная влажность 95%.
Процесс пробоподготовки проходит по адаптированному протоколу «в одной пробирке» (one-pot). В целом, все этапы повторяют процесс обычной пробоподготовки образцов к масс-спектрометрическому анализу. Вкратце, после переноса образцов в каждую ячейку чипа, с помощью микроскопической визуализации проводится количественная оценка — подсчитывается количество клеток, размер ткани и так далее. Затем образцы лизируют и проводят протеолитическое расщепление белков более или менее стандартным способом с поправкой на то, что реагенты на чип наносятся прецизионным роботом в маленьких объемах. На время длительных инкубаций поверхность чипа закрывается для минимизации испарения с поверхности капель.
На платформе nanoPOTS вся процедура пробоподготовки проводится в капле объемом 200 нл, которая по сути является реакционным сосудом без стенок (рис. 9). При диаметре капли 1 мм, общая площадь соприкосновения жидкости со стеклянной поверхностью составляет ≈0,8 мм2. По сравнению с обычной пробоподготовкой в пробирке объемом 0,5 мл, в которой площадь поверхности составляет ≈130 мм2, такое значительное уменьшение взаимодействия образца с сосудом (более чем на 99%) позволяет существенно уменьшить потери материала из-за адсорбции. Кроме того, благодаря сохранению оптимального соотношения белка и протеаз при использовании nanoPOTS [44], степень гидролитического расщепления потенциально увеличивается по сравнению с обычной пробоподготовкой образца стандартного объема для того же числа клеток.

Рисунок 9. Миниатюрность системы nanoPOTS. а — Фотография рабочей поверхности чипа nanoPOTS с нанокаплями на поверхности. Каждая капля содержит 200 нл красителя для визуализации. Для сравнения приложена монета в 25 центов. б — Фотография одной капли с единичным островком поджелудочной железы внутри.
[41], с изменениями
Использование nanoPOTS позволяет получить около 200 пикограммов пептидов в капле объемом 200 нл от единичной клетки HeLa. Но несмотря на то, что эта система эффективно решает проблемы, связанные с подготовкой образца, она несовместима с современными автоматизированными системами LC-MS из-за крайне малых объемов жидкости в образцах. Большинство имеющихся в продаже устройств для автоматической подачи проб способны забирать пробы только из обычных флаконов или планшетов, содержащих не менее 5 мкл образца, и, таким образом, не подходят для образцов, генерируемых nanoPOTS. Следовательно, каждый образец ранее должен был вводиться в хроматограф вручную, путем аспирации нанокапель в специальный капилляр. Затем капилляр соединялся с колонкой твердофазной экстракции — по сути, миниатюризованной хроматографической колонкой [4], в которой происходили очистка и концентрирование образца. Наконец, образец с колонки элюировали в классическую аналитическую колонку и после разделения на ней анализировали в масс-спектрометре. Такие манипуляции трудоемки и ограничивали производительность до шести анализов (то есть отдельных клеток) в день. Поэтому для ускорения процесса коллективу, разработавший систему nanoPOTS, пришлось разработать специальное устройство для интеграции пробоподготовки образца в nanoPOTS и хроматомасс-спектрометрического анализа [45].
Протеомика стремится к автоматизации большинства этапов анализа в двух основных направлениях. Первое — стремление поставить на поток работу с клиническим материалом. Второе — находящийся на переднем крае науки и техники анализ единичных клеток. Оба направления энергично развиваются, но остаются на данный момент уделом научных лабораторий и, изредка, стартапов. По объективным причинам (основная из которых — проблема чувствительности из-за отсутствия амплификации белков) протеомный анализ не настолько успешен в практических областях по сравнению с секвенированием нуклеиновых кислот. Но это не отменяет потребность в высокоэффективном анализе белков — мишеней для лекарств, вакцин, важных биотехнологических продуктов. Поэтому в ближайшее время стоит ожидать новых технических решений для автоматизации протеомного анализа.
* * *
Автоматизация омиксных технологий движется семимильными шагами и касается не только пробоподготовки, но и, в случае геномики, непосредственно анализа полученного материала. Чем сложнее система, тем большей автоматизации она подвергается. Так, работа с единичными клетками целиком построена на применении высокотехнологичного оборудования, что позволяет выполнять высокоточные процедуры и существенно снизить риск ошибки. В одной из будущих статей спецпроекта мы поговорим о том, как автоматизированные омиксные технологии внедряются в современную фармацевтику — например, в драг-дизайн.
Литература
- Кто все эти люди?!;
- Генофонд австралийских аборигенов хранит ключ к тайне выхода человека из Африки;
- Скандалы, интриги, расследования: с какими гоминидами спали наши предки?;
- 12 методов в картинках: очистка молекул и разделение смесей;
- 12 методов в картинках: полимеразная цепная реакция;
- 12 методов в картинках: секвенирование нуклеиновых кислот;
- Андреева А. (2019). Топ-5 современных методик выделения нуклеиновых кислот. PCR.news;
- Nicholas M. Adams, Hali Bordelon, Kwo-Kwang A. Wang, Laura E. Albert, David W. Wright, Frederick R. Haselton. (2015). Comparison of Three Magnetic Bead Surface Functionalities for RNA Extraction and Detection. ACS Appl. Mater. Interfaces. 7, 6062-6069;
- Геном человека: как это было и как это будет;
- Технология: $1000 за геном;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Нанопоровое секвенирование: на пороге третьей геномной революции;
- 12 методов в картинках: протеомика;
- Thierry Rabilloud, Mireille Chevallet, Sylvie Luche, Cécile Lelong. (2010). Two-dimensional gel electrophoresis in proteomics: Past, present and future. Journal of Proteomics. 73, 2064-2077;
- Thierry Rabilloud, Cécile Lelong. (2011). Two-dimensional gel electrophoresis in proteomics: A tutorial. Journal of Proteomics. 74, 1829-1841;
- Julia M. Hille, Anita L. Freed, Hermann Wätzig. (2001). Possibilities to improve automation, speed and precision of proteome analysis: A comparison of two-dimensional electrophoresis and alternatives. Electrophoresis. 22, 4035-4052;
- Ruedi Aebersold, Matthias Mann. (2003). Mass spectrometry-based proteomics. Nature. 422, 198-207;
- Philipp E Geyer, Lesca M Holdt, Daniel Teupser, Matthias Mann. (2017). Revisiting biomarker discovery by plasma proteomics. Mol Syst Biol. 13, 942;
- Yangyang Bian, Runsheng Zheng, Florian P. Bayer, Cassandra Wong, Yun-Chien Chang, et. al.. (2020). Robust, reproducible and quantitative analysis of thousands of proteomes by micro-flow LC–MS/MS. Nat Commun. 11;
- Andrew J Percy, Yassene Mohammed, Juncong Yang, Christoph H Borchers. (2015). A standardized kit for automated quantitative assessment of candidate protein biomarkers in human plasma. Bioanalysis. 7, 2991-3004;
- Секвенирование единичных клеток (версия — Metazoa);
- 12 методов в картинках: проточная цитофлуориметрия;
- Johannes Alneberg, Christofer M. G. Karlsson, Anna-Maria Divne, Claudia Bergin, Felix Homa, et. al.. (2018). Genomes from uncultivated prokaryotes: a comparison of metagenome-assembled and single-amplified genomes. Microbiome. 6;
- Charles Gawad, Winston Koh, Stephen R. Quake. (2016). Single-cell genome sequencing: current state of the science. Nat Rev Genet. 17, 175-188;
- Tuomas Tammela, Julien Sage. (2020). Investigating Tumor Heterogeneity in Mouse Models. Annu. Rev. Cancer Biol.. 4, 99-119;
- Lindsey W. Plasschaert, Rapolas Žilionis, Rayman Choo-Wing, Virginia Savova, Judith Knehr, et. al.. (2018). A single-cell atlas of the airway epithelium reveals the CFTR-rich pulmonary ionocyte. Nature. 560, 377-381;
- Pallavi Shinde, Loganathan Mohan, Amogh Kumar, Koyel Dey, Anjali Maddi, et. al.. (2018). Current Trends of Microfluidic Single-Cell Technologies. IJMS. 19, 3143;
- Allon M. Klein, Linas Mazutis, Ilke Akartuna, Naren Tallapragada, Adrian Veres, et. al.. (2015). Droplet Barcoding for Single-Cell Transcriptomics Applied to Embryonic Stem Cells. Cell. 161, 1187-1201;
- Ramunas Stepanauskas, Elizabeth A. Fergusson, Joseph Brown, Nicole J. Poulton, Ben Tupper, et. al.. (2017). Improved genome recovery and integrated cell-size analyses of individual uncultured microbial cells and viral particles. Nat Commun. 8;
- Мошковский С.А., Лобас А.А., Горшков М.В. (2020). Протеогеномика единичных клеток — ближайшая перспектива. «Биохимия». 2, 165–173;
- Hugo Marcel Johan Vanderstichele, Shorena Janelidze, Leentje Demeyer, Els Coart, Erik Stoops, et. al.. (2016). Optimized Standard Operating Procedures for the Analysis of Cerebrospinal Fluid Aβ42 and the Ratios of Aβ Isoforms Using Low Protein Binding Tubes. JAD. 53, 1121-1132;
- Haixing Wang, Wei-Jun Qian, Heather M. Mottaz, Therese R. W. Clauss, David J. Anderson, et. al.. (2005). Development and Evaluation of a Micro- and Nanoscale Proteomic Sample Preparation Method. J. Proteome Res.. 4, 2397-2403;
- Qi Chen, Guoquan Yan, Mingxia Gao, Xiangmin Zhang. (2015). Ultrasensitive Proteome Profiling for 100 Living Cells by Direct Cell Injection, Online Digestion and Nano-LC-MS/MS Analysis. Anal. Chem.. 87, 6674-6680;
- Jacek R. Wiśniewski, Pawel Ostasiewicz, Matthias Mann. (2011). High Recovery FASP Applied to the Proteomic Analysis of Microdissected Formalin Fixed Paraffin Embedded Cancer Tissues Retrieves Known Colon Cancer Markers. J. Proteome Res.. 10, 3040-3049;
- Wendong Chen, Shuai Wang, Subash Adhikari, Zuhui Deng, Lingjue Wang, et. al.. (2016). Simple and Integrated Spintip-Based Technology Applied for Deep Proteome Profiling. Anal. Chem.. 88, 4864-4871;
- Leonie F. Waanders, Karolina Chwalek, Mara Monetti, Chanchal Kumar, Eckhard Lammert, Matthias Mann. (2009). Quantitative proteomic analysis of single pancreatic islets. PNAS. 106, 18902-18907;
- Siyang Li, Brian D. Plouffe, Arseniy M. Belov, Somak Ray, Xianzhe Wang, et. al.. (2015). An Integrated Platform for Isolation, Processing, and Mass Spectrometry-based Proteomic Profiling of Rare Cells in Whole Blood*. Molecular & Cellular Proteomics. 14, 1672-1683;
- Nan Wang, Mingguo Xu, Peng Wang, Liang Li. (2010). Development of Mass Spectrometry-Based Shotgun Method for Proteome Analysis of 500 to 5000 Cancer Cells. Anal. Chem.. 82, 2262-2271;
- Camille Lombard-Banek, Sally A. Moody, Peter Nemes. (2016). Single-Cell Mass Spectrometry for Discovery Proteomics: Quantifying Translational Cell Heterogeneity in the 16-Cell Frog (Xenopus) Embryo. Angew. Chem. Int. Ed.. 55, 2454-2458;
- Liangliang Sun, Kyle M. Dubiak, Elizabeth H. Peuchen, Zhenbin Zhang, Guijie Zhu, et. al.. (2016). Single Cell Proteomics Using Frog (Xenopus laevis) Blastomeres Isolated from Early Stage Embryos, Which Form a Geometric Progression in Protein Content. Anal. Chem.. 88, 6653-6657;
- Ying Zhu, Paul D. Piehowski, Rui Zhao, Jing Chen, Yufeng Shen, et. al.. (2018). Nanodroplet processing platform for deep and quantitative proteome profiling of 10–100 mammalian cells. Nat Commun. 9;
- Yongzheng Cong, Khatereh Motamedchaboki, Santosh A. Misal, Yiran Liang, Amanda J. Guise, et. al.. (2021). Ultrasensitive single-cell proteomics workflow identifies >1000 protein groups per mammalian cell. Chem. Sci.. 12, 1001-1006;
- Miriam Goebel-Stengel, Andreas Stengel, Yvette Taché, Joseph R. Reeve. (2011). The importance of using the optimal plasticware and glassware in studies involving peptides. Analytical Biochemistry. 414, 38-46;
- Elien Vandermarliere, Michael Mueller, Lennart Martens. (2013). Getting intimate with trypsin, the leading protease in proteomics. Mass Spec Rev. 32, 453-465;
- Sarah M. Williams, Andrey V. Liyu, Chia-Feng Tsai, Ronald J. Moore, Daniel J. Orton, et. al.. (2020). Automated Coupling of Nanodroplet Sample Preparation with Liquid Chromatography–Mass Spectrometry for High-Throughput Single-Cell Proteomics. Anal. Chem.. 92, 10588-10596.