
Биоинформатика: большие БД против «большого Р»
10 июня 2014
Биоинформатика: большие БД против «большого Р»
- 2148
- 0
- 2
-
Автор
-
Редакторы
Мощный поток данных, который генерируют современные онкологические исследования, позволяет по-новому взглянуть на страшную, но в тоже время загадочную болезнь — Рак.
В 2013 году генетик Стивен Элледж (Stephen Elledge) ответил на вопрос, который тревожит онкологов уже около сотни лет. В 1914 году немецкий биолог Теодор Бовери (Theodor Boveri) предположил, что аномальное количество хромосом (анеуплоидия), которое часто наблюдают в опухолях, может стимулировать их рост. На протяжении большей части следующего столетия ученые добились крайне скромного прогресса по этому вопросу. Они обнаружили, что в опухолях часто присутствуют или отсутствуют дополнительные хромосомы или кусочки хромосом [1]. К сожалению, до недавнего времени никто не мог выяснить — именно это ли важно для роста опухоли, или такое явление — лишь побочный эффект.
Элледж, работающий генетиком в больнице Brigham and Women’s Hospital в Бостоне (Массачусетс, США), имеет свое мнение по этому поводу: «Люди игнорировали открытие Бовери в течение столь длительного времени из-за отсутствия веских доказательств. Мы просто не знали, правда ли то, что анеуплоидия в самом деле может быть причиной развития рака».
Элледж обнаружил, что опухоли растут более агрессивно в тех случаях, где анеуплоидия привела к потере генов-онкосупрессоров или к вставке дополнительных копий онкогенов [2]. Идея, что анеуплоидия — это не просто странная особенность опухолей, а двигатель их роста, основана на компьютерном анализе большого объема данных, собранных об опухолях. По словам Элледжа, это показывает, как способность компьютеров фильтровать все быстрее появляющуюся информацию может помочь нам углубить понимание онкологического процесса и распахнуть двери для новых открытий.
Создание подробных баз данных (БД) неизбежно придаст импульс современным методам диагностики и лечения онкозаболеваний. Когда пациенту ставят такой диагноз, не лишним будет секвенировать геном опухоли — это поможет установить генетические «поломки» и подобрать эффективные препарат и методику лечения (подробнее о методах см. [3]). В дальнейшем, секвенирование можно повторять для выявления изменений, появляющихся в геноме. Конечно, для контроля нужно получить геном и «здоровых» клеток: биоинформатический анализ различий может выявить причину возникновения онкологии, а сравнение с каталогами известных злокачественных мутаций позволят подтвердить найденную связь [4].
Такая практика, скорее всего, будет набирать популярность, тогда как расходы на нее будут снижаться (попутно с удешевлением секвенирования генома [5]). Врачи будут записывать результаты анализов и анамнез пациента (в том числе систему его питания и вредные привычки) в виде электронных историй болезни. Также желательно делать компьютерную (КТ) и магнитно-резонансную томографии (МРТ), которые помогут определить стадию заболевания. Если умножить всё вышесказанное на ≈1,7 млн. человек с диагнозом «рак» (статистика за 2013 год, и это только в одних Соединенных Штатах!), тогда станет окончательно ясно, что насколько огромными объемами данных оперирует сегодняшняя онкология.
Открытие Элледжа возн//икло вследствие разработки компьютерного алгоритма, который он и его коллеги называют «Исследователь Опухолей» (Tumor Suppressor and Oncogene Explorer). Этот метод использовался для сканирования двух генетических баз данных по раку, содержащих данные о 1,2 млн. мутаций из 8207 образцов тканей, относящихся к 20 видам опухолей:
- атлас Ракового Генома (Cancer Genome Atlas), созданный Национальным институтом рака США;
- каталог соматических мутаций при раке (Catalogue of Somatic Mutations in Cancer (COSMIC)), которым управляет институт Сенгера (Великобритания).
Для выявления «подозрительных» генов при сканировании этих баз данных исследователи особенное внимание уделяли таким свойствам как частота мутаций и соотношение «молчащих» мутаций и тех, что нарушают функцию гена. Затем они применяли статистические методы классификации для выявления генов-супрессоров и онкогенов. Около 70 генов-супрессоров и 50 онкогенов уже были известны для определенных типов опухолей, но Элледж и его коллеги увеличили этот список до 320 и 200, соответственно (эта информация приблизительная — предсказания могут быть ложноположительными, и требуют экспериментальной проверки). Они также идентифицировали биохимические пути, задействованные в тех или иных типах опухолей, что может быть крайне полезным при подборе прицельного лечения.
Такого рода исследования требуют обширных наборов данных. «Любая отдельная раковая клетка может показаться кашей, но скрупулёзный анализ на уровне целых опухолей может воссоздать целостную картину, — говорит Элледж. — Только рассмотрение онкологических процессов в совокупности позволит разглядеть в них закономерность».
Тяжело в учении...
Анализ геномов 8200 опухолей — это только начало. Исследователи «пытаются найти способ проанализировать миллион раковых геномов в ближайшие несколько лет», — говорит Роберт Гроссман (Robert Grossman), руководитель программы Data Intensive Science в Чикагском университете (Иллинойс, США). Это грандиозная затея: скомбинированный геном опухоли и здоровых тканей от одного пациента составляет около терабайта (1012 байт) данных, тогда как для работы с миллионом таких геномов потребуется около экзабайта памяти (1018 байт). По словам Гроссмана, хранение и исследование такого количества данных может стоить 100 миллионов $ в год. Для того чтобы ученым стало проще получать информацию, Гроссман и его коллеги разработали «облачную» (распределенную) платформу BioNimbus, обеспечивающую доступ к данным Атласа Ракового Генома и их анализ.
Такая объединенная база данных по раку может стать настоящим Клондайком для онкологии. Например, Меган МакНерни (Megan McNerney) — патологоанатом из Чикагского университета, — используя BioNimbus, разыскала ген, который инициирует острый миелоидный лейкоз (ОМЛ). Ученые уже знали, что при ОМЛ у некоторых пациентов наблюдается повреждение седьмой хромосомы (миссенс-мутации в некоторых генах). МакНерни выбрала 23 пациента из БД и с помощью компьютерной программы сравнила последовательности их РНК для поиска недостающих частей хромосомы. Она обнаружила, что у этих больных отсутствовала одна копия гена CUX1, кодирующего супрессор опухолей [6]. В опытах по удалению одной копии этого гена у плодовых мушек и мышей обнаруживается чрезмерно быстрое деление некоторых типов клеток крови и, как следствие, лейкемия. Открытие МакНерни не переросло в создание лекарств от ОМЛ (при этом диагнозе средняя продолжительность жизни составляет менее года!), но способствовало более глубокому изучению болезни и в дальнейшем позволит создать метод ранней диагностики и лечения этого заболевания. МакНерни говорит, что даже ее небольшой проект показал преимущества поиска информации в БД: «гигантский скачок в развитии баз данных позволил нам поставить исследования на новый уровень. Это кардинально изменит наши познания в биологии раковых заболеваний».
Геномика и другие -омики (о том, что это такое, см. в статье «„Омики“ — эпоха большой биологии» [7]) — не единственные источники биологической информации для такого рода исследований. Американское общество клинической онкологии (The American Society of Clinical Oncology, ASCO) разрабатывает платформу под названием CancerLinQ, призванную интегрировать электронные истории болезни онкобольных в единую медицинскую карту. Анализируемые источники всё чаще включают информацию о геноме, диагнозе, также примечания о лечении и показатели эффективности выбранной для пациента терапии. На данный момент в системе собраны сведения 177000 людей с раком молочной железы. Разработчики надеются, что система полностью вступит в строй к лету 2015 года, что позволит исследовать другие онкозаболевания.
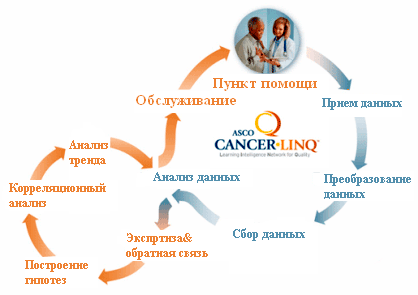
Рисунок 1. Второе рождение раковой терапии с помощью масштабных БД: прототип экспертной системы CancerLinQ
Клиффорд Худис (Clifford Hudis), специалист по раку молочной железы в Мемориальном раковом центре Слоун-Кеттеринг (MSKCC) в Нью-Йорке и президент ASCO, говорит, что CancerLinQ будет совершать открытия, остающиеся без внимания при клинических испытаниях. Например, накопление статистики по использованию лекарств позволит собрать данные о побочных эффектах, взаимодействии с другими лекарствами и эффективности лечения у разных популяций пациентов. Система также может регистрировать отклонение назначаемых дозировок от рекомендованных FDA. «Если обнаружится сотня случаев, когда врачи независимо друг от друга не соблюдают рекомендации, компьютер подметит это, из чего будет сделан вывод о неэффективности данных рекомендаций», — говорит Худис. Компьютер может обнаружить, например, что врачи достигают более заметного успеха в лечении, когда они варьируют дозировку в зависимости от возраста пациента.
Сочетание геномных данных и медицинской визуализации также внесет новизну в сферу диагностики. «Высокопроизводительные вычислительные базы данных позволяют нам автоматические сопоставлять информацию, полученную из разных методик», — говорит Дэвид Форан (David Foran), патологоанатом и глава отдела информатики в раковом центре Ратгерса в Нью-Джерси. В центре делают цифровые фотографии тканей с высоким разрешением, которые сравниваются с образцами других пациентов для правильной диагностики опухоли.
Исследователи будут анализировать геном пациента, чтобы оценить эффективность того или иного вида терапии в его случае, и подтверждать это на томографии. Или же они могут найти корреляцию между курением, мутациями и выбором терапии. «Компьютерная программа может одновременно просмотреть все образцы, которые находятся в базе, и сделать обобщение», — говорит Форан.
Сопоставление и анализ такого количества информации позволит аккумулировать весь медицинский опыт в одном месте, добавляет Форан. «Когда вы обращаетесь к врачу, особенно онкологу, вы полагаетесь на его опыт и компетенцию. Сейчас мы разрабатываем систему хранения и анализа информации о сотне тысяч онкобольных.» Это похоже на то, как если бы врач принимал решение по диагностике и терапии, опираясь на личный опыт при лечении всех этих пациентов.
Геномная информация и электронные медицинские карты являются новыми, но не единственными источниками данных. Например, архивы больницы Джонса Хопкинса в Балтиморе (штат Мэриленд) хранят более полумиллиона бумажных историй болезни, начиная с момента основания клиники в 1889 году до введения в эксплуатацию электронных историй болезни в 1984-м. И в других странах, и в каждом штате США сохраняется раковая статистика за десятилетия. Например, Дания ведет такую документацию с 1943 года. В Великобритании в прошлом году запущена база данных, содержащая информацию обо всех случаях заболевания раком на территории страны по сегодняшний день (11 миллионов анамнезов за 30-летний период). Добавляя все эти исторические сведения в компьютерную систему, можно увеличить количество важных находок и преодолеть многие трудности в области онкологии.
...легко в бою
Проблема в том, что все эти новые технологии создают настоящий информационный бум. «Данные накапливаются намного быстрее, чем мы успеваем научиться использовать их по назначению», — говорит Маниш Парашар (Manish Parashar) — биоинформатик, который сотрудничает с Фораном в области алгоритмов обработки информации. По его словам, «наше желание охватить такое количество данных порождает некоторые проблемы».
Одна из основных проблем с (много)терабайтными данными — это как работать с такими объемами информации. Медицинские изображения высокого разрешения могут занимать с десяток гигабайт, а исследователь, возможно, захочет, чтобы компьютер сравнил десятки тысяч таких изображений. Обработка одного такого файла может занять более 10 минут, а копирование их по сети и вовсе может затянуться на неопределенное время. «Зачастую проще переписать эти файлы на портативный винчестер и носить с собой, чем ждать, пока они скопируются по локальной сети или тем более интернету», — говорит Парашар.
Для преодоления этих проблем информатики разрабатывают алгоритмы разбивки данных на меньшие части для параллельной обработки на отдельных процессорах и методики сжатия файлов (контролируя, конечно, чтобы оно не уничтожило важные медицинские подробности).
Форан подчеркивает, что в лечении рака произошел заметный сдвиг, — онкология взяла курс на персонализацию. Однако рак — всегда сложное многофакторное заболевание, развитие которого зависит как от генетических причин, так и от некоторых факторов окружающей среды. Именно поэтому большие БД помогут родиться новым, более целенаправленным методикам борьбы с «большим Р».
Перевод с английского по материалам Nature [9].
Литература
- Как распознать рак при помощи биомаркеров?;
- Teresa Davoli, Andrew Wei Xu, Kristen E. Mengwasser, Laura M. Sack, John C. Yoon, et. al.. (2013). Cumulative Haploinsufficiency and Triplosensitivity Drive Aneuploidy Patterns and Shape the Cancer Genome. Cell. 155, 948-962;
- Types of Cancer Treatment. National Cancer Institute;
Скверный анекдот: негр, китаец и Крейг Вентер...;- Технология: $1000 за геном;
- Megan E. McNerney, Christopher D. Brown, Xiaoyue Wang, Elizabeth T. Bartom, Subhradip Karmakar, et. al.. (2013). CUX1 is a haploinsufficient tumor suppressor gene on chromosome 7 frequently inactivated in acute myeloid leukemia. Blood. 121, 975-983;
- «Омики» — эпоха большой биологии;
- George W. Sledge, Clifford A. Hudis, Sandra M. Swain, Peter M. Yu, Joshua T. Mann, et. al.. (2013). ASCO's Approach to a Learning Health Care System in Oncology. JOP. 9, 145-148;
- Neil Savage. (2014). Bioinformatics: Big data versus the big C. Nature. 509, S66-S67.






