Технология: $1000 за геном
01 июня 2014
Технология: $1000 за геном
- 3118
- 0
- 3
Обложка книги The $1,000 Genome: The Revolution in DNA Sequencing and the New Era of Personalized Medicine
-
Автор
-
Редакторы
С помощью уникальной программы финансирования перспективных разработок в области изучения генома человека правительству США практически удалось достичь долгожданной цели: снизить стоимость секвенирования генома до $1000.
Складывается такое впечатление, что в Силиконовой долине закон Мура стоит на равных с законами природы, — например, открытыми Исааком Ньютоном. В эмпирическом наблюдении Гордона Мура — соучредителя компании Intel — говорится, что основные вычислительные характеристики компьютеров улучшаются в два раза каждые два года, и при этом технологии становятся дешевле. Постепенно эта закономерность начала проникать в смежные сферы науки и техники, добравшись и до исследований генома.
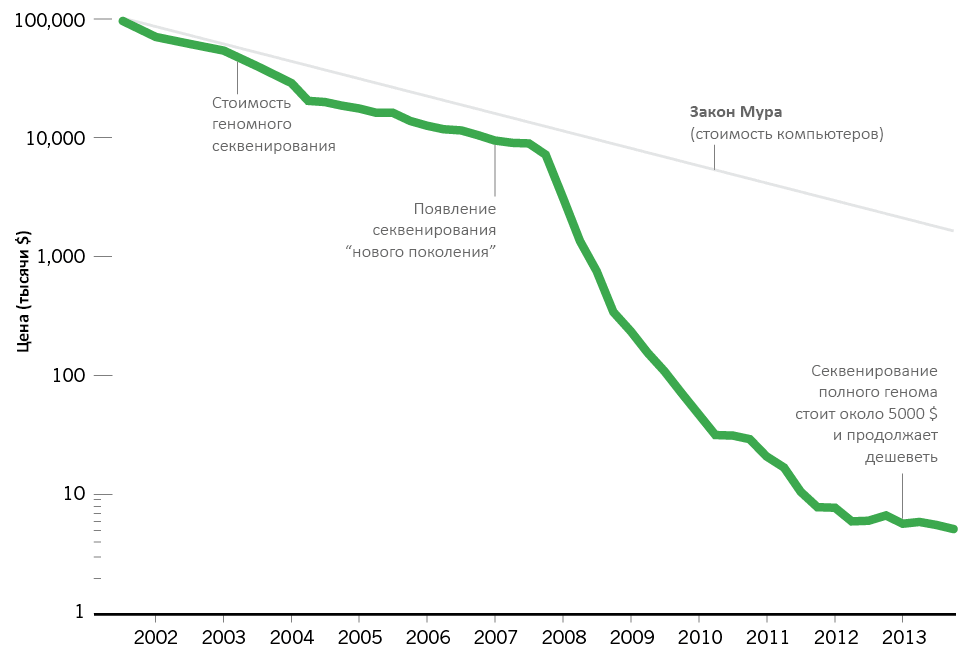
В течение последних нескольких лет ученые в сотнях докладов сравнивали наклон прямой из закона Мура с быстрым снижением затрат на секвенирование ДНК (была эта картинка и на «Биомолекуле» [1]). Некоторое время статистические данные совпадали, но в 2007 году наметился качественный скачок. Цена за секвенирование человеческого генома резко упала с 10 млн. долларов США до всего нескольких тысяч буквально за шесть лет. Это не просто опередило закон Мура, но и поставило под сомнение авторитет этого «бессмертного» постулата. И так же, как легкий доступ к персональным компьютерам изменил мир, быстрые темпы развития геномных технологий совершили революцию среди биологических исследований. Что, в свою очередь, может привести к кардинальным прорывам в медицине.
Большинство участников «глубокосеквенируемого движения» считает, что изрядная доля успеха в этой отрасли заложена схемой финансирования, организованной американским Национальным исследовательским институтом генома человека (NHGRI). Официально называемая Премией за прогрессивные технологии секвенирования, это программа шире известна как $1000 и $100 000 за геном. Начиная с 2004 года, по этой программе были выделены гранты для 97 академических и коммерческих исследовательских групп, включая лаборатории в крупнейших компаниях по секвенированию.
Программа катализировала мобильность и сотрудничество между разработчиками и помогла запустить работу в десятках конкурирующих компаний, предупредив застой, который мог бы сковать все после завершения проекта «Геном человека» в 2003 году [2]. «Лидирующие компании на рынке действительно преобразили подход к секвенированию, и всё это началось с финансирования NHGRI», — говорит Джина Коста (Gina Costa), которая работала в пяти влиятельных компаниях и в настоящее время является вице-президентом фирмы Cypher Genomics (Сан-Диего, США), занимающейся интерпретацией геномных данных.

Рисунок 2. На иллюстрации Кристофера Шорта (Christopher Short) показаны стадии расшифровки ДНК: от клеточного уровня до молекулярного
National Human Genome Research Institute, Associated Press
Стоя на плечах гигантов
На сегодняшний день программа «$1000 за геном» близка к достижению своей цели, и уже в этом году будет проводиться награждение и предоставление окончательных грантов. Возникает вопрос о том, как 230-миллионная государственная программа достигла таких результатов, и могла бы такая формула успеха применяться в других отраслях? Но Джеффри Шлосс (Jeffery Schloss), директор отдела геномных наук в NHGRI в Бетесде, штат Мэриленд, который самого начала руководит программой, говорит, что достижение аналогичного успеха возможно лишь при правильном и регулируемом партнерстве государственных и частных компаний: «...Одной из наших задач является выбрать правильную тактику для сотрудничества с правительством, чтобы не мешать, а поддерживать развитие технологий в частном секторе».
Секвенирование первого генома человека было масштабным событием и настоящим технологическим вызовом. В период между 1990 годом и публикацией черной версии генома 2001 году более 200 ученых объединили свои усилия, чтобы прочитать примерно 3 млрд. оснований ДНК, которые содержат наш генетический материал [3]. Фрэнсис Коллинз (Francis S. Collins) — руководитель проекта «Геном человека» — сказал: «мы выпускаем в свет первое издание „Книги жизни“», но на самом деле наш геном довольно трудно интерпретировать как конкретные инструкции, которые регулируют все метаболические и биологические функции. Понимание того, как ДНК на самом деле влияет на здоровье человека, требует изучения и обобщения примеров связи между генами и биологическими процессами у тысяч, а, возможно, и миллионов людей.
Некоторое время доминирующей технологией секвенирования ДНК был метод Сэнгера — довольно медленный и трудоемкий процесс. Суть его заключается в создании копий ДНК, при синтезе которых происходит включение нуклеотидов с химически модифицированными флуоресцентными метками. В период господства этой технологии на рынке секвенаторов царила компания Applied Biosystems (Фостер-Сити, Калифорния), которая поставляла оборудование для узкого круга заказчиков (в основном, государственных лабораторий), и ничто не предвещало кардинальных перемен.
Учитывая актуальность быстрого и безошибочного секвенирования ДНК, ученые внедряли параллельные технологии и роботизировали процесс. На совещании, созванном NHGRI в 2002 году, было высказано предположение, что такое развитие событий могло бы снизить расходы в 100 раз в течение следующих пяти лет. Но и этого было недостаточно.
На собрании была высказана мысль, что уменьшение цены позволит сделать секвенирование генома рутиной, и врачи смогут проводить данную процедуру наравне со сканирующей магнитно-резонансной томографией. Это казалось слишком амбициозным, учитывая тогдашнее состояние технологии. «Существует сомнение в готовности инвесторов тратить деньги на такую технологию», — говорит Эрик Айзенштадт (Eric Eisenstadt), отставной чиновник, который в прошлом руководил оборонными научно-исследовательскими проектами правительства США.
Однако Дж. Шлосс и NHGRI начали финансировать разработку совершенно новых методов секвенирования, а также коммерческое внедрение таких технологий. Сочетание фундаментальных и прикладных разработок в рамках одной программы было редкостью для Национальных институтов здравоохранения (NIH) — вышестоящей организации над NHGRI. Проект был более перспективным, чем типичное исследование, поддержанное грантом NIH, поскольку это позволило агентству выделять небольшие награды за многообещающую, но рискованную работу. Шлосс отмечает, что «такая гибкость является необычной для NIH».
Кроме того, программа оказала поддержку компаниям, которые могли бы конкурировать с Applied Biosystems. Одной из таких компаний стала 454 Life Sciences. Деятельность фирмы 454 была направлена на разработку метода секвенирования, который был бы принципиально намного быстрее и дешевле, чем метод Сэнгера. Технология заключалась в более простой процедуре по подготовке проб и проведению большинства реакций на твердой поверхности [4]. Однако, когда компания пыталась привлечь финансирование, инвесторы уклонялись от сотрудничества, объясняя это преждевременностью появления такой технологии: «Люди говорят: почему вы хотите так быстро секвенировать ДНК? Ведь мы же уже закончили проект „Геном человека“».
Технология пиросеквенирования, разработанная 454 Life Sciences благодаря инвестиции от NHGRI размером в $7 млн., стала достаточно прибыльной. Таким образом, эта компания первой начала постепенную отмену монополии Applied Biosystems.
Полученное финансирование в итоге помогло убедить частных инвесторов тоже выйти на рынок. Стивен Тернер (Stephen Turner), основатель и главный технический директор Pacific Biosciences, говорит, что грант в $6,6 млн. от NHGRI, полученный его компанией в 2005 году, помог дальнейшему привлечению венчурного капитала. Окно, «прорубленное» NHGRI, убедило инвесторов вкладывать в компанию более существенные деньги для того, чтобы довести технологию, основанную на наблюдении за синтезом ДНК в реальном времени, до ума. По словам Тернера, «можно иметь высококвалифицированных специалистов в технологии секвенирования, но большее влияние будет оказывать размер счета компании, которая проводит исследования».
Инвестиции NHGRI, которые обычно составляют несколько миллионов долларов, не могли сами по себе взрастить технологию и вывести ее из лаборатории на рынок. Но они могли финансировать ее отдельные части, — например, работы по совершенствованию красителей, разработке схем, лазеров и тестирование комбинаций отдельных компонентов.
В рамках программы было проинвестировано $88 млн. в технологии, основанные на нанопорах — здесь последовательность нуклеотидов ДНК «считывается» по мере ее протягивания, подобно нити, через пору [5]. Эта технология освободила бы от необходимости проводить дорогие и медленные реакции, что на данный момент используются для амплификации молекулы ДНК. Но решение фундаментальных вопросов — в том числе, как заставить ДНК двигаться через пору достаточно медленно и с постоянной скоростью, — стало достаточно проблематичным. NHGRI профинансировал работу, чтобы преодолеть эти препятствия, — в том числе $9,3 млн. было выделено компании Oxford Nanopore Technologies. Тернер говорит, что такие инвестиции помогают сократить расходы на секвенирование, прежде чем технология попадает в массовое использование.
Специалисты в области секвенирования заявляют, что проект «$1000 за геном» взрастил множество компаний и лабораторий, фактически создав новую экспертную область. Из наиболее ярких таких примеров — компания Illumina в Сан-Диего, что в настоящее время стала лидером по продаже секвенаторов. Illumina, технология которой заключается в считывании множества коротких отрезков ДНК, приобрела несколько других компаний и переманила многих ученых, которые в прошлом финансировались NHGRI. «За счет новых приобретений Illumina стремительно развивается», — говорит Мустафа Ронакхи (Mostafa Ronaghi), технический директор компании.

Рисунок 3. Достижения в области оптики, дизайна проточной ячейки и химической кластериза-ции позволяют исследователям одновременно обрабатывать множество образцов цельного генома человека. Слева: Прибор HiSeq X может секвенировать до 16 геномов менее чем за 3 дня. Каждый прибор генерирует до 1,7 Тб «сырых» данных. Справа: Комплекты реагентов для HiSeq X HD, использующиеся в форм-факторе проточных ячеек.
Конкурирующие компании по разработке технологий секвенирования принимают участие в ежегодном собрании, посвященном отраслевому прогрессу. По словам Тернера, «эта встреча является одним из важнейших мест для обсуждения того, что происходит в сфере геномного секвенирования, и позволяет обмениваться необходимыми знаниями».
Утряска и усушка
Некоторые ученые ставят под вопрос достижения программы NHGRI. Например, Коста выражает недовольство, что «на разработку нанопор было выделено много денег, а толку что-то маловато». Кевин Маккернан (Kevin McKernan) сотрудничал с Коста для развития технологии секвенирования SOLiD (основанной на ферментах, «сшивающих» фрагменты ДНК вместе). Он указывает, что многие из компаний, которые финансировались в рамках программы «$1000 за геном», в конечном счете, не оправдали надежд, которые были на них возложены: «доля успеха у этих компаний, вероятно, ненамного выше, чем в венчурном инвестировании».
Однако прочие отдают Шлоссу и его программе должное за создание благоприятного инвестиционного климата в сфере научных и коммерческих приложений. Многие фирмы, которые финансировались NHGRI, уже не существуют — например, 454 и Helicos BioSciences, — но другие получатели грантов продолжают работать, постоянно совершенствуя технологию.
«NHGRI финансирует небольшие компании и академические группы для создания оптимального источника информации и разработки технологий», — говорит Ронакхи. Однако они не определились с технологиями, на которые нужно сделать ставку.
Секвенирование все еще нуждается в большом количестве доработок, особенно с точки зрения качества прочтения последовательности ДНК. Несмотря на высокую стоимость и сложность, секвенирование по Сэнгеру продолжает оставаться эталоном точности. А затраты на секвенирование уже не снижаются так быстро, как это было несколько лет назад. Но исследователи настроены оптимистично, и считают, что на смену Illumina должна прийти другая технология.
Теперь, когда секвенирование становится по-настоящему дешевым, можно начинать говорить о сканировании полных геномов отдельных пациентов, ну или по крайней мере тех его областей, которые кодируют белки. Но, впрочем, пока все равно совершенно не ясно, как эта информация поможет увеличить качество медицинских услуг и приблизит эру пресловутой персонализованной медицины [6]. Поиск и нахождение ответов на такие сложные вопросы помогут сделать еще один большой скачок в геномике, по сравнению с которым закон Мура покажется просто мелочью.
Перевод с английского по материалам Nature [7].
Литература
- Огурцы-убийцы, или Как встретились Джим Уотсон и Гордон Мур;
- Геном человека: как это было и как это будет;
- Eric S. Lander, Lauren M. Linton, Bruce Birren, Chad Nusbaum, Michael C. Zody, et. al.. (2001). Initial sequencing and analysis of the human genome. Nature. 409, 860-921;
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Katharine Sanderson. (2008). Personal genomes: Standard and pores. Nature. 456, 23-25;
- Упреки в нарциссомике;
- Erika Check Hayden. (2014). Technology: The $1,000 genome. Nature. 507, 294-295.