
Вычислительное будущее биологии
29 ноября 2013
Вычислительное будущее биологии
- 3402
- 3
- 9
Смогут ли компьютерные исследования заменить экспериментатора?
-
Автор
-
Редакторы
Темы
Появление большого количества баз данных, хранящих в открытом доступе последовательности ДНК, структуры белков и фенотипические описания тысяч организмов, привело к перевороту в биологии. Теперь ученые могут совершать открытия, ни разу не прикоснувшись к пипетке и не проведя ни одного эксперимента. В статье рассказывается об успехах и перспективах применения компьютерных технологий в биологических исследованиях.
Большинство ученых, работающих в области естественных наук, строят свои научные карьеры вокруг одного конкретного организма или заболевания, иногда — одного молекулярного пути или семейства белков. Это неудивительно, ведь часто для того, чтобы научиться культивировать в чашке Петри определенный тип клеток или освоить новый лабораторный метод, нужны месяцы упорной работы. Но есть и такие ученые, которые легко переходят от одной темы к другой, что не мешает им регулярно получать важные научные результаты и совершать открытия. Пример этому — Атул Бьют (Atul Butte), который в свои 44 года благодаря открытиям в изучении диабета, ожирения, трансплантологии и обнаружению новых лекарств для лечения рака легких и других заболеваний уже является одним из ведущих исследователей медицинского факультета Стэнфордского университета (рис. 1).

Рисунок 1. Атул Бьют выступает на конференции «Базы данных в биомедицине» (Big Data in BioMedicine)
Видео 1. Выступление Атула Бьюта на конференции TedMed 2012: как извлечь знания из всех тех данных, что мы уже имеем (на английском языке)
Как карьера Бьюта отличается от карьеры классического биолога, так же и его лаборатория отличается от лабораторий большинства его университетских коллег. Она не заполнена реагентами и инкубаторами для клеточных культур, а больше напоминает рабочее место инженера или разработчика программного обеспечения: большую часть времени Бьют работает на своем ноутбуке, иногда обращаясь к большому компьютерному кластеру Стэнфордского университета или другому суперкомпьютеру. Вместо выращивания клеток и секвенирования ДНК, его коллеги по лаборатории проводят время перед мониторами и тщательно анализируют находящиеся в свободном доступе биологические базы данных, такие как последовательность генома человека, генетические данные больных раком, записи сканирования головного мозга, наборы биомаркеров различных заболеваний, таких как болезнь Альцгеймера и диабет.
В английском языке такие лаборатории жаргонно называют «сухими» (dry lab), в противовес классическим «мокрым» лабораториям (wet lab), где основу исследований составляют сделанные руками эксперименты. Два года назад Бьют и его коллеги, используя публично доступные данные, исследовали активность большого набора человеческих генов и пришли к неожиданным заключениям. Были исследованы две группы данных: активность этих генов у людей, больных сотней различных заболеваний, и их активность в клеточных культурах, обработанных разными лекарственными препаратами. После этого ученые сопоставили, какие гены становятся более, а какие — менее активными при различных болезнях и при применении лекарственных средств. Оказалось, что лекарство, используемое сейчас для лечения язвы, также может оказаться полезным в лечении рака легких, а антиэпилептический препарат, вероятно, поможет справиться с воспалением кишечника (рис. 2). Последующие лабораторные исследования на модельных животных подтвердили оба предположения, так что впереди — проверка в клинических испытаниях. Такой же биоинформатический подход показал, что антидепрессант имипрамин может быть эффективным лекарством при мелкоклеточном раке легких, устойчивом к стандартной химиотерапии. Это открытие уже привело к запуску клинических испытаний нового эффекта имипрамина. «Сейчас удивительное и волнующее время для того, чтобы заниматься биологическими исследованиями в „сухой“ лаборатории», — говорит Бьют.
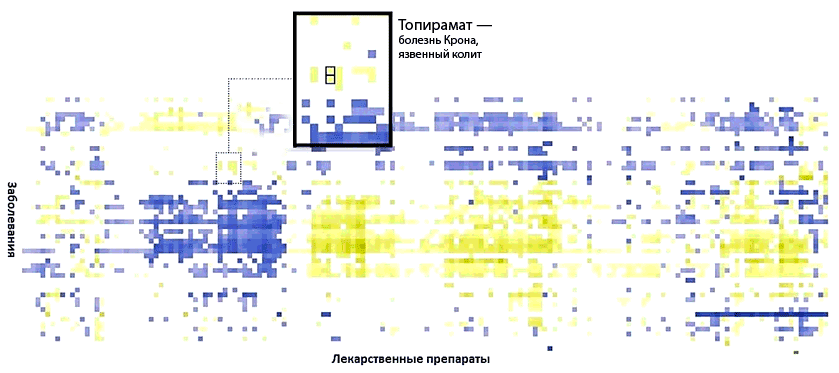
Рисунок 2. Визуальное представление результатов анализа, проведенных командой Бьюта. Синим обозначены лекарства, которые могут вызвать ухудшение болезни, желтым — те, которые могут помочь в лечении. Например, антиэпилептический препарат топирамат может быть использован и для лечения болезни Крона и язвенного колита.
Рост количества публично доступных баз биологических данных с последовательностями генов, описанием их активности, структурой белков и их взаимодействиями открывает новые перспективы для ученых. Последние разработки в области компьютерной техники — рост вычислительных мощностей, возможность хранения больших объемов информации и новые алгоритмы, которые помогают эффективно отделить зерна от плевел, — дают исследователям-биоинформатикам возможность совершать фундаментальные открытия без необходимости брать в руки пипетку. Например, благодаря проекту iPlant Colaborative появляется новое поколение ботаников, которые занимаются лишь анализом данных и при этом ни разу не испачкали руки, копая землю или поливая молодые ростки.
«Сейчас не обязательно работать в классической „мокрой“ лаборатории для того, чтобы заниматься биологическими исследованиями», — подтверждает Дэвид Хекерман (David Heckerman), информатик в Microsoft Research в Лос-Анжелесе (рис. 3). Но не стоит думать, что биоинформатики предлагают заменить традиционные методы компьютерными вычислениями. Наоборот, они призывают к большей интеграции и тесному взаимодействую двух методологических подходов современной биологии, что по их мнению должно привести к удивительным результатам.

Рисунок 3. Так выглядит рабочее место Дэвида Хекермана — ни колб с реактивами, ни пипеток
Данные, доступные всем
Большие объемы данных — не новость для науки. Например, большой адронный коллайдер в ЦЕРНе получает 15 петабайт (1015) данных ежегодно, а Слоановский цифровой небесный обзор в год сохраняет несколько терабайт (1012) информации. Нет ничего необычного в больших базах данных и в биологии. Например, на конец августа 2013 года в базе генетической информации GenBank, которой в этом году исполнился 31 год, содержалось примерно 167 миллионов последовательностей генов, или 154 миллиарда нуклеотидов.
Не в новинку для биологов и использование компьютерных технологий. Уже многие годы ученые активно используют алгоритмические подходы для оперирования с базами биологических данных, создав с их помощью геномику, протеомику, метаболомику и другие «-омики» (см. «„Омики“ — эпоха большой биологии» [1]). Однако большинство из этих исследований до недавнего времени велись большими исследовательскими группами, которые имели исключительное право на обработку полученных данных до того, как сделать их публично доступными. Теперь значительная часть этой информации свободно может быть использована другими исследователями.
Для решения объемных задач необходимы хорошее компьютерное обеспечение и специализированные алгоритмы, и они становятся лучше с каждым годом. Хекерман и его коллеги из Microsoft Research недавно произвели фурор в биоинформатике, разработав улучшенный алгоритм для работы с большими объемами генетических данных и полногеномного поиска ассоциаций. В таких исследованиях изучают последовательности ДНК большой группы больных людей и сравнивают их с аналогичными результатами для группы людей здоровых, пытаясь обнаружить последовательности ДНК, характерные для того или иного заболевания. Чаще всего эти характерные последовательности очень сложно заметить, т.к. наследование заболеваний не похоже на наследование простых генетических признаков, таких как цвет зерен у гороха в экспериментах Менделя. Зеленый или желтый цвет зерен гороха кодируется одним геном, но предрасположенность к тому или иному заболеванию чаще всего связана с особенностями многих генов.
«Чтобы заметить эти маленькие особенности, необходимо обработать тонны данных. Нужно изучить десятки тысяч или даже сотни тысяч людей, — говорит Хекерман. — Но в большом объеме данных кроется свой подвох. При анализе большого количества информации вы можете упустить, что в ней есть внутренняя структура». Эта структура связана с тем, что некоторые из изучаемых индивидуумов могут иметь множество сходных генетических особенностей, которые не имеют отношения к изучаемому заболеванию. Это, как правило, объясняется тем, что эти индивидуумы более родственны друг другу с точки зрения популяционной генетики, чем другие. В результате исследователь получает данные, которые похожи на значимый результат, но при ближайшем рассмотрении им не являются.
Один из способов решить эту проблему — использовать подход под названием линейная смешанная модель (linear mixed model). Математическая точность этого метода помогает уменьшить количество ложноположительных результатов, но компьютерные мощности, необходимые для применения этого метода — это количество анализируемых объектов, возведенное в третью степень. Это не проблема, если анализируются данные чуть более десятка пациентов, но если нужно изучить сотни геномных последовательностей, применение такого подхода становится невозможным.
После нескольких попыток решить проблему ложноположительных результатов Хекерман и его коллеги придумали то, что он называет «простой алгебраической хитростью». Новый алгоритм, названный FaST-LMM (Factored Spectrally Transformed Linear Mixed Models), не требует больших вычислительных мощностей, но не менее эффективен. Он помог избавиться от спорных результатов, позволил увеличить допустимый размер анализируемой выборки и смог, таким образом, повысить шансы обнаружить что-то действительно важное при полногеномном поиске ассоциаций или других видах анализа больших наборов данных. В 2012 году группа Хекермана использовала FaST-LMM и суперкомпьютер Microsoft Azure для сопоставления геномов нескольких тысяч человек из базы данных благотворительной британской биомедицинской организации Wellcome Trust. Они проанализировали в общей сложности 63 524 915 020 пар генетических маркеров и обнаружили множество новых ассоциаций, которые могут служить биомаркерами биполярного расстройства , ишемической болезни сердца, гипертонии, воспаления кишечника, ревматоидного артрита и диабета 1 и 2 типов [2].
Своевременная идентификация биполярного расстройства поможет, в том числе, предотвращать самоубийства: «Как предотвратить самоубийство» [3]. — Ред.
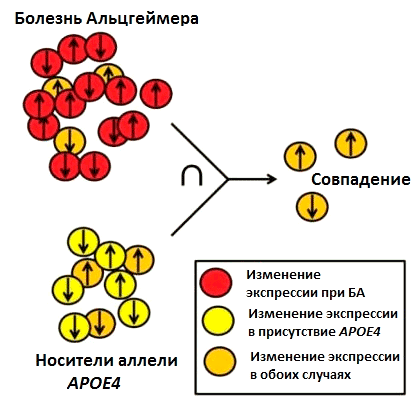
Рисунок 4. В исследованиях Абелиовича сравнивались два набора данных об экспрессии генов: экспрессия генов при болезни Альцгеймера (БА) и экспрессия генов у носителей аллели APOE4. В результате удалось выявить гены, экспрессия которых изменяется в обоих случаях. Вероятно, именно от работы этих генов и зависит вероятность развития БА у людей с аллелью APOE4.
Подходы «сухой» лаборатории нашли свое применение не только в полногеномном анализе ассоциаций, но и в других областях биомедицинских исследований. Например, научная группа из Колумбийского университета под руководством Азы Абелиовича (Asa Abeliovich) недавно опубликовала в журнале Nature статью с результатами анализа большого массива данных для выявления новых факторов риска развития болезни Альцгеймера [4]. Ранее было известно, что люди, являющиеся носителями аллеля APOE4, подвержены высокому риску болезни Альцгеймера, однако оставалось неясным, есть ли какие-либо дополнительные генетические факторы, повышающие или снижающие этот риск. Проанализировав публично доступные данные об экспрессии генов в головном мозге людей, у которых диагностировали болезнь Альцгеймера, и здоровых людей, ученые выяснили, что два гена — SVA2A и RNF219 — были значительно менее активны у больных по сравнению со здоровыми (рис. 4).
Эта новая информация, объединенная с более ранними данными о возможных функциях этих двух генов, подсказала, что SVA2A и RNF219, вероятно, играют важную роль в накоплении амилоидных агрегатов. Амилоидные агрегаты представляют собой скопления белка β-амилоид, появляющиеся в мозге больных людей и являющиеся возможной причиной развития болезни Альцгеймера . Группа Абелиовича подтвердила полученные результаты в лабораторных исследованиях на мышах, а чтобы получить более веские подтверждения своей гипотезы, привлекла к анализу публично доступные данные о сканировании головного мозга пациентов с болезнью Альцгеймера. Оказалось, что различные варианты гена RNF219 коррелируют с количеством амилоидных агрегатов в мозге пациентов.
Справедливости ради нужно уточнить, что точная роль патогенного β-амилоида до конца не понята (см. «Альцгеймеровский нейротоксин: ядовиты не только фибриллы» [5]), а его «нормальная» форма и вовсе может быть частью системы врожденного иммунитета (см. «Возможно, β-амилоид болезни Альцгеймера — часть врождённого иммунитета» [6]). — Ред.
Это исследование важно не только потому, что в перспективе приведет к разработке новых лекарственных средств, но и потому, что может помочь врачам применять более индивидуальный подход к пациентам. Генетический анализ поможет разделять пациентов на группы, как это сейчас делают при лечении рака, а эффект от индивидуальной терапии значительно выше. Это комплексное исследование является примером того, что было сложно себе представить еще пять лет назад: совместное использование анализа молекулярно-генетических данных, лабораторных экспериментов и привлечение результатов сканирования мозга привело к важному открытию.
Не только биология и медицина
То, что за последние годы были определены последовательности большого количества геномов растений, и многие из них появились в свободном доступе, не могло не подтолкнуть ботаников к проведению собственных биоинформатических исследований. Такими исследованиями в Корнельском университете занимается лаборатория Эда Баклера (Ed Buckler) — специалиста по генетике кукурузы. Баклера и его коллег интересует устойчивость к заболеваниям различных видов кукурузы. В одной из своих недавних публикаций они сравнивают геномы 103 видов кукурузы, изучив тысячу последовательностей генов и некодирующих участков (рис. 5). Было обнаружено, что некоторые признаки растения, такие как устойчивость к патогенам или время цветения, связаны с определенными некодирующими участками ДНК [7]. Теперь ученые помогают селекционерам в отбирать линии растений с необходимыми признаками по последовательности некодирующей ДНК.
Рисунок 5. Так могут выглядеть исследования многих современных ботаников: сравнение последовательностей ДНК, а не выращивание рассады
Биоинформатика помогает ответить и на более отвлеченные вопросы о жизни растений. Дэвид Санкофф (David Sankoff) — математик из университета Оттавы — изучил последовательности геномов 30 видов покрытосеменных растений для того, чтобы попытаться реконструировать геном их общего предка, жившего около 120 миллионов лет назад. При этом его интересует не точная последовательность ДНК этого древнего растения, а принципы устройства его генома. Недавно были сделаны первые успешные шаги в этом направлении. После анализа количества дупликаций и трипликаций в геномах современных эудикот — одной из групп покрытосеменных растений — Санкофф и его коллеги заключили, что у общего предка этих растений было семь хромосом, содержавших 20–30 тысяч генов (то есть, значительно меньше, чем есть в геноме современных растений). Несмотря на то, что подобные открытия вряд ли принесут пользу сельскому хозяйству или другим коммерческим отраслям, многих ученых интересуют фундаментальные вопросы биологии, а биоинформатика помогает находить на них ответы.
Проблема совместимости
Активно развивающаяся вычислительная биология сталкивается с большим количеством сложностей, и одна из главных — получение доступа к данным других исследователей. Чаще всего люди, потратившие годы или даже десятки лет на создание базы данных, не горят желанием поделиться результатом со всеми. Они надеются самостоятельно обработать полученную информацию до того, как другие совершат революционное открытие, опираясь на их данные. А может быть, данные еще слишком сырые и нуждаются в доработке. «Это действительно непростые и важные проблемы, — комментирует Бьют. — Нам нужна хорошая система для поощрения людей, готовых делиться результатами своей работы».
Еще одна проблема, мешающая биоинформатике — отсутствие стандартов записи биологической информации. Проблема не только в том, что разные исследовательские группы используют разные файловые системы для хранения данных, но и в том, что иногда дизайн экспериментов может значительно разниться, из-за чего обязательно возникнут споры о том, что же все-таки было изучено в ходе работы. Бьют и некоторые из его коллег соглашаются, что проблема разных форматов файлов неприятная, но решаемая, чего не скажешь о проблеме различий в дизайне экспериментов, которые трудно учесть при крупномасштабном анализе.
Но есть и успехи в решении этой проблемы. Годы работы, потраченные на то, чтобы стандартизовать экспериментальные методики, начинают приносить свои плоды. Благодаря развитию РНК- и ДНК-микрочипов и масс-спектрометрии в последнее время биологические данные становятся все более стандартизованными.
Другим поводом для беспокойства становится сохранение конфиденциальности генетической информации. Как утверждают генетики, даже анонимные генетические данные могут выдать своего владельца и, таким образом, обнаружить не только его медицинские проблемы, но и предрасположенность к заболеваниям у его родственников. Одно из возможных решений этой проблемы уже существует и используется на практике. Для того, чтобы получить доступ к базе данных генотипов и фенотипов американского Национального центра биотехнологической информации (NCBI), необходимо пройти регистрацию и получить одобрение администрации этого центра. Более того, все запросы в базу доступны публично, а значит, любой человек может узнать, кто и зачем пытался получить доступ к определенной информации.
Летом 2013 года Национальные Институты Здравоохранения США (NIH) запустили новый проект под названием Big Data to Knowledge (BD2T, «От баз данных — к новым знаниям»). У этого проекта будет два направления работы. С одной стороны, он должен способствовать развитию и появлению новых биоинформатических центров, с другой — организовать сеть экспертных групп в различных институтах для того, чтобы решить проблемы стандартизации данных и доступа исследователей к информации при условии сохранения необходимой конфиденциальности. Возможно, что в ближайшее время исследования в «сухой» лаборатории получат еще один толчок к развитию, т.к. правительство США планирует ввести открытый доступ к базам данных как обязательное условие для исследований, финансируемых государством.
Конечно, не стоит забывать, что кроме бесспорного блага для исследователей, открытый доступ к научной информации может принести им и немало проблем. Необходимо будет заботиться о сохранении конфиденциальности и о том, чтобы все данные были представлены в одинаковом формате. Для ученых это означает дополнительные заботы: кто-то должен тратить свое время на подготовку информации и приведение ее в стандартный формат для размещения в публичных источниках. А значит, часть денег и времени исследователей будет тратиться уже не на эксперименты. Особенно важной эта проблема может стать для небольших лабораторий. И захотят ли вообще ученые из традиционных лабораторий отвлекаться на решение подобных вопросов, раз для них это не несет никакой выгоды?
Есть мнение, что большинство российских исследований, участвующих в федеральных целевых программах, давно уже решили для себя этот вопрос, а ведь здесь требование пудовых отчетов с формальными показателями куда более бессмысленное, чем выкладывание данных исследований в онлайн. — Ред.
Несмотря на ряд очевидных проблем новое поколение биологов-информатиков полно энтузиазма. Они видят большой потенциал в вычислительном подходе к биологии и верят, что все препятствия на пути к новым открытиям возможно преодолеть. «Я чувствую себя как ребенок в магазине со сладостями», — улыбается Атул Бьют. — «Перед нами столько возможностей».
Перевод редакционной колонки журнала Science [8].
Литература
- «Омики» — эпоха большой биологии;
- Christoph Lippert, Jennifer Listgarten, Robert I. Davidson, Jeff Baxter, Hoifung Poon, et. al.. (2013). An Exhaustive Epistatic SNP Association Analysis on Expanded Wellcome Trust Data. Sci Rep. 3;
- Как предотвратить самоубийство;
- Herve Rhinn, Ryousuke Fujita, Liang Qiang, Rong Cheng, Joseph H. Lee, Asa Abeliovich. (2013). Integrative genomics identifies APOE ε4 effectors in Alzheimer's disease. Nature. 500, 45-50;
- Альцгеймеровский нейротоксин: ядовиты не только фибриллы;
- Возможно, β-амилоид болезни Альцгеймера — часть врождённого иммунитета;
- José Crossa, Yoseph Beyene, Semagn Kassa, Paulino Pérez, John M. Hickey, et. al.. (2013). Genomic Prediction in Maize Breeding Populations with Genotyping-by-Sequencing. G3. 3, 1903-1926;
- R. F. Service. (2013). Biology's Dry Future. Science. 342, 186-189.






