
Компьютерные игры в молекулярную биофизику биологических мембран
23 ноября 2010
Компьютерные игры в молекулярную биофизику биологических мембран
- 6598
- 4
- 6
Нет, это не ландшафт далёкой планеты, выдуманный иллюстратором дешёвого сборника фантастики. Так учёные изображают мембрану клетки и расположенные в ней белки.
коллаж по [26]
-
Автор
-
Редакторы
Двадцатый век стал веком междисциплинарных исследований, когда грань между «классическими» науками практически стёрлась. В XXI столетии тенденция усиливается — слияние «обычной» (хотя тоже междисциплинарной!) молекулярной биофизики и того, что по-английски называют theoretical computer science, породило необыкновенную область — компьютерный, или in silico, эксперимент. В этой статье рассказывается о методической подоплёке подхода in silico и о его конкретном использовании для имитационного изучения «жизни» биологических мембран и населяющих их белковых молекул.
Вся современная биологическая наука вдохновлена и пронизана термином «молекулярный». Ни одна отрасль, даже из числа максимально «традиционных» — ботаника, зоология, экология — нынче не обходит своим вниманием молекулярные аспекты изучаемых явлений, стараясь представить суть объектов своего пристального внимания в свете процессов, протекающих на уровне отдельных клеток и молекул. Даже генетика, которая во времена своего «лжебуржуазного» прошлого была едва ли не сельскохозяйственной наукой, стала одной из первых «молекулярщиц», объясняя основополагающие явления жизни — наследственность и эволюцию — на языке ДНК, РНК и белков, «дирижирующих» всеми процессами в клетке.
«Родословная» молекулярных наук
Молекулярные науки, объединённые под знамёнами биологии, делятся на биохимический и биофизический лагеря, с разных позиций изучающие примерно одни и те же объекты и явления. Например, биохимию, интересуют больше всего реакции с участием низкомолекулярных веществ, которые можно объединить под общим названием «метаболизм». Её «дочка» — молекулярная биология — изучает процессы передачи информации по «классическому» пути ДНК → РНК → белок (а кое-где и в обратную сторону). Вирусология концентрируется исключительно на вирусах, уже потеряв надежду на существование какой-нибудь единой молекулярно-биологической схемы для этого поистине потрясающего своим разнообразием царства «бесклеточной жизни».

Рисунок 1. Одним из первых биофизических экспериментов может считаться знаменитый опыт Гальвани — во времена, когда не только о компьютерах никто не слыхивал, но и до открытия молекул как таковых оставалось ещё минимум столетие
В центре внимания молекулярной биофизики находятся структура, динамика и функция биомолекул, их способность к самоорганизации и бесчисленным, но, по-видимому, очень точно скоординированным взаимодействиям между собой. Как синтезированная белковая цепь, изначально «одномерная», принимает уникальную, только ей свойственную, пространственную укладку, обеспечивающую выполнение назначенной этому белку функции? Каким образом молекулы липидов, будучи смешаны с водой, образуют пузырьки и другие структуры, очень напоминающие мембраны живых клеток? Как происходит «перекачка» протонов с одной стороны мембраны на другую в дыхательной цепи, при фотосинтезе или в фотоцикле многих бактерий? Каким образом молекулы, которых в клетке считанные штуки, находят друг друга в «чаще», называемой цитоплазмой, чтобы провзаимодействовать и запустить какой-нибудь жизненно важный процесс? Как транскрипционным белкам удаётся не пропустить нужный сайт в ДНК хромосомы, по сравнению с которой километр запутанной колючей проволоки — это просто аккуратный «бантик»?

Рисунок 2. Одним из первых молекулярных биофизиков может считаться Эрвин Шрёдингер, чья книга «Что такое жизнь с точки зрения физики?», написанная ещё в 1944 году, и по сей день не утратила своей актуальности
На все эти вопросы отвечает (ну или, по крайней мере, пытается ответить) молекулярная биофизика. Её цель — описать на строгом языке точных наук, как «живут» биологические молекулы в клетке и каким образом взаимодействуют друг с другом, инициируя сложнейшие последовательности событий, которые уже на более «формальном» уровне — то есть, не опускаясь до физических деталей межмолекулярных взаимодействий — изучают молекулярная биология, генетика и другие биологические отрасли более «высокого уровня».
Поставленные молекулярной биофизикой цели очень важны: несмотря на обвинения в редукционизме, понимание молекулярных «тонкостей» кроет в себе фантастическую возможность — создание новых молекул, которые могли бы желаемым для «создателя» образом играть свою партию во внутриклеточной молекулярной «симфонии», предотвращая болезни или управляя синтезом нужных человеку веществ. А перспективы использования этой способности поистине безграничны — от инновационных отраслей медицины, пищевой и энергетической промышленностей до совершенно нового технологического уклада всего общества, избавленного от неискоренимых проблем экономики перепроизводства сегодняшнего дня.
У истоков молекулярной биофизики
Молекулярная биофизика стала активно развиваться в 1950–1960-х годах, когда идеи и подходы молекулярной физики, коллоидной химии и химии высокомолекулярных соединений стали пытаться применять к изучению строения белковых молекул, важность которых для любых жизненных процессов уже была в полной мере осознана. Уже были созданы биохимические методы секвенирования (то есть, определения аминокислотной последовательности) белковых молекул, уже принесли первые плоды эксперименты по рентгеновской дифракции на белковых кристаллах, в результате которых стало известно пространственное строение первых белков — миоглобина и гемоглобина.
Биофизиков, занимавшихся в то время теоретическими вопросами биологической термодинамики и теории информации и изучавших действие радиации на живую материю, а в более молекулярной области — искусственные однослойные и бислойные мембраны, стало живо интересовать строения белковых молекул и возможность их моделирования с использованием статистических подходов теории полимеров. Однако очень быстро стало ясно, что белки, хотя и представляют собой линейные (то есть неветвящиеся) полимеры, в корне отличаются от гомополимеров полиэтилена или, скажем, каучука, со строением которых уже более или менее разобралась полимерная химия того времени. Белки являются гетерополимерами, то есть цепочками из звеньев-мономеров двадцати основных разновидностей, и именно это определяет богатейший арсенал функций, которыми наделила их природа и перечень ролей, которые возложила на них жизнь.
Белки, несомненно, устроены принципиально более сложно и «умно», чем каучук или другие похожие полимеры, и это исключительно благодаря тому, что образующие их «звенья» — аминокислоты — несут боковые радикалы с сильно различающимися свойствами. Ровно в силу этой же причины структура белков намного сложнее и разнообразнее, нежели структура гомополимеров, и здесь-то и выяснилось, что все существующие полимерные теории не описывают многообразия форм, которые могут принимать белковые молекулы.
Проблему самопроизвольного сворачивания белковой цепи в уникальную пространственную структуру в настоящее время называют фолдингом (от англ. to fold — складывать, сворачивать), и она считается крупнейшей неразрешённой задачей биофизики современности. Структура белковых молекул интересует учёных не напрасно: согласно максиме, называемой также «основной догмой молекулярной биофизики», структура белка, его динамическая подвижность и выполняемые им функции неразрывно связаны между собой. А это обозначает, что ключом к изучению всего белкового «царства» является проблема структуры и динамики полипептидных молекул.
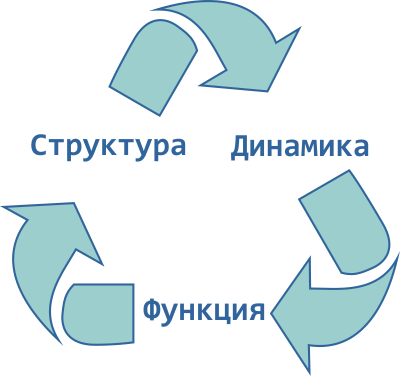
Рисунок 3. Основная догма молекулярной биофизики: структура, функция и динамика биологических макромолекул неразрывно связаны между собой
Однако в чём же здесь проблема и при чём же тут, наконец, компьютеры, упомянутые в названии этой статьи?
Дело в том, что далеко не всегда возможностей экспериментальных методик, которые используются для установления пространственной организации белковых молекул, достаточно для практического решения задачи. В отличие от технологий секвенирования последовательности ДНК геномов живых организмов [1–3], определение пространственной структуры ещё не стало настолько автоматизированным, чтобы структуру любого интересующего белка можно было бы установить с фиксированными временными и материальными затратами. Например, в случае техники рентгеноструктурного анализа наиболее сложным моментом является получение белкового кристалла, образующего при облучении чёткую и качественную дифракционную картину, пригодную для «расшифровки» трёхмерной структуры. Условия, в которых белки образуют упорядоченную фазу, называемую белковым кристаллом, зачастую подобрать очень сложно, и во многих случаях уходят годы упорных экспериментов, прежде чем появляются первые результаты. Другой популярный метод изучения пространственной структуры биологических молекул — спектроскопия ядерного магнитного резонанса (ЯМР) — также имеет ряд ограничений, связанных с размером исследуемых молекул, получением достаточных количеств изотопно-меченных образцов белков и расшифровкой получающихся ЯМР-спектров.
Особенно остро проблемы обоих упомянутых методов встают в случае работы со сложными белковыми комплексами или интегральными белками клеточных мембран, — для последних дело усугубляется гетерогенностью и сложной организацией молекулярного окружения, в котором существуют эти молекулы. В изучении мембранных белков, например, один из самых крупных «камней преткновения» — добиться корректного встраивания очищенной или синтезированной белковой молекулы в липидные агрегаты, «изображающие» мембрану в эксперименте, — например, мицеллы или липосомы [4].
Разумной альтернативой в случае, если экспериментальных данных об организации интересующего белка нет, становится область теоретической молекулярной биофизики, которую в общем виде именуют компьютерным экспериментом, или экспериментом in silico.
In vivo, in vitro, in silico
Вторая половина XX века и начало нынешнего столетия дали науке множество теоретических подходов, позволяющих взглянуть на биологические системы глазами физика, что значит — проводить различные расчёты, заменяющие прямые измерения в эксперименте. Например, из анализа кристаллических структур органических соединений составлены таблицы, описывающие длины и углы ковалентных химических связей, ван-дер-ваальсовы радиусы атомов, параметры водородных связей в белках. По спектроскопическим данным сделаны оценки энергетических характеристик химических связей и конформационной подвижности молекул. Эксперименты по изучению переноса аминокислот из неполярной среды (например, октанола) в полярную (вода) позволили численно описать гидрофобность соответствующих аминокислотных остатков, а значит, и дать возможность предсказывать гидрофобные сегменты в белках (которые, согласно физико-химическим расчётам, соответствуют либо трансмембранным доменам, либо «ядрам» сворачивания глобулярных белков). Сопоставление экспериментально полученных пространственных структур родственных белков показало, что эволюционно родственные (имеющие общее происхождение) молекулы очень часто имеют близкое строение, а значит, факт биологического родства можно использовать для теоретического предсказания строения белков.
Рисунок 4. Первые крупные успехи структурной биологии — такие как установление принципов строения ДНК и белков — были сделаны за рубежом (см., например, «На заре молекулярной графики»), однако России тоже есть чем похвастаться. Один из очагов становления молекулярной биофизики в 1950-е годы возник именно в нашей стране, на физфаке ЛГУ. Тут работали такие замечательные ученые как М. В. Волькенштейн, Э. В. Фрисман, А. Н. Тернин и другие (см. подробнее на сайте музея истории физики в СПбГУ).
монография М.В. Волькенштейна «Конфигурационная статистика полимерных цепей» (Москва-Ленинград, Изд-во АН СССР, 1959)
Постепенно стали обрисовываться контуры новой парадигмы в биологических исследованиях, получившей название компьютерного, или in silico-эксперимента [5]. Этимологически этот термин развивает известные в биологии латинизмы — in vivo («в живом») и in vitro («в стекле», применяется для лабораторных экспериментов на упрощённых системах, имитирующих живые). “In silico” буквально обозначает «в кремнии», что метафорически отсылает к микропроцессорам, способным выполнять огромные объёмы вычислений и имитировать биологические процессы на модельной системе, существующей лишь в памяти электронно-вычислительной машины (ЭВМ).
Про прогресс ЭВМ и сетей связи подробно можно не рассказывать — каждый человек старше 25 лет помнит времена, когда просто немыслимо было представить, что вскоре почти у каждого будет свой собственный мобильный телефон и портативный компьютер с доступом в интернет.
С самого «рождения» новая концепция in silico сопровождается предостережением: вряд ли когда-нибудь точность описания процессов, протекающих в живой клетке, достигнет таких высот, чтобы можно было моделировать жизнь с безукоризненной точностью — само понятие жизни вряд ли допускает такой «бездушный» подход к тайнам природы. Но, вооружившись современными знаниями и здоровым скепсисом (чтобы не выдавать на каждом шагу желаемое за действительное), с помощью in silico-исследований зачастую можно получить об исследуемой частичке живого массу информации, которую вряд ли добудешь иным способом.
Речь тут идёт, в первую очередь, о высокой контролируемости и воспроизводимости in silico-имитации: мы можем с требуемой точностью задать начальные параметры системы и измерить получающееся на выходе, а также повторить моделирование любое число раз, меняя только те параметры, которые нас в данный момент интересуют. А ведь именно этого часто бывает практически невозможно добиться в «настоящем» in vitro- или, тем более, in vivo-эксперименте. Таким образом, новая парадигма достаточно органично сочетается с «традиционными» экспериментальными подходами, и их взаимное обогащение преимуществами друг друга даёт исследователям больше свободы в бесконечной погоне за тайнами жизни.
Если говорить о математическом аппарате для моделирования поведения и взаимодействия молекул, то первым делом вспоминают, конечно, квантовую механику — теорию, которая наиболее корректно на сегодняшний день описывает строение и свойства молекул — квантовых объектов. Однако загвоздка тут в том, что для системы, существенно превышающей по размеру, например, атом гелия (а любые белки существенно превышают этот размер), решение уравнения Шрёдингера практически невозможно из-за астрономической вычислительной сложности, экспоненциально возрастающей с увеличением числа элементарных частиц, образующих систему. Практически невозможно и приближённое решение этого уравнения, или даже приближённое решение его приближённого аналога (а именно этим занимаются так называемые полуэмпирические методы квантовой механики).
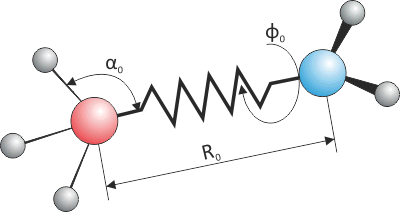
Рисунок 5. Представление молекулы с точки зрения молекулярной механики. Параметры молекулы описываются не уравнением Шрёдингера, а суммой «классических» взаимодействий, самое сложное из которых — формула для упругости, описывающая колебание пружинки. (На рисунке схематично показаны лишь три таких слагаемых: валентная связь, валентный угол и торсионный угол.)
Основной подход, в настоящее время используемый для описания структуры и динамических свойств биологических молекул, — это молекулярная механика, или метод эмпирических силовых полей. Главная идея, заключающаяся в этой концепции, — упрощение физических законов: молекулу рассматривают не как квантово-механическую систему, а как набор классических частиц («шариков»), связанных между собой парными взаимодействиями («пружинками») — ковалентными, электростатическими, ван-дер-ваальсовыми и другими связями (рис. 5). Параметры, характеризующие все взаимодействия в системе и тем самым её энергетическую функцию, подбирают эмпирически (из условия самосогласованности теоретических и экспериментальных характеристик системы), а набор всех таких параметров образует эмпирическое силовое поле (ЭСП).
Численное решение уравнения движения частиц в составе молекулы (а это всем известный Второй закон Ньютона) позволяет изучить динамику молекулы во времени, — отсюда и название одного из основных алгоритмов, основанных на ЭСП — метод молекулярной динамики (МД) [6]. Кроме этого конкретного метода, существует масса других приложений ЭСП, объединённых общей идеей — вычисляя потенциальную энергию системы, установить её динамическое поведение во времени либо близкую к существующей в природе пространственную структуру.
Другой задачей молекулярного моделирования является теоретическое предсказание пространственной структуры биологических молекул (в основном, белков), — поскольку, как уже говорилось ранее, далеко не во всяком случае можно рассчитывать на экспериментальное установление этой структуры. Молекулярная динамика и другие алгоритмы, основанные на ЭСП, пока сравнительно немного могут предложить для решения этой проблемы. Первая, наиболее очевидная причина тому — длительность МД-имитации, достижимая на современном компьютерном оборудовании, в большинстве случаев слишком мала для рассмотрения тех временных интервалов, в течение которых на самом деле в природе происходит самопроизвольное сворачивание биомолекул (хотя этот разрыв стремительно сокращается [7]). Однако есть и другое, более принципиальное ограничение: не исключено, что основополагающее приближение всей идеи эмпирических силовых полей — в первую очередь, отказ от рассмотрения квантовой природы молекул, — закрывает дорогу к реалистичной имитации такого сложнейшего физического процесса, как фолдинг (самопроизвольное сворачивание биомолекулы).
В связи с этим в большинстве практических приложений теоретическое моделирование строения белковых молекул основано на уже известных структурах эволюционно родственных белков, поскольку эмпирически показано, что очень часто пространственная структура намного более консервативна, чем аминокислотная последовательность. Метод, с помощью которого возможно построить трёхмерную модель структуры белка, основываясь на пространственной структуре белка-«родственника» (называемого в данном случае шаблоном), называется моделированием на основании гомологии, или сопоставительным моделированием [8], [9]. Именно этот алгоритм даёт «стартовое приближение» для разнообразных задач теоретической молекулярной биофизики, имеющих дело с молекулами, структура которых не охарактеризована экспериментально.
Ещё один часто употребляемый алгоритм молекулярного моделирования — молекулярный докинг, предназначенный для изучения механизмов взаимодействия двух молекул между собой (от англ. to dock — стыковаться, пришвартовываться). Этот метод, имея на входе структуры двух молекул, ставит своей целью предсказать наиболее вероятную структуру комплекса, который они могут образовать между собой, а также оценить величину свободной энергии связывания, характеризующей взаимодействие между этими молекулами.
Подводя промежуточный итог, можно резюмировать, что методы молекулярного моделирования, хотя зачастую и основаны на чрезвычайно упрощённых предпосылках, уже сегодня приносят заметную пользу в биологических исследованиях. Далее речь пойдёт о конкретной области применения этих алгоритмов для изучения одного из важнейших компонент жизни — биологических мембран и «населяющих» их белков.
Биологические мембраны — капсула жизни
Биологии известны и бесклеточные формы жизни — вирусы. Хотя многие из них и облачены в оболочку, истинной жизни в ней все равно не происходит. Вся активность начинается и заканчивается исключительно под покровом мембраны клеток заражённого организма, потому что любой вирус — лишь молекулярный паразит, «взламывающий» программу, по которой работает живая клетка, и заставляющий её использовать свои мощности для «штамповки» новых копий вирусов.
Однако все остальные, по-настоящему живые организмы — и эукариоты, и прокариоты, — заключены в мембрану (а иногда ещё и в дополнительную клеточную стенку), чётко разделяющую весь мир на внешний и внутренний. В последнем и творится всё таинство жизни. Именно по этой причине огромное множество жизненных процессов так или иначе связано с работой мембраны клетки, а интегральные белки мембраны играют необычайное многообразие ролей, без которых вообразить существование жизни просто невозможно. Транспорт веществ, передача любых сигналов от клетки к клетке, создание и поддержание трансмембранных потенциалов, без которых не живёт ни одна клетка, и даже рецепция внешних импульсов, таких как свет или запахи, — всё это функции мембранных белков.
Однако изучать строение и работу этих белков очень сложно, потому что их немыслимо рассматривать в отрыве от мембран, представляющих естественную среду их «обитания», а это неизбежно влечёт за собой многократное увеличение сложности рассматриваемой системы. Кроме того, сворачивание мембранных белков протекает в гетерогенном окружении, включающем как гидрофобный слой липидов мембраны, так и полярную среду вне её, — а это задаёт очень непростые «граничные» условия для процесса фолдинга. Молекулярное моделирование мембранных белков сталкивается и с такими плохо формализуемыми (а значит, и поддающимися учёту в виде формул) явлениями, как влияние растворителя в этой сложной «многослойной» системе и гидрофобный эффект, играющий в жизни белков мембраны ещё более важную роль, чем в случае растворимых белков цитоплазмы.
Однако понимание всей важности ролей, которые играют мембранные белки (рис. 6), а также огромной практической значимости изучения их строения и функций только разжигает интерес исследователей к проблеме биологических мембран и их белковой «начинки», — и это касается не только экспериментаторов, но и биофизиков-теоретиков, которых занимают молекулярные аспекты течения жизни.
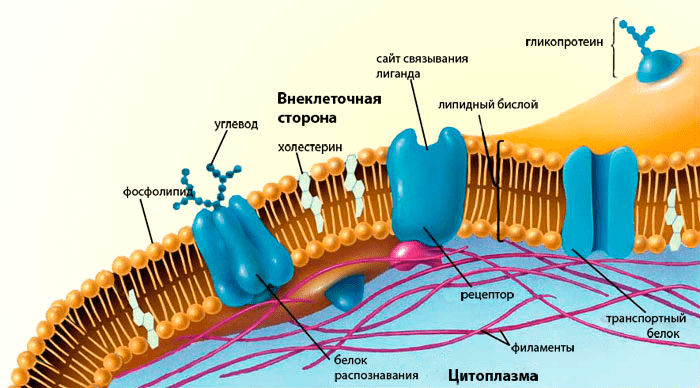
Рисунок 6. Биологическая мембрана и её «начинка». Липидный бислой мембраны, а также её интегральные и периферические белки, играют в жизни клетки роль, которую сложно переоценить. Далеко неполный перечень функций белков мембраны — трансмембранная передача сигналов и веществ, межклеточное распознавание, создание электрохимических потенциалов, реакция на свет и другие импульсы и др.
Лаборатория моделирования биомолекулярных систем в Институте биоорганической химии им. академиков М. М. Шемякина и Ю. А. Овчинникова Российской академии наук специализируется именно на этом вопросе. Основной её профиль — молекулярное моделирование биологических мембран и процессов, проходящих с участием мембранных белков. В следующем разделе в общих чертах будет обрисован фронт исследований, проводимых в Лаборатории.
Не только игры: как с помощью компьютеров моделируют белки биологических мембран
Для компьютерных имитаций мембранных белков в первую очередь необходимы, конечно, реалистичные молекулярные модели мембран, в которых эти белки «живут». Мембраны состоят из липидов — молекул амфифильной природы, включающих гидрофильную головку и гидрофобные хвосты. Простейший аналог мембраны — это липидный бислой, состоящий из двух «листочков», «склеенных» гидрофобными хвостами молекул. Противоположные части молекул — полярные головки — формируют внешние поверхности бислоя, граничащие с водой. Конечно, реальные биологические мембраны состоят не из одного типа молекул, а из множества компонентов — фосфолипидов различного заряда, ганглиозидов, сфингомиелина, кардиолипина, стероидных липидов и прочих соединений (и это не считая белков, которыми «нашпигованы» мембраны). Причем липидный состав мембран существенным образом меняется для разных клеток и разных организмов в связи со спецификой возложенных на эти клетки функций.
Молекулярно-динамическая модель мембраны, даже если это бислой, состоящий из одинаковых молекул, — это уже достаточно большая система. В её состав входит несколько сотен молекул липида, упорядоченных в форме бислоя, и десятков тысяч молекул воды, изображающих окружающую мембрану водную среду. Для каждого атома в этой системе заданы координаты, химические связи и нековалентные взаимодействия с другими атомами в системе. Компьютерный расчёт динамики такой элементарной ячейки проводят на многопроцессорных системах, и длится он обычно несколько недель, описывая при этом всего лишь несколько десятков или сотен наносекунд (10−9 с) «реальной» жизни мембраны.
Несмотря на столь малые времена, которые удаётся проследить с помощью такого моделирования, система начинает вести себя подобно реальному бислою, — в частности, толщина мембраны и плотность упаковки липидных «головок» становятся очень близки к значениям, известным из эксперимента, подтверждая реалистичность проводимых расчётов. Однако кроме получения и так уже известных параметров моделирование позволяет проникнуть в детали устройства границы вода / мембрана, чего вряд ли можно добиться другими средствами — очень уж тонко устроена и подвижна эта граница.

Рисунок 7. Молекулярные модели клеточных мембран. Варьирование липидного состава позволяет имитировать мембраны эукариот и прокариот: «эритроциты» (слева, изображают эукариотическую мембрану) и «грам−» (справа, изображают мембрану бактерий). Бислои представлены в виде поверхности, доступной растворителю (вид «сверху»). Цветом показаны липиды: ПОФЭ, ПОФХ, ПОФГ и ХОЛ.
В Лаборатории накоплен банк динамических моделей бислоёв различного состава — с разной длиной и структурой гидрофобных «хвостов» и природой полярных «головок» (заряженных или нейтральных) [10]. Созданы и модели двухкомпонентных мембран, в том числе несущих на своей поверхности отрицательный заряд. Наличие заряда в модельных мембранах важно потому, что это позволяет имитировать мембраны бактерий (и не только их), заряженные отрицательно, а значит, и изучать влияние различных веществ на «бактериальные» мембраны в сравнении с мембранами «эукариот». Почему это важно? Потому что такие вещества могут обладать антимикробной активностью, а, значит, — использоваться в медицине!
Важнейшие из таких веществ, найденные в живых организмах, — это небольшие молекулы белковой природы, которые назвали мембрано-активными пептидами. Делятся они на три группы — антимикробные пептиды (избирательно убивающие бактерий), фузионные пептиды (вызывающие слияние двух клеток между собой) и транслокационные пептиды, самопроизвольно проникающие сквозь мембрану и способные «протащить» за собой полезный груз. Практическое значение этих молекул — альтернатива традиционным антибиотикам, утратившим былую универсальность из-за появления резистентности к ним микроорганизмов [11], направленная доставка веществ (например, лекарств или же, наоборот, ядов) в заданные типы клеток и дизайн противовирусных препаратов, блокирующих слияние мембраны клетки с мембраной «захватчика» (например, гриппа).
Кроме того, из-за небольшого размера (обычно это не более 20–30 аминокислотных остатков) такие молекулы можно исследовать на компьютере, имитируя их взаимодействие с различными типами модельных мембран [12]. В результате таких расчётов стало понятно, что взаимодействие пептид-мембрана — очень тонкий процесс, существенно зависящий от свойств последней и не оставляющий без изменений ни пептид, ни мембрану. И тот, и другая точно подстраиваются друг к другу, формируя селективные области взаимодействия с особыми свойствами, отличными от «незатронутой» мембраны [13], [27]. Подробный анализ на атомном уровне механизма таких взаимодействий позволяет «вмешиваться» в процесс и увеличивать или уменьшать «сродство» молекулы к мембранам определённого типа — в частности, увеличивать антимикробный потенциал и уменьшать токсичность по отношению к клеткам эукариот. В результате такого «дизайна» были созданы аналоги антимикробного пептида латарцина, убивающие бактерий (например, стафилококков или патогенные штаммы кишечной палочки), но не затрагивающие эритроциты, а, значит, являющиеся потенциальным прототипом лекарственного препарата [14] (рис. 8).

Рисунок 8. Моделирование взаимодействия антимикробного пептида латарцина с мембраной «эритроцитов». Анализ механизма взаимодействия позволяет выявить молекулярные детерминанты, управляющие сродством пептида к тому или иному типу мембраны, и направленно регулировать это сродство. Такая возможность открывает путь к дизайну аналога пептида, обладающему прежней антимикробной активностью, но уменьшенной токсичностью по отношению к клеткам животных и человека.
Более сложная задача — моделирование структуры интегральных белков мембраны, например, мембранных рецепторов, роль которых в работе всех без исключения клеток сложно переоценить. Экспериментально строение таких рецепторов установлено лишь для небольшого числа представителей этого важнейшего семейства [15–17], — в связи с чем молекулярное моделирование приобретает особенное значение. Первым приближением в моделировании их структуры являются модели, построенные на основе гомологии с родственными рецепторами, структура которых уже установлена, однако точность таких моделей почти всегда оказывается недостаточной для практических применений [9]. Вызвано это тем, что, несмотря на сходный план строения, разнообразие выполняемых функций определяет существенные отличия в устройстве активных центров и близлежащих областей.
Для оптимизации моделей структуры можно использовать статистические закономерности упаковки мембранных белков, наблюдаемые в уже известных структурах. Выраженные численно, эти закономерности помогают уточнить модель, получая более «правильно» упакованную структуру [18], [19], [28]. Кроме того, использование алгоритмов для учёта гидрофобного эффекта, важного не только для сворачивания белковых молекул в мембране, но и для процессов межмолекулярного распознавания рецептор–лиганд, даёт возможность ещё точнее «настроить» расположение функционально важных остатков в активном центре рецепторов [20], [21].
Эти технологии использовали для моделирования пространственной организации ряда рецепторов из крупного семейства, действующего через активацию G-белка — внутриклеточного мессенджера, запускающего биохимические каскады внутри клетки. Среди таких белков — рецепторы нейрогормонов мелатонина [22], [23] и вазоактивного интестинального пептида [24] (рис. 9), играющих важную роль в иммунитете и адаптационных процессах (в том числе — циркадных ритмах).
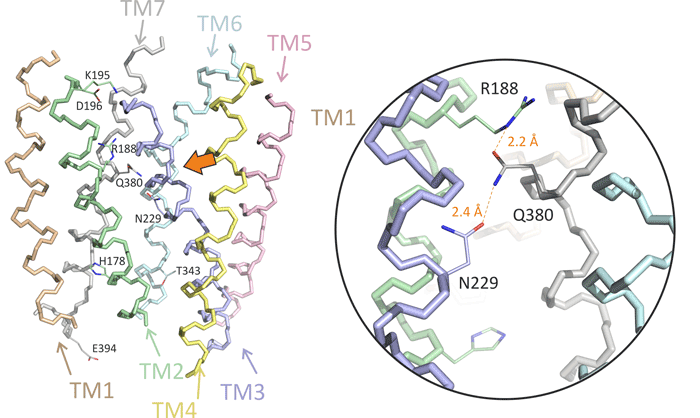
Рисунок 9. Молекулярная структура трансмембранного домена рецептора вазоактивного интестинального пептида (ВИП). Каждая из семи ТМ-α-спиралей окрашена в свой цвет; функционально важные аминокислотные остатки подписаны. Справа показан крупный план, отмеченный слева оранжевой стрелкой.
Модели упомянутых рецепторов, обозначаемых также аббревиатурой GPCR (от англ. G-protein coupled receptors), несут в себе большой практический потенциал — ведь с их помощью в научных лабораториях и исследовательских подразделениях фармацевтических гигантов занимаются дизайном новых лекарственных форм, которые через десятилетие придут на смену сегодняшним лекарствам. Новое поколение лекарств упрочнит сильные стороны препаратов сегодняшнего дня и сведёт к минимуму их многочисленные недостатки (такие как токсичность, малая эффективность и избирательность действия, чрезмерная зависимость от фенотипа больного и другие). Кроме того, лекарства будущего будут нацелены на «новые» биохимические пути, пока никак не задействованные в фармакологии, чтобы дать врачам средства более тонкой и направленной регуляции жизненных процессов для сохранения пациентам жизни и возвращения здоровья.
Компьютерные эксперименты будущего
Несмотря на огромный объём проделанной работы, сегодня in silico-эксперимент делает лишь первые шаги, уже став, однако, существенным подспорьем для «обычного» эксперимента, позволяя формулировать новые гипотезы и выявлять возможные механизмы молекулярных процессов, протекающих внутри живой клетки. Что ждёт эту область в будущем, предсказать под силу, наверное, только фантастам, однако точно сказать можно одно — имитация жизненных процессов на уровне молекул, выполняемая на электронных (а в будущем — кто знает — квантовых?) вычислительных машинах прочно заняла своё место в науках о жизни, гармонично дополняя лабораторные эксперименты. Аналогичным образом около ста лет назад ставшие сегодня рутиной in vitro-опыты «подвинули» исследования на подопытных животных, — так что новый виток в развитии науки начинается уже сегодня.
Эта статья написана при поддержке РФФИ (конкурс на написание научно-популярных статей), № проекта: 10-04-11516-с. В сокращённом виде она опубликована в журнале «Природа» [25].
Литература
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
- Геном человека: как это было и как это будет;
- Перевалило за тысячу: третья фаза геномики человека;
- Шибаев А.В. и Карабельская О.А. (2010). Мицеллы — самоорганизующиеся структуры. «Природа». 2, 9–17;
- In vivo — in vitro — in silico;
- Молекулярная динамика биомолекул. Часть I. История полувековой давности;
- Миллисекундный барьер взят!;
- Чугунов А.О. и Ефремов Р.Г. (2009). Предсказание пространственной структуры белков: акцент на мембранных мишенях. «Биоорганическая химия». 35, 744–760;
- Торжество компьютерных методов: предсказание строения белков;
- Anton A. Polyansky, Pavel E. Volynsky, Dmitry E. Nolde, Alexander S. Arseniev, Roman G. Efremov. (2005). Role of Lipid Charge in Organization of Water/Lipid Bilayer Interface: Insights via Computer Simulations. J. Phys. Chem. B. 109, 15052-15059;
- Антимикробные пептиды — возможная альтернатива традиционным антибиотикам;
- Anton Polyansky, Anton Chugunov, Alexander Vassilevski, Eugene Grishin, Roman Efremov. (2012). Recent Advances in Computational Modeling of α.-Helical Membrane- Active Peptides. CPPS. 13, 644-657;
- Anton A. Polyansky, Pavel E. Volynsky, Alexander S. Arseniev, Roman G. Efremov. (2009). Adaptation of a Membrane-active Peptide to Heterogeneous Environment. I. Structural Plasticity of the Peptide. J. Phys. Chem. B. 113, 1107-1119;
- Anton A. Polyansky, Alexander A. Vassilevski, Pavel E. Volynsky, Olga V. Vorontsova, Olga V. Samsonova, et. al.. (2009). N-terminal amphipathic helix as a trigger of hemolytic activity in antimicrobial peptides: A case study in latarcins. FEBS Letters. 583, 2425-2428;
- Зрительный родопсин — рецептор, реагирующий на свет;
- Новый рубеж: получена пространственная структура β2-адренорецептора;
- Рецепторы в активной форме;
- Чугунов А.О., Новоселецкий В.Н., Арсеньев А.С., Ефремов Р.Г. (2007). Новый метод оценки качества упаковки трансмембранных α-спиральных доменов в белках. «Биохимия». 72, 358–367;
- Anton O. Chugunov, Valery N. Novoseletsky, Dmitry E. Nolde, Alexander S. Arseniev, Roman G. Efremov. (2007). Method To Assess Packing Quality of Transmembrane α-Helices in Proteins. 1. Parametrization Using Structural Data. J. Chem. Inf. Model.. 47, 1150-1162;
- Roman Efremov, Anton Chugunov, Timothy Pyrkov, John Priestle, Alexander Arseniev, Edgar Jacoby. (2007). Molecular Lipophilicity in Protein Modeling and Drug Design. CMC. 14, 393-415;
- Timothy V. Pyrkov, Anton O. Chugunov, Nikolay A. Krylov, Dmitry E. Nolde, Roman G. Efremov. (2009). PLATINUM: a web tool for analysis of hydrophobic/hydrophilic organization of biomolecular complexes. Bioinformatics. 25, 1201-1202;
- Anton O. Chugunov, Amaury Farce, Philippe Chavatte, Roman G. Efremov. (2006). Differences in Binding Sites of Two Melatonin Receptors Help to Explain Their Selectivity to Some Melatonin Analogs: A Molecular Modeling Study. Journal of Biomolecular Structure and Dynamics. 24, 91-107;
- Amaury Farce, Anton O. Chugunov, Cédric Logé, Ahmed Sabaouni, Saïd Yous, et. al.. (2008). Homology modeling of MT1 and MT2 receptors. European Journal of Medicinal Chemistry. 43, 1926-1944;
- A. O. Chugunov, J. Simms, D. R. Poyner, Y. Dehouck, M. Rooman, et. al.. (2010). Evidence that Interaction between Conserved Residues in Transmembrane Helices 2, 3, and 7 Are Crucial for Human VPAC1 Receptor Activation. Molecular Pharmacology. 78, 394-401;
- Чугунов А.О. (2010). Компьютерные игры в молекулярную биофизику. «Природа». 12, 36–43;
- Donald M. Engelman. (2005). Membranes are more mosaic than fluid. Nature. 438, 578-580;
- Anton A. Polyansky, Pavel E. Volynsky, Alexander S. Arseniev, Roman G. Efremov. (2009). Adaptation of a Membrane-active Peptide to Heterogeneous Environment. II. The Role of Mosaic Nature of the Membrane Surface. J. Phys. Chem. B. 113, 1120-1126;
- Anton O. Chugunov, Valery N. Novoseletsky, Dmitry E. Nolde, Alexander S. Arseniev, Roman G. Efremov. (2007). Method To Assess Packing Quality of Transmembrane α-Helices in Proteins. 2. Validation by “Correct vs Misleading” Test. J. Chem. Inf. Model.. 47, 1163-1170.






