Чтение молекулярных отпечатков
04 мая 2009
Чтение молекулярных отпечатков
- 557
- 0
- 1
Так выглядел один из первых препаратов полирибосом (полисом) под электронным микроскопом. Полирибосомы окрашены методом негативного контрастирования.
-
Автор
-
Редакторы
Сочетание рибосомного профилирования и параллельного высокопроизводительного секвенирования нового поколения породило метод, с помощью которого учёные могут наблюдать трансляцию тысяч мРНК одновременно.
Поразительные возможности технологий секвенирования нового поколения оказывают огромное влияние на самые различные сферы современной молекулярной биологии [1], [2]. Например, при полной расшифровке новых геномов или повторном прочтении (ресеквенировании) уже расшифрованных геномов быстрота и относительная дешевизна новых технологий позволяют проводить эксперименты такого масштаба, какого раньше и представить нельзя было. В случае, когда объектом исследования является РНК (при этом секвенируют ДНК-копии транскриптов — так называемые кДНК), параллельное секвенирование делает возможным проведение более точного количественного анализа экспрессии генов с более широким динамическим диапазоном, чем у традиционных методов. Наконец, при профилировании мРНК, вовлечённых в трансляцию (процесс белкового синтеза), применение новых технологий секвенирования позволяет не только эффективно и очень точно изучать данный этап экспрессии генов, но и дает массу качественно новой информации. Недавно опубликованная в журнале Science статья, вышедшая из лаборатории Джонатана Вайссмана (Jonathan Weissman) из Калифорнийского университета в Сан-Франциско, сообщает первые результаты такого подхода.
Рибосома, «сидящая» на мРНК и ведущая её трансляцию, закрывает собой сегмент длиной в 30 нуклеотидов, делая их недоступными для воздействия рибонуклеазы, уничтожающей остальную (незащищённую) РНК. Как правило, одну молекулу РНК транслирует полисома, или полирибосома — комплекс из нескольких рибосом (рис. 1). Количество рибосом в полисоме зависит от скорости инициации, элонгации и терминации на конкретной РНК. В настоящее время принята модель, в которой у эукариот начало мРНК (5′ нетранслируемый участок) и её конец (3′ нетранслируемый участок) расположены близко друг другу за счёт взаимодействия одного из факторов инициации трансляции (еIF4F) с поли(А)-связывающим белком (ПАБ), ассоциированным с 3′ нетранслируемым участком. Параллельное секвенирование кДНК-библиотек, полученных из всех защищённых фрагментов РНК, оставшихся в клетке, позволило Вайссману и его коллегам получить исчерпывающую картину белкового синтеза в клетке [3], [4].
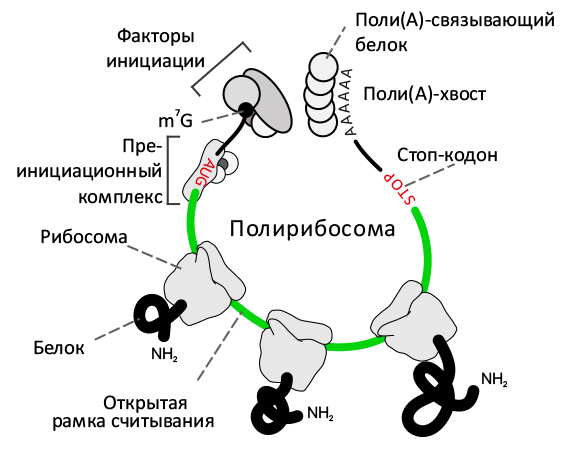
Рисунок 1. Анатомия полирибосомы
Такой подход (рис. 2) может быть использован для решения множества разнообразных задач. Во-первых, весьма вероятно, что данный подход будет использован для подробного исследования протеома [5], что позволит составить полный «каталог» всех полипептидов, синтезируемых клеткой. По словам самого Вайссмана: «Для сложных геномов (таких как человеческий) аннотировать все экспрессируемые белки практически невозможно. Однако наш подход, по существу, позволяет вам сделать это». В статье, опубликованной Вайссманом и его соавторами, сообщается о результатах, полученных на пекарских дрожжах, однако, в принципе, подобные исследования можно проводить на любом организме. Более того, применение рибосом, содержащих искусственно введённые эпитопы (эпитоп — участок молекулы, специфически узнаваемый антителом), позволит изучать трансляцию в отдельных группах клеток (тканях). «Я думаю, что для некоторых областей [исследований], таких как молекулярная нейроанатомия, это послужит началом новой эры», — с примерной скромностью говорит Вайссман.
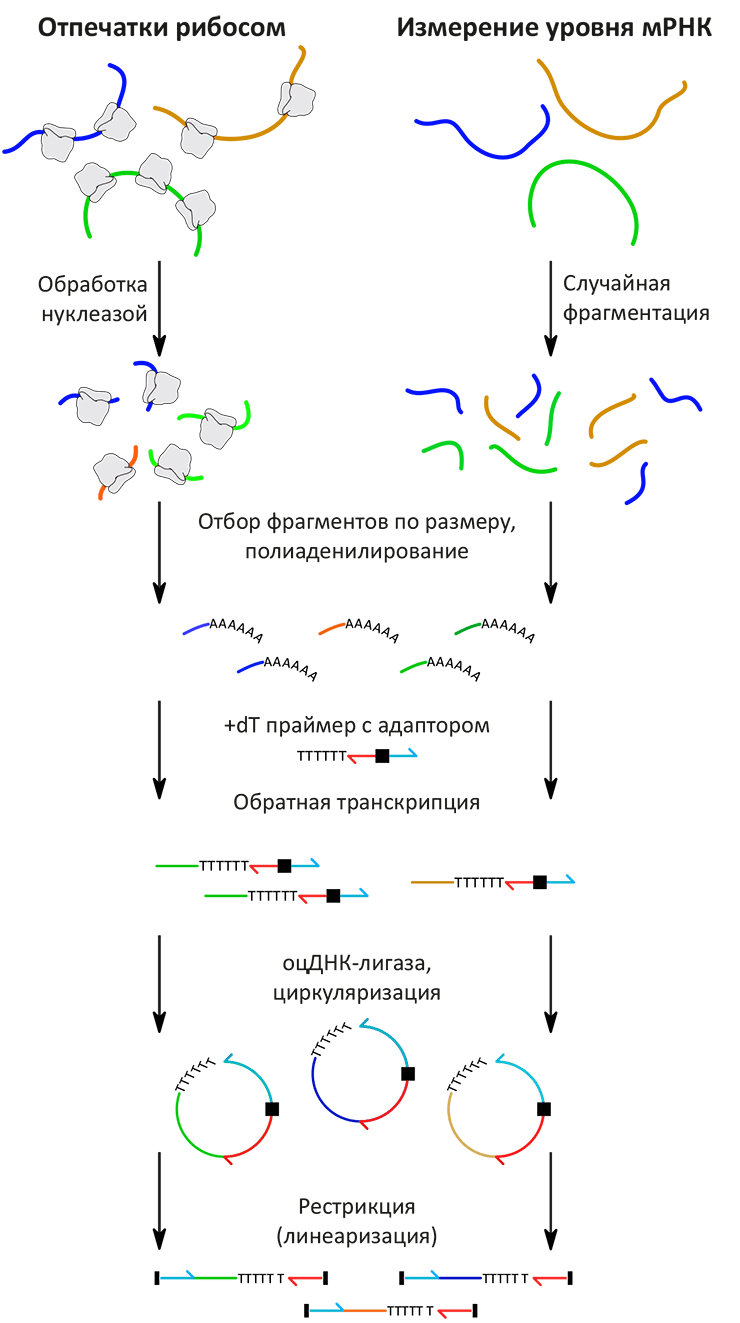
Рисунок 2. Схема эксперимента по рибосомному профилированию (чтение отпечатков рибосом на мРНК) или секвенированию молекул мРНК, фрагментированных случайным образом
Во-вторых, построение полирибосомных профилей является более точным способом изучения производства белков в клетке, чем просто измерение количества той или иной мРНК. Учёные из лаборатории Вайссмана использовали профилирование рибосом в сочетании с параллельным секвенированием для картирования плотности «отпечатков» рибосом на тысячах различных мРНК. Эти измерения затем были использованы для вычисления скоростей трансляции белковых молекул. Исследователи утверждают, что рассчитанная таким образом скорость трансляции позволяет более точно предсказать количество синтезированного белка, чем измерение количества соответствующей мРНК. «Чем привлекательна количественная протеомика, — сообщает Вайссман, — так это тем, что позволяет нам оценить, насколько успешно мы продвигаемся». Действительно, внеся поправку на повышенную плотность рибосом на 5′-конце мРНК, учёные обнаружили существенную взаимосвязь между уровнем трансляции и количеством синтезированного белка (коэффициент корреляции ≈0,6).
Третья область, в которой рибосомные профили, несомненно, найдут применение, — это изучение контроля трансляции. В статье калифорнийских учёных этот метод использовался для изучения влияния аминокислотного голодания на трансляцию у дрожжей. Нет никаких сомнений, что данный метод также будет использован для изучения регуляции белкового синтеза при различных болезнях и стрессовых состояниях в клетках высших организмов.
Наконец, следует отметить, что описанный метод обладает высоким разрешением (до одного нуклеотида), позволяющим однозначно определять открытую рамку считывания мРНК, участвующую в трансляции. Следовательно, метод может быть использован для изучения запрограммированных сдвигов рамки считывания и «проскоков» стоп-кодонов. Или же, как было в работе лаборатории Вайссмана, метод оказывается полезным для картирования неканонических сайтов инициации трансляции в 5′-нетранслируемых областях молекул мРНК.
Согласно заключению самого Вайссмана, «сегодня стало возможным напрямую проводить высокоточные измерения уровня трансляции белков. Этот подход будет применяться для выяснения того, какие именно белки и в каком количестве производятся в клетке. Кроме того, данный метод сам по себе является прекрасным аналитическим инструментом для изучения процесса трансляции как такового».
Литература
- 454-секвенирование (высокопроизводительное пиросеквенирование ДНК);
Скверный анекдот: негр, китаец и Крейг Вентер...;- N. T. Ingolia, S. Ghaemmaghami, J. R. S. Newman, J. S. Weissman. (2009). Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science. 324, 218-223;
- Natalie de Souza. (2009). Deep sequencing of ribosome footprints. Nat Methods. 6, 244-244;
- Миллиард на протеомику;
- Warner J.R., Knopf P.M., Rich A. (1963). A multiple ribosomal structure in protein synthesis. Proc. Natl. Acad. Sci. USA. 49, 122–129.